R语言学习 - 非参数法生存分析--转载
生存分析指根据试验或调查得到的数据对生物或人的生存时间进行分析和推断,研究生存时间和结局与众多影响因素间关系及其程度大小的方法,也称生存率分析或存活率分析。常用于肿瘤等疾病的标志物筛选、疗效及预后的考核。
简单地说,比较两组或多组人群随着时间的延续,存活个体的比例变化趋势。活着的个体越少的组危险性越大,对应的基因对疾病影响越大,对应的药物治疗效果越差。
生存分析适合于处理时间-事件数据,如下
生存时间数据有两种类型:
- 完全数据 (complete data)指被观测对象从观察起点到出现终点事件所经历的时间; 一般用状态值1或TRUE表示。
- 截尾数据 (consored data)或删失数据,指在出现终点事件前,被观测对象的观测过程终止了。由于被观测对象所提供的信息是不完全的,只知道他们的生存事件超过了截尾时间。截尾主要由于失访、退出和终止产生。一般用状态值0或FALSE表示。
- TCGA中的临床数据标记也符合这个规律,在下面软件运行时也可修改状态值的含义, 但一般遵循这个规律。
生存概率 (survival probability)指某段时间开始时存活的个体至该时间结束时仍然存活的可能性大小。
生存概率=某人群活过某段时间例数/该人群同时间段期初观察例数。
生存率 (Survival rate),用S(t)表示,指经历t个单位时间后仍存活的概率,若无删失数据,则为活过了t时刻仍然存活的例数/观察开始的总例数。如果有删失数据,分母则需要按时段进行校正。
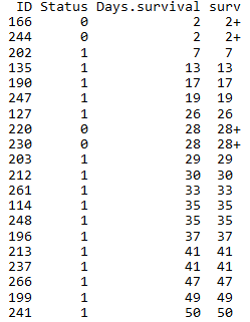
生存分析一个常用的方法是寿命表法。
寿命表是描述一段时间内生存状况、终点事件和生存概率的表格,需计算累积生存概率即每一步生存概率的乘积 (也可能是原始生存概率),可完成对病例随访资料在任意指定时点的生存状况评价。
R做生存分析
R中做生存分析需要用到包survival和survminer。输入数据至少两列,存活时间和生存状态,也就是测试数据中的Days.survial和vital_status列。如果需要比较不同组之间的差异,也需要提供个体的分组信息,如测试数据中的PAM50列。对应TCGA的数据,一般根据某个基因的表达量或突变有无对个体进行分组。
读入数据
library(survival)
BRCA <- read.table('BRCA.tsv', sep="\t", header=T)
head(BRCA)- 1
- 2
- 3
ID SampleType PAM50Call_RNAseq Days.survival pathologic_stage
1 TCGA-E9-A2JT-01 Tumor_type LumA 288 stage iia
2 TCGA-BH-A0W4-01 Tumor_type LumA 759 stage iia
3 TCGA-BH-A0B5-01 Tumor_type LumA 2136 stage iiia
4 TCGA-AC-A3TM-01 Tumor_type Unknown 762 stage iiia
5 TCGA-E9-A5FL-01 Tumor_type Unknown 24 stage iib
6 TCGA-AC-A3TN-01 Tumor_type Unknown 456 stage iib
vital_status
1 0
2 0
3 0
4 0
5 0
6 0- 1
- 2
- 3
- 4
- 5
- 6
- 7
- 8
- 9
- 10
- 11
- 12
- 13
- 14
简单地看下每一列都有什么内容,方便对数据整体有个了解,比如有无特殊值。
summary(BRCA)- 1
ID SampleType PAM50Call_RNAseq Days.survival
TCGA-3C-AAAU-01: 1 Tumor_type:1090 Basal :138 Min. : 0.0
TCGA-3C-AALI-01: 1 Her2 : 65 1st Qu.: 450.2
TCGA-3C-AALJ-01: 1 LumA :415 Median : 848.0
TCGA-3C-AALK-01: 1 LumB :194 Mean :1247.0
TCGA-4H-AAAK-01: 1 Normal : 24 3rd Qu.:1682.8
TCGA-5L-AAT0-01: 1 Unknown:254 Max. :8605.0
(Other) :1084
pathologic_stage vital_status
stage iia :359 Min. :0.0000
stage iib :259 1st Qu.:0.0000
stage iiia:156 Median :0.0000
stage i : 90 Mean :0.1394
stage ia : 85 3rd Qu.:0.0000
stage iiic: 67 Max. :1.0000
(Other) : 74 - 1
- 2
- 3
- 4
- 5
- 6
- 7
- 8
- 9
- 10
- 11
- 12
- 13
- 14
- 15
- 16
计算寿命表
# Days.survival:跟踪到的存活时间
# vital_status: 跟踪到的存活状态
# ~1表示不进行分组
fit <- survfit(Surv(Days.survival, vital_status)~1, data=BRCA)
# 获得的survial列就是生存率
summary(fit)
Call: survfit(formula = Surv(Days.survival, vital_status) ~ 1, data = BRCA)
time n.risk n.event survival std.err lower 95% CI upper 95% CI
116 1021 1 0.999 0.000979 0.997 1.000
158 1017 1 0.998 0.001386 0.995 1.000
160 1016 1 0.997 0.001697 0.994 1.000
172 1010 1 0.996 0.001962 0.992 1.000
174 1008 1 0.995 0.002195 0.991 0.999
197 1003 1 0.994 0.002406 0.989 0.999
224 993 1 0.993 0.002604 0.988 0.998
227 990 1 0.992 0.002788 0.987 0.998
239 987 1 0.991 0.002961 0.985 0.997
255 981 1 0.990 0.003125 0.984 0.996
266 978 1 0.989 0.003282 0.983 0.996
295 965 1 0.988 0.003435 0.981 0.995
302 962 1 0.987 0.003581 0.980 0.994
304 958 1 0.986 0.003723 0.979 0.993
320 948 1 0.985 0.003862 0.977 0.993
322 946 1 0.984 0.003995 0.976 0.992- 1
- 2
- 3
- 4
- 5
- 6
- 7
- 8
- 9
- 10
- 11
- 12
- 13
- 14
- 15
- 16
- 17
- 18
- 19
- 20
- 21
- 22
- 23
- 24
- 25
- 26
- 27
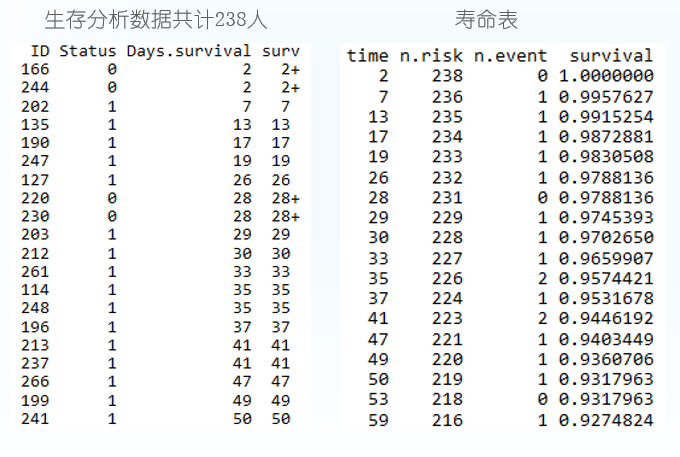
绘制生存曲线,横轴表示生存时间,纵轴表示生存概率,为一条梯形下降的曲线。下降幅度越大,表示生存率越低或生存时间越短。
library(survminer)
# conf.int:是否显示置信区间
# risk.table: 对应时间存活个体总结表格
ggsurvplot(fit, conf.int=T,risk.table=T)- 1
- 2
- 3
- 4
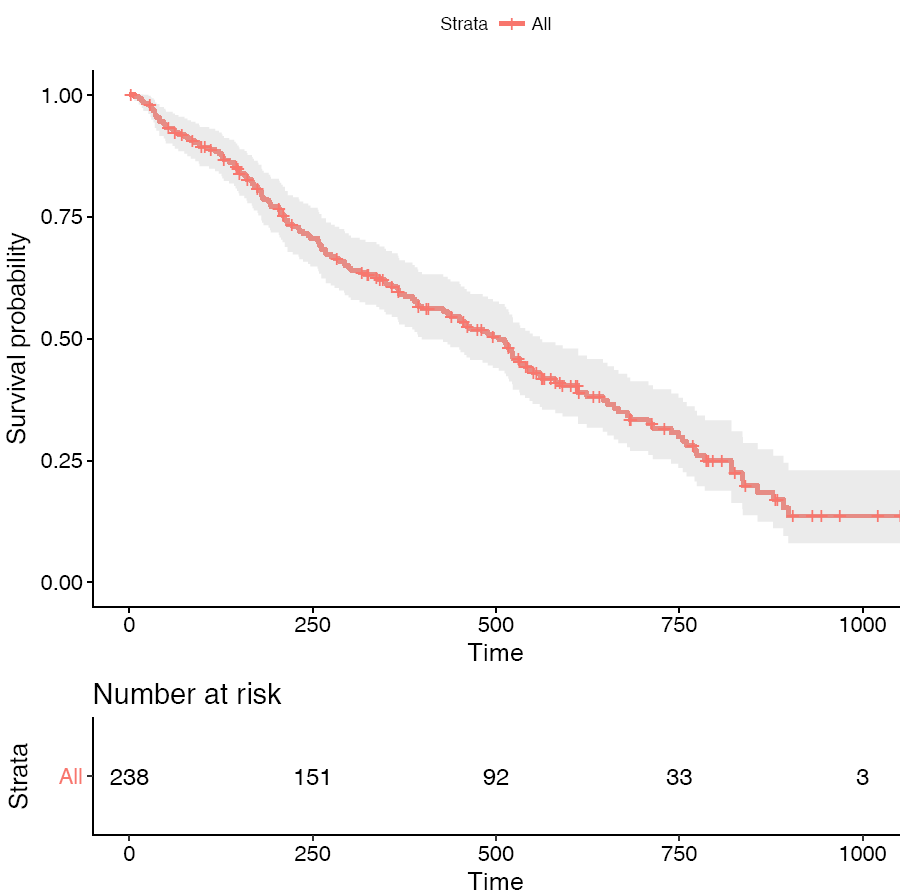
PAM50是通过50个基因的表达量把乳腺癌分为四种类型 (Luminal A, Luminal B, HER2-enriched, and Basal-like)作为预后的标志。根据PAM50属性对病人进行分组,评估比较两组之间生存率的差别。
# 这三步不是必须的,只是为了方便,选择其中的4个确定了的分组进行分析
# 同时为了简化图例,给列重命名一下,使得列名不那么长
BRCA_PAM50 <- BRCA[grepl("Basal|Her2|LumA|LumB",BRCA$PAM50Call_RNAseq),]
BRCA_PAM50 <- droplevels(BRCA_PAM50)
colnames(BRCA_PAM50)[colnames(BRCA_PAM50)=="PAM50Call_RNAseq"] <- 'PAM50'
# 按PAM50分组
fit <- survfit(Surv(Days.survival, vital_status)~PAM50, data=BRCA_PAM50)
# 绘制曲线
ggsurvplot(fit, conf.int=F,risk.table=T, risk.table.col="strata", pval=T)- 1
- 2
- 3
- 4
- 5
- 6
- 7
- 8
- 9
简化Stage信息,先只查看大的阶段
BRCA_PAM50$pathologic_stage <- gsub('(i+v*).*', "\\1", BRCA_PAM50$pathologic_stage)
BRCA_PAM50$pathologic_stage <- as.factor(BRCA_PAM50$pathologic_stage)
colnames(BRCA_PAM50)[colnames(BRCA_PAM50)=="pathologic_stage"] <- 'PS'
fit <- survfit(Surv(Days.survival, vital_status)~PS, data=BRCA_PAM50)
# 绘制曲线
ggsurvplot(fit, conf.int=F,risk.table=T, risk.table.col="strata", pval=T)- 1
- 2
- 3
- 4
- 5
- 6
参考资料
- http://rpubs.com/xuefliang/153247
- http://www.sthda.com/english/wiki/survminer-r-package-survival-data-analysis-and-visualization
R语言学习 - 非参数法生存分析--转载的更多相关文章
- R语言学习笔记:分析学生的考试成绩
孩子上初中时拿到过全年级一次考试所有科目的考试成绩表,正好可以用于R语言的统计分析学习.为了不泄漏孩子的姓名,就用学号代替了,感兴趣可以下载测试数据进行练习. num class chn math e ...
- R语言︱词典型情感分析文本操作技巧汇总(打标签、词典与数据匹配等)
每每以为攀得众山小,可.每每又切实来到起点,大牛们,缓缓脚步来俺笔记葩分享一下吧,please~ --------------------------- 笔者寄语:情感分析中对文本处理的数据的小技巧要 ...
- R语言学习 第四篇:函数和流程控制
变量用于临时存储数据,而函数用于操作数据,实现代码的重复使用.在R中,函数只是另一种数据类型的变量,可以被分配,操作,甚至把函数作为参数传递给其他函数.分支控制和循环控制,和通用编程语言的风格很相似, ...
- R语言学习-(金融数据获取和简单的分析)
利用R语言中的quantmod包和fBasics对股票数据的获取和简要的分析, 通过获取的数据进行典型图像绘制,使用JB正态性检验来检验是否服从于正态分布. 前提概要:quantmod 包默认是访问 ...
- R语言学习路线和常用数据挖掘包(转)
对于初学R语言的人,最常见的方式是:遇到不会的地方,就跑到论坛上吼一嗓子,然后欣然or悲伤的离去,一直到遇到下一个问题再回来.当然,这不是最好的学习方式,最好的方式是——看书.目前,市面上介绍R语言的 ...
- R语言学习路线图-转帖
本文分为6个部分,分别介绍初级入门,高级入门,绘图与可视化,计量经济学,时间序列分析,金融等. 1.初级入门 <An Introduction to R>,这是官方的入门小册子.其有中文版 ...
- R语言学习 第一篇:变量和向量
R是向量化的语言,最突出的特点是对向量的运算不需要显式编写循环语句,它会自动地应用于向量的每一个元素.对象是R中存储数据的数据结构,存储在内存中,通过名称或符号访问.对象的名称由大小写字母.数字0-9 ...
- R语言学习笔记:基础知识
1.数据分析金字塔 2.[文件]-[改变工作目录] 3.[程序包]-[设定CRAN镜像] [程序包]-[安装程序包] 4.向量 c() 例:x=c(2,5,8,3,5,9) 例:x=c(1:100) ...
- R语言学习(一)前言
本系列文章由 @YhL_Leo 出品,转载请注明出处. 文章链接: http://blog.csdn.net/yhl_leo/article/details/49768161 R是一个有着统计分析功能 ...
随机推荐
- golang学习笔记12 beego table name `xxx` repeat register, must be unique 错误问题
golang学习笔记12 beego table name `xxx` repeat register, must be unique 错误问题 今天测试了重新建一个项目生成新的表,然后复制到旧的项目 ...
- Spring Boot 实现RESTful webservice服务端示例
1.Spring Boot configurations application.yml spring: profiles: active: dev mvc: favicon: enabled: fa ...
- JSP Servlet javaben
CLASSPATH=D:\Dev\jdk\lib\tools.jar;D:\Dev\jdk\lib\dt.jarJAVA_HOME=D:\Dev\jdkPath=%JAVA_HOME%\bin 一:T ...
- Netty1
基于Netty4的HttpServer和HttpClient的简单实现 Netty的主页:http://netty.io/index.html 使用的Netty的版本:netty-4.0.23.Fin ...
- fjwc2019 D1T3 不同的缩写(dinic+trie+dfs)
#180. 「2019冬令营提高组」不同的缩写 乍看之下没有什么好的方法鸭.......于是考虑暴力. 长度?二分似乎可行. 于是我们二分最长子串的长度(设为$len$),蓝后暴力查找. 先在每个串内 ...
- oracle orion hugepages_settings.sh(支持OEL 7,4.1内核)
orion需要首先配置hugepage,否则会出现下列错误. [root@yyxxdb01 ~]# /opt/app/11.2.0/grid_home/bin/orion -run oltp -tes ...
- JSch : channel never closed or EOF 通道未关闭
最近,我们的项目在开发远程节点管理的时候,使用了jsch库.在测试的时候发现有个节点在cmd执行完成之后,channel.isClosed()一直都是false,导致请求无法返回,但是其它有些节点就没 ...
- Lyft高管的技术团队管理实战
Lyft 的技术总监沈思维分享了他对于管理技术团队和打造工程文化的经验,也欢迎添加他的微信公众号"人家的屋顶"了解更多(微信公众号ID: othersroof).沈思维毕业于密歇根 ...
- WEB后台认证机制
mark to :http://www.cnblogs.com/xiekeli/p/5607107.html HTTP Basic Auth HTTP Basic Auth简单点说明就是每次请求API ...
- shell &&,||,()
做个笔记. 1. linux命令返回值介绍 shell 在执行某个命令时,会有一个返回值,该值保存在shell变量$?中.当$?为0时,表示命令执行成功:当$?为1时,表示命令执行失败. 2. &am ...
