R数据分析:孟德尔随机化实操
好多同学询问孟德尔随机化的问题,我再来尝试着梳理一遍,希望对大家有所帮助,首先看下图1分钟,盯着看将下图印在脑海中:
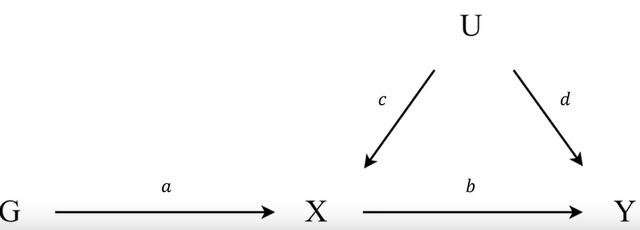
上图是工具变量(不知道工具变量请翻之前的文章)的模式图,明确一个点:我们做孟德尔的时候感兴趣的是x和y的关系,也就是小b,但是我们直接去跑x对y的回归肯定是不对的,因为有很多的U,因此我们借助工具变量G(关于工具变量我们之前的文章有详细的解释,请自行查阅),去估计我们感兴趣的小b。
现在有天然良好的工具变量G,也就是我们的基因变量,此时有上面的图,再次重申:我们感兴趣的,最终希望得到准确估计的值是小b,按照上图我们应该有GY的关系是ab,GX的关系是a,于是乎b可以写成ab/a,就是我们感兴趣的b可以换一种思路得到,如下:

上面的式子要跑通的话,我们需要知道G-Y的关系和G-X的关系。
但是我们GY也就是基因和结局的关系已经有人给我们研究好了,我们可以直接去GWAS里面找研究好的summarydata拿来用就行。
但是我们的的GX也就是基因和暴露的关系也已经有人给我们研究好了,我们可以直接去GWAS里面找研究好的summarydata拿来用就行。
也就是说,通过孟德尔随机化,我们完全可以毫不费力地估计出我们需要的小b,也就是暴露和结局的关系----就是今天要再次给大家介绍的孟德尔随机化研究。
思路就是这么清晰。就是这么清晰。搞不明白的同学再多读几遍。
术语解析
为了帮助大家理解思想,在孟德尔随机化的实操中有几个术语得提点一波:
连锁不平衡(linkage disequilibrium):刚刚讲我们可以有很多的基因结局/暴露的关系的,就是GWAS里面好些基因可以用,这个时候我们不希望基因之间有相关(会造成double counting,使得结果偏倚):
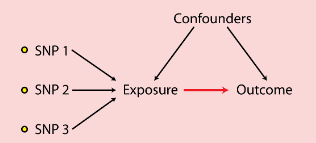
我们实际做的时候,模式是像上图,snp之间你说不相干就不相干?当两个位点的不同等位基因的关联频率高于或低于独立随机关联的条件下的期望频率,这种情况是客观存在的,此时时这些工具变量之间相关性就叫连锁不平衡,其大小可以用LD r方来表示,这个指标也是我们在操作时需要设定的指标之一。
水平基因多效性(Horizontal Pleiotropy):理解这个概念先看下图:
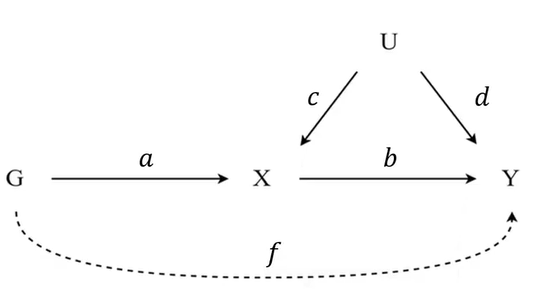
意思是我的理想的情况是通过ab/a的操作估计出b,但是看上图,是不是免不了会出现f这条路径,如果出现了f,我们的基因和结局之间的关系就是f+ab,此时,我用原来的方法估计的就不是b了,而是b+f/a了,就不对了(始终记住我们关心的是b)。
但是如果我的基因变量很多,从而有很多的f,如果所有f的期望均值为0,那么最后我们汇总一下得到的结果也基本上就是b了,无伤大雅。但是就怕所有的f都是一边偏向的(都大于0或都小于0),此时就有问题了,叫做定向多效性directional pleiotropy,这也是为什么我们最后要做漏斗图的原因。
就是通过漏斗图一看都是所有的工具变量都是呈漏斗分布的,就说明没有偏向,这个时候我们认为定向多效性都被冲掉了,不影响。
好,解释了上面的一些术语之后,我们实操一波。
实操
最基本的例子:BMI on CHD的例子,我想看一下BMI作为暴露,CHD作为结局的mr,代码就4条:
bmi_exp_dat <- extract_instruments(outcomes = 'ieu-a-2')chd_out_dat <- extract_outcome_data(snps = bmi_exp_dat$SNP, outcomes = 'ieu-a-7')dat <- harmonise_data(bmi_exp_dat, chd_out_dat)res <- mr(dat)
结果如下,下图中有不同方法出来的我们关心的小b:
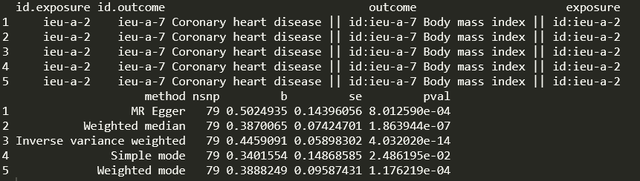
这个就算做完了,就这么简单快速。
接下来就是敏感性分析,首先是各个工具变量的异质性检验:
mr_heterogeneity(dat)
运行代码后可以得到Cochran’s Q统计量
然后是水平基因多效性检验,代码如下:
mr_pleiotropy_test(dat)
运行代码可以得到egger_intercept
然后是单个SNP结果检验,代码如下:
res_single <- mr_singlesnp(dat)
运行后可以得到每个SNP的小b
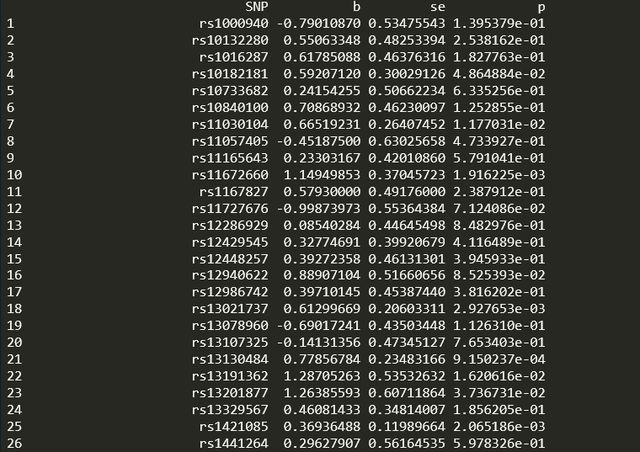
然后是留一检验,代码如下:
mr_leaveoneout(dat)
接下来,论文中还会有几个图,首先是点图,代码如下:
mr_scatter_plot(res, dat)
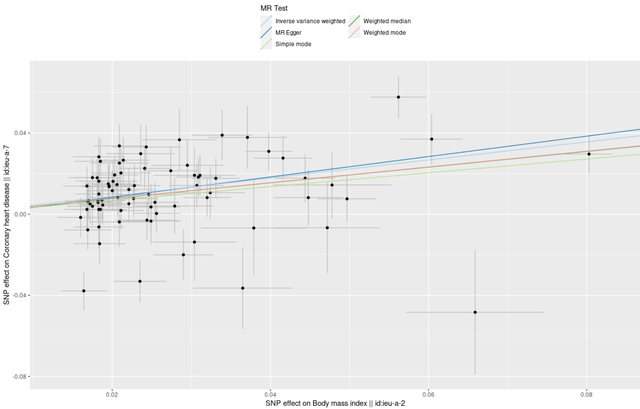
点图是将同一个SNP对暴露的效果放在横轴,对结局的效果放在纵轴,此时图中的斜率就是我们的估计的小b。
然后是单个SNP效应组合的森林图用mr_forest_plot函数可以得到,mr_leaveoneout_plot可以得到留一分析的森林图,mr_funnel_plot可以帮我们得到漏斗图。
到这就出了所有需要报告的东西,做完了。
但是上面的流程有很多的前提,比如你得知道暴露和结局的GWASid才能进行下去,GWAS又有很多,比如你直接用上面的代码的话其实是MR Base GWAS catalog里面的GWAS,当然你还可以选别的,或者用自己找来的最新的GWAS都是可以的。

第一步首先是在相应的GWAS中找到暴露的summary data:
那么有那些GWAS可以供我们使用呢?我们可以直接把GWAS的目录调出来瞅瞅,代码如下:
data(gwas_catalog)
运行后大约可以得到15万个全基因组关联研究的数据,截图如下:
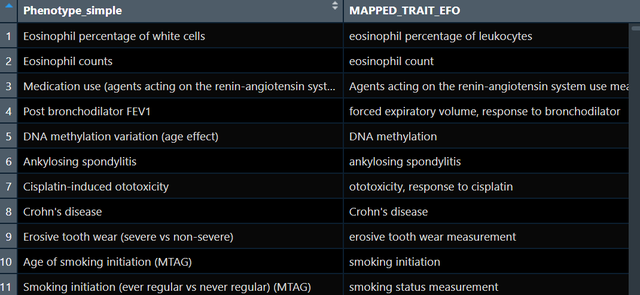
那么对我们而言,我们现在需要找到我们关心的暴露对应的GWAS,比如我现在要找与“blood”表型相关的GWAS,我可以写出如下代码:
exposure_gwas <- subset(gwas_catalog, grepl("Blood", Phenotype_simple))
上面的代码相当于只用Phenotype_simple这一列做筛选,当然你也可以结合其它的列比如人群,比如作者,比如地区等等,都是可以的。
选好暴露相关的GWAS之后要做的就是进一步确定基因工具变量和暴露的强度,在论文中一般是这么描述:First, relevance assumption was met considering that all SNPs have reached genome-wide significance (p < 5 × 10−8)
具体的操作如下:
exposure_gwas<-exposure_gwas[exposure_gwas$pval<5*10^-8,]
通过上面的步骤保证我们的基因工具变量一定是和暴露强相关。
然后就是将准备好的暴露的GWAS数据形成可以用来做MR分析的数据格式,需要用到format_data()函数:
exposure_data<-format_data(exposure_gwas)
此时的exposure_data大概长这样:
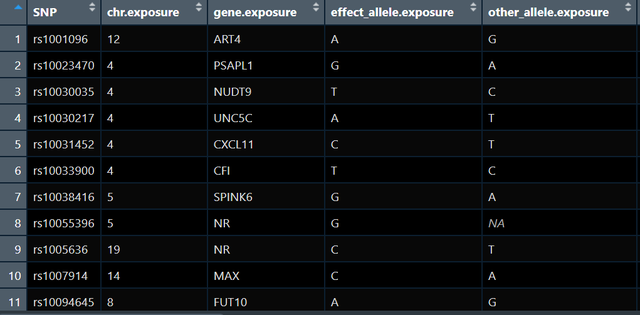
可以看到有很多个基因工具变量SNP,这个时候我们需要考虑连锁不平衡(linkage disequilibrium):
exposure_data<-clump_data(exposure_data, clump_r2 = 0.001)
上面的代码中clump_r2则是设定的容许相关性,到这儿我们算是手动地将工具变量都筛出来了,解决了找工具变量的问题,还有一个方法是自动筛选工具变量,比如我暴露是bmi,我可以写出如下代码:
subset(ao, grepl("body mass", trait))
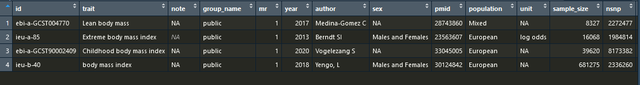
运行后我知道我可以选的gwasid是ieu-b-40,这个时候我也可以自动提取出工具变量,这两种方法达到的目的都是一样的:
extract_instruments('ieu-b-40')
然后依照工具变量进行结局的summary estimates的提取,提取结局的summary data也需要是需要知道GWASid的对吧,比如我现在关心的结局是收缩压,我就可以写出如下代码:
outcome_gwas <- subset(ao, grepl("Systolic", trait))
运行后我就可以知道所有的和收缩压相关的gwasid了,我选一个最新的,比如我就选下面的2021年的:
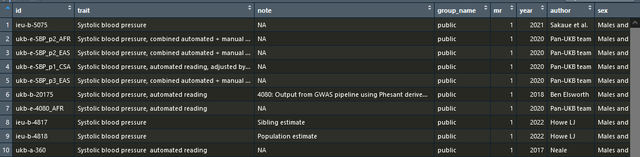
看图我们知道它对于的id是ieu-b-5075,我就这么写:
outcome_data <- extract_outcome_data(snps = exposure_data$SNP, outcomes = "ieu-b-5075")
后续通过合并直接做mr分析就可以,流程就没有不同了。
小结
今天给大家写了孟德尔随机话的实操,文章图示例来自【中文孟德尔随机化】英国布里斯托大学MRC-IEU《R语言做孟德尔随机化》第一章:用MRBase网页工具和R包TwoSampleMR做两样本孟德尔随机化_哔哩哔哩_bilibili,感谢大家耐心看完
R数据分析:孟德尔随机化实操的更多相关文章
- R数据分析:样本量计算的底层逻辑与实操,pwr包
样本量问题真的是好多人的老大难,是很多同学科研入门第一个拦路虎,今天给本科同学改大创标书又遇到这个问题,我想想不止是本科生对这个问题不会,很多同学从上研究生到最后脱离科研估计也没能把这个问题弄得很明白 ...
- Python相关分析—一个金融场景的案例实操
哲学告诉我们:世界是一个普遍联系的有机整体,现象之间客观上存在着某种有机联系,一种现象的发展变化,必然受与之关联的其他现象发展变化的制约与影响,在统计学中,这种依存关系可以分为相关关系和回归函数关系两 ...
- R数据分析:跟随top期刊手把手教你做一个临床预测模型
临床预测模型也是大家比较感兴趣的,今天就带着大家看一篇临床预测模型的文章,并且用一个例子给大家过一遍做法. 这篇文章来自护理领域顶级期刊的文章,文章名在下面 Ballesta-Castillejos ...
- R数据分析:潜类别轨迹模型LCTM的做法,实例解析
最近看了好多潜类别轨迹latent class trajectory models的文章,发现这个方法和我之前常用的横断面数据的潜类别和潜剖面分析完全不是一个东西,做纵向轨迹的正宗流派还是这个方法,当 ...
- R数据分析:临床预测模型中校准曲线和DCA曲线的意义与做法
之前给大家写过一个临床预测模型:R数据分析:跟随top期刊手把手教你做一个临床预测模型,里面其实都是比较基础的模型判别能力discrimination的一些指标,那么今天就再进一步,给大家分享一些和临 ...
- 号外号外:9月13号《Speed-BI云平台案例实操--十分钟做报表》开讲了
引言:如何快速分析纷繁复杂的数据?如何快速做出老板满意的报表?如何快速将Speed-BI云平台运用到实际场景中? 本课程将通过各行各业案例背景,将Speed-BI云平台运用到实际场景中 ...
- QVM 实操记 - 18.12.28
视频回放地址:https://i.iamlj.com/mp4/QVM-IMC-12.27-1080P.mp4 目录 目录 常规开发部署流程 准备工作 开发准备 网站部署 操作步骤 重装系统 LANP环 ...
- Python关于类的实操
实操一:总结 1.什么是绑定到对象的方法,如何定义,如何调用,给谁用?有什么特性? 2.什么是绑定到类的方法,如何定义,如何调用,给谁用?有什么特性? 3.什么是解除绑定的函数,如何定义,如何调用,给 ...
- Linux基础实操五
实操一:nginx服务 二进制安装nginx包1) 1)#yum clean all 2)#yum install epel-release -y 3)#yum install nginx -y 1) ...
随机推荐
- 【java】基础1-字符串、堆、栈、静态与引用类型
/*结论:1,一般变量(int,float,boolean..)使用==比较,引用类型(String,int[],对象)使用equals比较.2,一般的变量存放在栈中,new出来的对象都存放在堆中,字 ...
- 如何结合整洁架构和MVP模式提升前端开发体验 - 整体架构篇
本文不详细介绍什么是整洁架构以及 MVP 模式,自行查看文章结尾相关链接文章. 整洁架构粗略介绍 下图为整洁架构最原始的结构图: Entities/Models:实体层,官方说法就是封装了企业里最通用 ...
- C# winfrom ListView控件实现自由设置每一行字体及背景色等
背景:公司经常会需要将日志信息,输出到一个对话框中显示出来.之前一直采用的listbox控件,操作简单,使用方便,但是遗憾的是,不能自由控制每一行的状态. 于是想了如下几个方案: (1)重绘listb ...
- 大促活动如何抵御大流量 DDoS 攻击?
每一次活动大促带来的迅猛流量,对技术人而言都是一次严峻考验.如果在活动期间遭受黑产恶意DDoS攻击,无疑是雪上加霜.电商的特性是业务常态下通常不会遭受大流量DDoS攻击,且对延迟敏感,因此只需要在活动 ...
- KingbaseES 中实现mysql的from_days和to_days
mysql中两个函数的说明: TO_DAYS(date)给出一个日期date,返回一个天数. FROM_DAYS(N)给出一个天数N,返回一个DATE值. 两个函数比较计算的日期都是 0000-01- ...
- 如何查找并简单分析core文件
当系统发生coredump时,通常需要通过分析core文件来定位问题所在,但实际工作中,有时却发现core 文件找不到,或者core文件被删除了. 一.core文件没有生成 KINGBASE core ...
- 最新一线大厂Redis使用21条军规及详细解读
说明:个人原创,本人在一线互联网大厂维护着几千套集群,关于redis使用的一些坑进行了经验总结,希望能给大家带来一些帮助 适用场景:并发量大.访问量大的业务 规范:介绍军规内容 解读:讲解军规设置原因 ...
- 安装ceph (快速) 步骤三: Ceph 客户端
大多数 Ceph 用户不会直接往 Ceph 存储集群里存储对象,他们通常会使用 Ceph 块设备. Ceph 文件系统.或 Ceph 对象存储这三大功能中的一个或多个. 前提条件 先完成存储集群快速入 ...
- Beats:在Docker里运行Filebeat
- 计算shell 脚本的执行时间
# shell_time.sh #!/bin/bash UseTime () { startTime_s=`date +%s` # 获取从1970-01-01 00:00:00 UTC到现在的秒数 $ ...
