吴裕雄--天生自然 R语言开发学习:聚类分析















#-------------------------------------------------------#
# R in Action (2nd ed): Chapter 16 #
# Cluster analysis #
# requires packaged NbClust, flexclust, rattle #
# install.packages(c("NbClust", "flexclust", "rattle")) #
#-------------------------------------------------------# par(ask=TRUE)
opar <- par(no.readonly=FALSE) # Calculating Distances
data(nutrient, package="flexclust")
head(nutrient, 2)
d <- dist(nutrient)
as.matrix(d)[1:4,1:4] # Listing 16.1 - Average linkage clustering of nutrient data
data(nutrient, package="flexclust")
row.names(nutrient) <- tolower(row.names(nutrient))
nutrient.scaled <- scale(nutrient)
d <- dist(nutrient.scaled)
fit.average <- hclust(d, method="average")
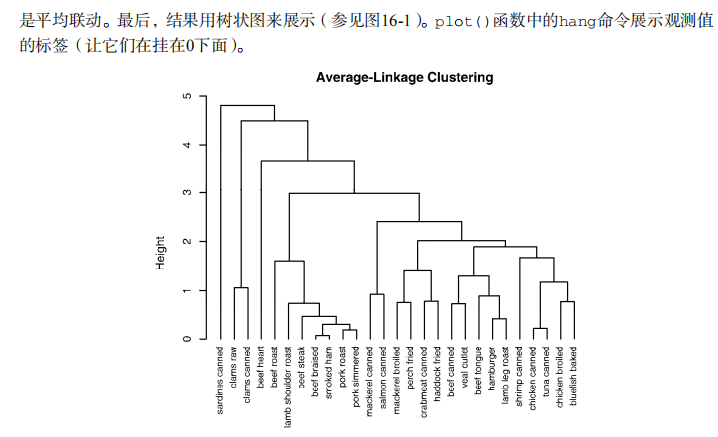
plot(fit.average, hang=-1, cex=.8, main="Average Linkage Clustering") # Listing 16.2 - Selecting the number of clusters
library(NbClust)
nc <- NbClust(nutrient.scaled, distance="euclidean",
min.nc=2, max.nc=15, method="average")
par(opar)
table(nc$Best.n[1,])
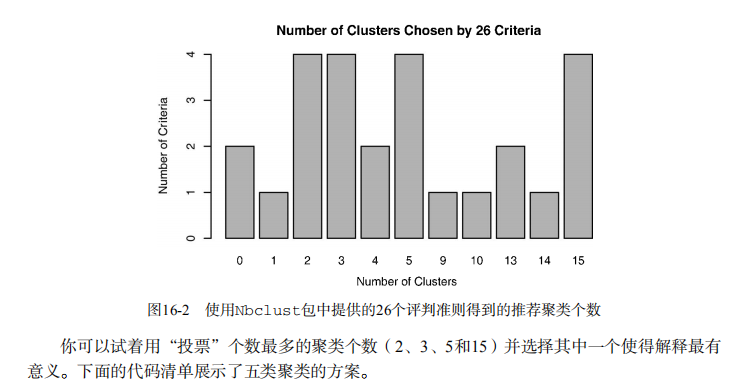
barplot(table(nc$Best.n[1,]),
xlab="Numer of Clusters", ylab="Number of Criteria",
main="Number of Clusters Chosen by 26 Criteria") # Listing 16.3 - Obtaining the final cluster solution
clusters <- cutree(fit.average, k=5)
table(clusters)
aggregate(nutrient, by=list(cluster=clusters), median)
aggregate(as.data.frame(nutrient.scaled), by=list(cluster=clusters),
median)
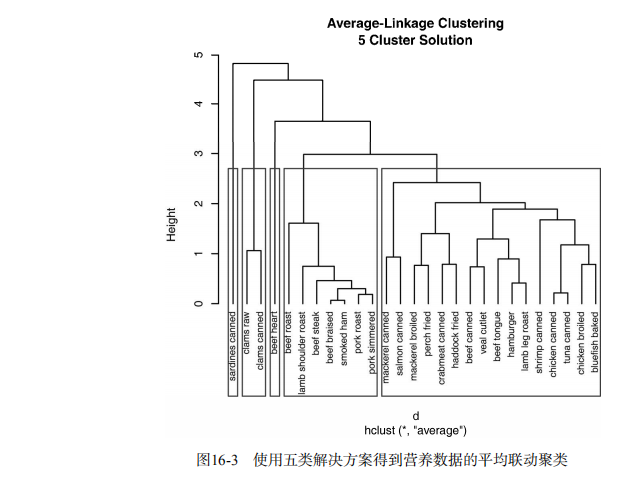
plot(fit.average, hang=-1, cex=.8,
main="Average Linkage Clustering\n5 Cluster Solution")
rect.hclust(fit.average, k=5) # Plot function for within groups sum of squares by number of clusters
wssplot <- function(data, nc=15, seed=1234){
wss <- (nrow(data)-1)*sum(apply(data,2,var))
for (i in 2:nc){
set.seed(seed)
wss[i] <- sum(kmeans(data, centers=i)$withinss)}
plot(1:nc, wss, type="b", xlab="Number of Clusters",
ylab="Within groups sum of squares")} # Listing 16.4 - K-means clustering of wine data
data(wine, package="rattle")
head(wine)
df <- scale(wine[-1])
wssplot(df)
library(NbClust)
set.seed(1234)
nc <- NbClust(df, min.nc=2, max.nc=15, method="kmeans")
par(opar)
table(nc$Best.n[1,])
barplot(table(nc$Best.n[1,]),
xlab="Numer of Clusters", ylab="Number of Criteria",
main="Number of Clusters Chosen by 26 Criteria")
set.seed(1234)
fit.km <- kmeans(df, 3, nstart=25)
fit.km$size
fit.km$centers
aggregate(wine[-1], by=list(cluster=fit.km$cluster), mean) # evaluate clustering
ct.km <- table(wine$Type, fit.km$cluster)
ct.km
library(flexclust)
randIndex(ct.km) # Listing 16.5 - Partitioning around mediods for the wine data
library(cluster)
set.seed(1234)
fit.pam <- pam(wine[-1], k=3, stand=TRUE)
fit.pam$medoids
clusplot(fit.pam, main="Bivariate Cluster Plot") # evaluate clustering
ct.pam <- table(wine$Type, fit.pam$clustering)
ct.pam
randIndex(ct.pam) ## Avoiding non-existent clusters
library(fMultivar)
set.seed(1234)
df <- rnorm2d(1000, rho=.5)
df <- as.data.frame(df)
plot(df, main="Bivariate Normal Distribution with rho=0.5") wssplot(df)
library(NbClust)
nc <- NbClust(df, min.nc=2, max.nc=15, method="kmeans")
par(opar)
barplot(table(nc$Best.n[1,]),
xlab="Numer of Clusters", ylab="Number of Criteria",
main ="Number of Clusters Chosen by 26 Criteria") library(ggplot2)
library(cluster)
fit <- pam(df, k=2)
df$clustering <- factor(fit$clustering)
ggplot(data=df, aes(x=V1, y=V2, color=clustering, shape=clustering)) +
geom_point() + ggtitle("Clustering of Bivariate Normal Data") plot(nc$All.index[,4], type="o", ylab="CCC",
xlab="Number of clusters", col="blue")
吴裕雄--天生自然 R语言开发学习:聚类分析的更多相关文章
- 吴裕雄--天生自然 R语言开发学习:R语言的安装与配置
下载R语言和开发工具RStudio安装包 先安装R
- 吴裕雄--天生自然 R语言开发学习:数据集和数据结构
数据集的概念 数据集通常是由数据构成的一个矩形数组,行表示观测,列表示变量.表2-1提供了一个假想的病例数据集. 不同的行业对于数据集的行和列叫法不同.统计学家称它们为观测(observation)和 ...
- 吴裕雄--天生自然 R语言开发学习:导入数据
2.3.6 导入 SPSS 数据 IBM SPSS数据集可以通过foreign包中的函数read.spss()导入到R中,也可以使用Hmisc 包中的spss.get()函数.函数spss.get() ...
- 吴裕雄--天生自然 R语言开发学习:使用键盘、带分隔符的文本文件输入数据
R可从键盘.文本文件.Microsoft Excel和Access.流行的统计软件.特殊格 式的文件.多种关系型数据库管理系统.专业数据库.网站和在线服务中导入数据. 使用键盘了.有两种常见的方式:用 ...
- 吴裕雄--天生自然 R语言开发学习:R语言的简单介绍和使用
假设我们正在研究生理发育问 题,并收集了10名婴儿在出生后一年内的月龄和体重数据(见表1-).我们感兴趣的是体重的分 布及体重和月龄的关系. 可以使用函数c()以向量的形式输入月龄和体重数据,此函 数 ...
- 吴裕雄--天生自然 R语言开发学习:基础知识
1.基础数据结构 1.1 向量 # 创建向量a a <- c(1,2,3) print(a) 1.2 矩阵 #创建矩阵 mymat <- matrix(c(1:10), nrow=2, n ...
- 吴裕雄--天生自然 R语言开发学习:图形初阶(续二)
# ----------------------------------------------------# # R in Action (2nd ed): Chapter 3 # # Gettin ...
- 吴裕雄--天生自然 R语言开发学习:图形初阶(续一)
# ----------------------------------------------------# # R in Action (2nd ed): Chapter 3 # # Gettin ...
- 吴裕雄--天生自然 R语言开发学习:图形初阶
# ----------------------------------------------------# # R in Action (2nd ed): Chapter 3 # # Gettin ...
- 吴裕雄--天生自然 R语言开发学习:基本图形(续二)
#---------------------------------------------------------------# # R in Action (2nd ed): Chapter 6 ...
随机推荐
- [Algo] 118. Array Deduplication IV
Given an unsorted integer array, remove adjacent duplicate elements repeatedly, from left to right. ...
- PAT Advanced A1104 Sum of Number Segments (20) [数学问题]
题目 Given a sequence of positive numbers, a segment is defined to be a consecutive subsequence. For e ...
- 寒假day05-spring框架
1.回顾事务 l 事务:一组业务操作ABCD,要么全部成功,要么全部不成功. l 特性:ACID 原子性:整体 一致性:完成 隔离性:并发 持久性:结果 l 隔离问题: 脏读:一个事务读到另一个事务没 ...
- MySQL笔记(一)
MySQL是目前最流行的关系型数据库管理系统之一,它是由MySQL AB公司开发.发布并支持.MySQL是一个跨平台的开源关系型数据库管理系统,被广泛地应用在internet上的中小型网站开发中.相比 ...
- Excel文件比较工具的使用
本工具用于比较两个文件夹中对应Excel工作簿中单元格数据是否不同. 如果有内容不同的单元格,就在结果报告中表示出来. 点击如下链接,下载. Excel文件比较工具.rar 解压缩后,看到1个exe文 ...
- 描述符(\_\_get\_\_和\_\_set\_\_和\_\_delete\_\_)
描述符(__get__和__set__和__delete__) 一.描述符 描述符是什么:描述符本质就是一个新式类,在这个新式类中,至少实现了__get__(),set(),delete()中的一个, ...
- SQL:找到特定日期每个顾客最高购买量:Find the highest purchase amount ordered by the each customer on a particular date, with their ID, order date and highest purchase amount.
A: SELECT customer_id,ord_date,MAX(purch_amt) FROM orders GROUP BY customer_id,ord_date; find the hi ...
- python爬虫破解带有CryptoJS的aes加密的反爬机制
发现问题 在一次偶然中,在爬取某个公开网站(非商业型网站)时,老方法,打开调试工具查看请求方式,请求拦截,是否是异步加载,不亦乐乎,当我以为这个网站非常简单的时候,发现二级网页的地址和源码不对应 Aj ...
- dubbo的重试原则
验证思路.使用超时来验证重试次数 XML 注解
- hibernate中session.flush()
flush()session flush在commit之前默认都会执行, 也可以手动执行,他主要做了两件事: 1) 清理缓存. 2) 执行SQL. flush: Session 按照缓存中对象属性变化 ...
