20180711-统计PDB中的蛋白质种类、膜蛋白文件个数及信息等
20180710完成这份工作。简单,但是完成了还是很开心。在我尝试如何使用pickle保存数据后,尝试保存PDB文件中“HEADER”中的信息。文件均保存于实验室服务器(97.73.198.168)/home/RaidDisk/BiodataTest/zyh_pdb_test/tests路径下。本文将记录流程并分享统计结果。
先插入一段代码看看
from PDBParseBase import PDBParserBase
import os
import json
import datetime
from DBParser import ParserBase
import pickle def length_counter(seqres_info):
pdb_id = seqres_info['pdb_id']
number = 0
for item in seqres_info.keys():
if item != 'pdb_id' and item != 'SEQRES_serNum':
number += int(seqres_info[item]['SEQRES_numRes'])
pass
#print(pdb_id)
#print(number)
id_and_lenth = []
id_and_lenth.append(pdb_id)
id_and_lenth.append(number)
return id_and_lenth def find_all_headers(rootdir,saveFilePath,saveFilePath2):
#with the help of PDBParserBase,just put HEADER inf into a pickle with the form of list
pdbbase = PDBParserBase()
start = datetime.datetime.now()
count = 0
f = open(saveFilePath,'wb')
f2 = open(saveFilePath2,'wb')
header_info_all = []
for parent,dirnames,filenames in os.walk(rootdir):
#print(dirnames)
#print(filenames)
for filename in filenames:
count = count + 1
try:
PDBfile = filename
#print(filename)
header_info = pdbbase.get_header_info(os.path.join(parent,filename))
#print(header_info)
header_info_all.append(header_info)
if count %1000== 0:
print(count)
end = datetime.datetime.now()
print (end-start)
pass
except:
print(filename)
end = datetime.datetime.now()
print (end-start)
pickle.dump(filename, f2)
#pdb1ov7.ent
else:
if count %1111 == 0:
print(count)
pickle.dump(header_info_all, f,protocol=2)
end = datetime.datetime.now()
print (end-start) print("Done")
return header_info_all def hd_ctr_clsfcsh(listpath):
#header_counter_classfication
#return a dic ,the key is the classfication and the value is the num
with open(listpath, 'rb') as f:
header_list = pickle.load(f)
#print(header_list)
dic = {}
for item in header_list:
classification = item['HEADER_classification']
print (classification)
if 'MEMBRAN' in classification:
if classification in dic.keys():
dic[classification] = dic[classification] +1
else:
dic[classification] = 1
else:
pass dict= sorted(dic.items(), key=lambda d:d[1], reverse = True)
print(dict)
return dict def get_filenames(listpath,keyword,resultpath):
with open(listpath, 'rb') as f:
header_list = pickle.load(f)
filenames = []
for item in header_list:
classification = item['HEADER_classification']
if keyword in classification:
#print(item)
#print (item['pdb_id'])
#print (classification)
id = item['pdb_id']
filenames.append(id) else:
#print ('dad')
pass print(len(filenames))
print(filenames)
f2 = open(resultpath,'wb')
pickle.dump(filenames, f2,protocol=2)
print("done")
return filenames if __name__ == "__main__":
rootdir = "/home/BiodataTest/updb"
#1 is to save list 2 is to save some wrang message
saveFilePath = "/home/BiodataTest/test_picale/header_counter.pic"
saveFilePath2 = "/home/BiodataTest/test_picale/header_counter_wrang.pic"
#all_header_list = find_all_headers(rootdir,saveFilePath,saveFilePath2) HEADER_LIST_FILE = "/home/BiodataTest/test_picale/header_counter_list.pic"
hd_ctr_clsfcsh_dic = hd_ctr_clsfcsh(HEADER_LIST_FILE) resultpath = "/home/BiodataTest/test_picale/Membrane_Filename_list.pic"
#wanted_filenames = get_filenames(HEADER_LIST_FILE,'MEMBRANE',resultpath)
昨天写的代码,第二天就忘记了。
函数一:find_all_headers(rootdir,saveFilePath,saveFilePath2),输入文件路径,找到PDB文件中所有的“HEADER”信息,其中包括,文件日期、蛋白质种类、蛋白质名字。存入pickle文件中,保存成一个list。难点在于pickle的第三个参数的值是“2”。
函数二:hd_ctr_clsfcsh(listpath):#header_counter_classfication,将刚才输入的list输入,统计出每个分类的数量。输出的是一个字典,键是分类名字,值是该分类的数量。
函数三:get_filenames(listpath,keyword,resultpath),根据关键词获取给定文件的文件名。在这里,我将所有分类这个信息中含有“MEMBRANE”的蛋白质名字找到,保存成列表输出到文件中。(蛋白质名字就是文件名字)
枯燥的代码介绍完了,来点好看的:大饼图。
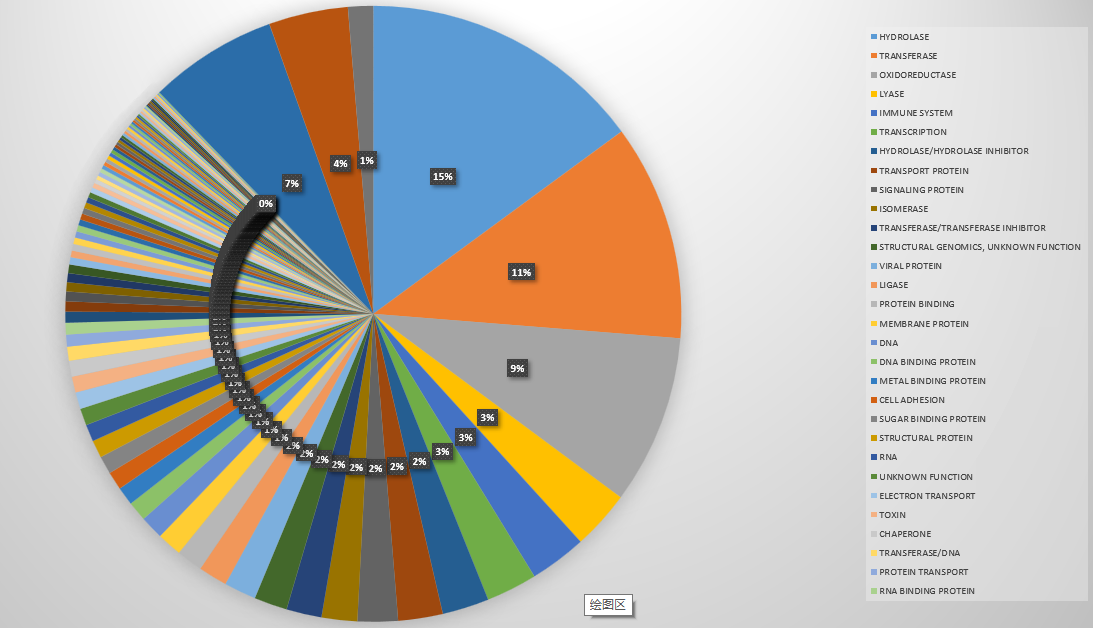
这个饼状图画出了PDB数据库中蛋白质种类的分布,其实是不准确的,比如有的蛋白分别属于膜蛋白和转运蛋白。但是标注为“MEMBRANE PROTEIN, TRANSPORT PROTEIN”,那我们把它归为一类。
我们统计了140946个文件,总共有96(100>x)+400(100>x>9)+1606(10>x>1) +1863(x = 1) = 2965个种类。我们选取前1%看看它们分别是什么:
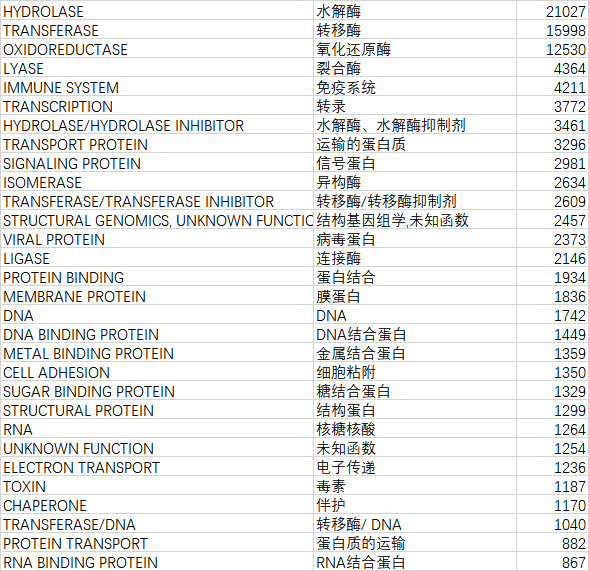
就是它们。总共105057,占总量140946的74.53%。也就是前百分之一的种类占了百分之七十五的数据量。(看起来好残酷的样子哦)
第二部分是有关自己的课题,膜蛋白究竟有多少呢?额,1836个,凄惨的很。所以我扩大了搜索范围,只要包含了“MEMBRANE”的就当作数据提取出来了。注意这个单词的末尾有个“E”,没有"E"的话,数量会增加5个。因为有五个文件叫做“MEMBRANCE PROTEIN”.

找到了它们之后,我们总共获得2335个膜蛋白文件名字,之后将它们解压好,放在预期的文件夹里去。
from PDBParseBase import PDBParserBase
import os
import json
import datetime
from DBParser import ParserBase
#import DataStorage as DS
import time, os,datetime,logging,gzip,configparser,pickle def UnzipFile(fileName,unzipFileName):
""""""
try:
zipHandle = gzip.GzipFile(mode='rb',fileobj=open(fileName,'rb'))
with open(unzipFileName, 'wb') as fileHandle:
fileHandle.write(zipHandle.read()) except IOError:
raise("Unzip file failed!") def mkdir(path):
#Created uncompress path folder
isExists=os.path.exists(path)
if not isExists:
os.makedirs(path)
print(path + " Created folder sucessful!")
return True
else:
#print ("this path is exist")
return False if __name__ == "__main__": #rootdir = "/home/BiodataTest/updb"
#rootdir = "/home/BiodataTest/pdb"
rootdir = "/home/BiodataTest/membraneprotein" count = 0
countcon = 0
start = datetime.datetime.now()
saveFilePath = "/home/BiodataTest/picale_zyh_1000/picale.pic"
#Storage = DS.DataStorage('PDB_20180410') mempt_path = "/home/BiodataTest/test_picale/Membrane_Filename_list.pic"
with open(mempt_path, 'rb') as f:
memprtn_file = pickle.load(f)
print(memprtn_file) count = 0
for parent,dirnames,filenames in os.walk(rootdir):
for filename in filenames:
count = count + 1 #start unzip,get the target name and make a files
dirname = filename[4:6]
filename_for_membrane_1 = filename[3:7]
filename_for_membrane = filename_for_membrane_1.upper()
print(filename_for_membrane)
if(filename_for_membrane in memprtn_file):
print(filename_for_membrane)
filename_with_rootdir = "/home/BiodataTest/pdb/pdb/" + str(dirname)+"/"+str(filename)
unzipFileName = "/home/BiodataTest/membraneprotein/" + str(dirname)+"/"+str(filename)
mkdir("/home/BiodataTest/membraneprotein/" + str(dirname)+"/")
try:
UnzipFile(filename_with_rootdir,unzipFileName[:-3])
pass
except: continue
else:
#print(filename)
pass pass
#print(filename) end = datetime.datetime.now()
print("alltime = ")
print (end-start)
print(count)
print("Done")
于是就获得了这些膜蛋白。
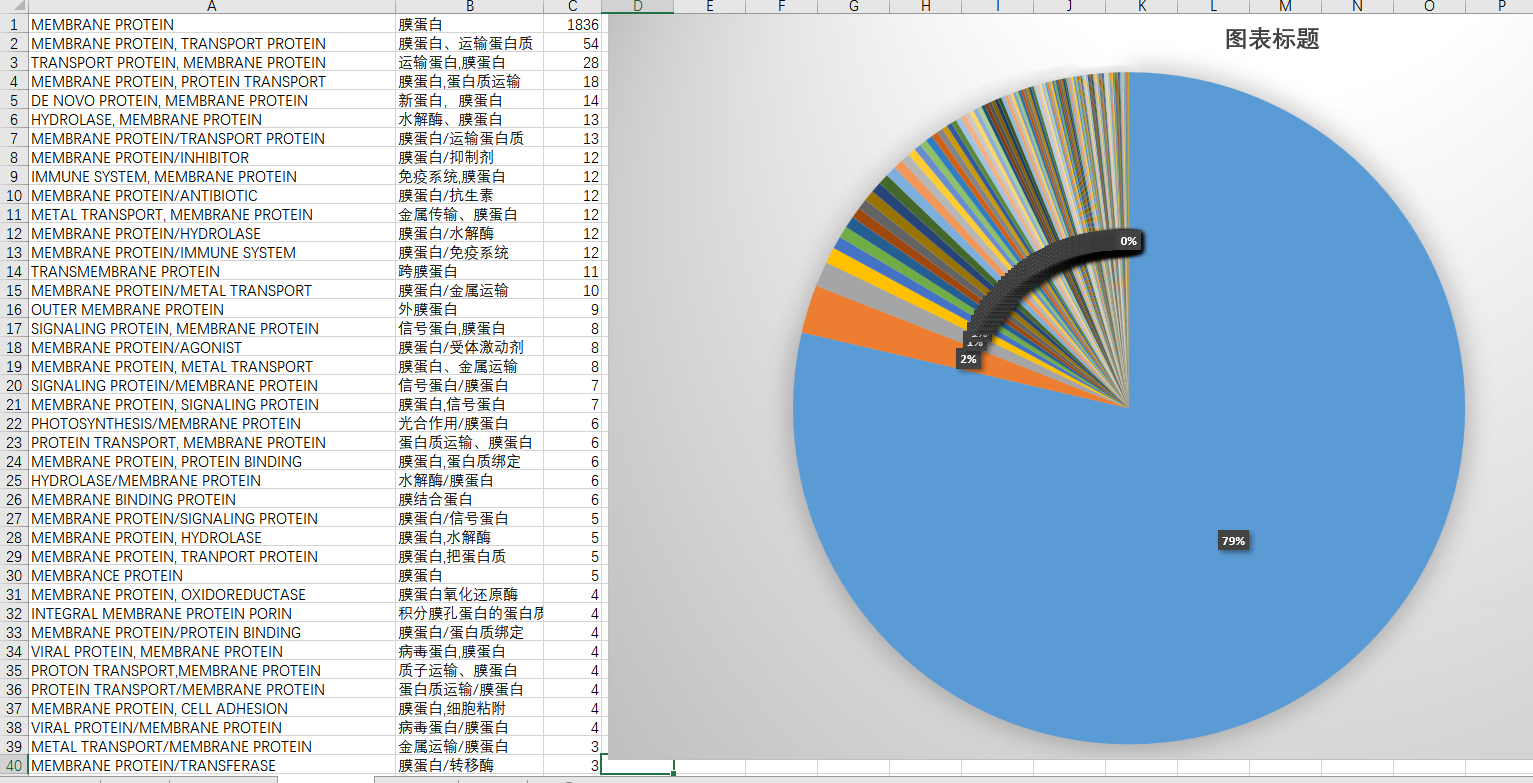
第三十行“MEMBRANCE”的多字母变体英文就大剌剌的显示在那里。。。总结工作也是需要做好的,不然可能之后会忘记吧。
20180711-统计PDB中的蛋白质种类、膜蛋白文件个数及信息等的更多相关文章
- 通过java api统计hive库下的所有表的文件个数、文件大小
更新hadoop fs 命令实现: [ss@db csv]$ hadoop fs -count /my_rc/my_hive_db/* 18/01/14 15:40:19 INFO hdfs.Peer ...
- js统计字符串中各种字符情况
问题描述:在一个字符串中,统计出大写字母.小写字母.数字和其他字符各数.这个算法以前在学习java的时候,老师说过,而且说了四种算法.在孔乙己的世界里,茴香豆的"茴"字有四种写法嘛 ...
- PHP array_count_values() 函数用于统计数组中所有值出现的次数。
定义和用法 array_count_values() 函数用于统计数组中所有值出现的次数. 本函数返回一个数组,其元素的键名是原数组的值,键值是该值在原数组中出现的次数. 语法 array_count ...
- 华为OJ平台——统计字符串中的大写字母
题目描述: 统计字符串中的大写字母的个数 输入: 一行字符串 输出: 字符串中大写字母的个数(当空串时输出0) 思路: 这一题很简单,直接判断字符串中的每一个字符即可,唯一要注意的一点是输入的字符串可 ...
- c程序设计语言_习题1-13_统计输入中单词的长度,并且根据不同长度出现的次数绘制相应的直方图
Write a program to print a histogram of the lengths of words in its input. It is easy to draw the hi ...
- Java基础知识强化之集合框架笔记61:Map集合之统计字符串中每个字符出现的次数的案例
1. 首先我们看看统计字符串中每个字符出现的次数的案例图解: 2. 代码实现: (1)需求 :"aababcabcdabcde",获取字符串中每一个字母出现的次数要求结果:a(5) ...
- 用SQL实现统计报表中的"小计"与"合计"的方法详解
本篇文章是对使用SQL实现统计报表中的"小计"与"合计"的方法进行了详细的分析介绍,需要的朋友参考下 客户提出需求,针对某一列分组加上小计,合计汇总.网上找 ...
- JAVA 统计字符串中中文,英文,数字,空格的个数
面试题:输入一行字符,分别统计出其中英文字母.中文字符.空格.数字和其它字符的个数 可以根据各种字符在Unicode字符编码表中的区间来进行判断,如数字为'0'~'9'之间,英文字母为'a'~'z'或 ...
- Python统计列表中的重复项出现的次数的方法
本文实例展示了Python统计列表中的重复项出现的次数的方法,是一个很实用的功能,适合Python初学者学习借鉴.具体方法如下:对一个列表,比如[1,2,2,2,2,3,3,3,4,4,4,4],现在 ...
随机推荐
- 1、Orcal下载安装步骤图文详解
1.Orcal官方下载地址: https://www.oracle.com/technetwork/cn/database/enterprise-edition/downloads/index.htm ...
- ZooKeeper(一)基本介绍
本文转载自LDB's Blog,原文链接 ZooKeeper原理及其在Hadoop和HBase中的应用 目录 一.简介 二.基本概念 1. 集群角色 2. 会话(Session) 3. 数据节点(ZN ...
- vbs获取当前主机IP
Function GetIP GetIP = "" Dim objWMIService, colAdapters, objAdapter strComputer ...
- oracle快速学习
- iOS Class结构分析
objc_class结构体 类在OC中是objc_class的结构体指针 typedef struct objc_class *Class; 在objc/runtime.h中objc_class结构体 ...
- canvas 绘制刮刮卡
思路=> 用div来展示刮奖结果,用canvas绘制刮奖前展示的图片或者文字:将canvas叠在div上方,刮奖是只需要操作canvas配合touch事件即可简单完成. canvas刮奖可以用g ...
- koa2实现简单的图片上传
1.安装koa-body 2.引入koa-body const koa = require('koa'); const fs = require('fs'); const koaBody = requ ...
- PHP 回调函数call_user_func和 call_user_func_array()的理解
call_user_func(function,param); // 第一个参数是回调函数的函数名,第二个参数是参数 call_user_func函数类似于一种特别的调用函数的方法.其主要有以下的类型 ...
- 10-[协程] greenlet模块、 gevent模块
1.greenlet模块:实现20个任务切换 如果我们在单个线程内有20个任务,要想实现在多个任务之间切换,使用greenlet模块可以非常简单地实现这20个任务直接的切换 使用yield生成器的方式 ...
- 一个java程序员该具备的工具
http://itindex.net/detail/37115-java-%E7%A8%8B%E5%BA%8F%E5%91%98-%E5%B7%A5%E5%85%B7 文章很详细.
