单细胞分析实录(2): 使用Cell Ranger得到表达矩阵
Cell Ranger是一个“傻瓜”软件,你只需提供原始的fastq文件,它就会返回feature-barcode表达矩阵。为啥不说是gene-cell,举个例子,cell hashing数据得到的矩阵还有tag行,而列也不能肯定就是一个cell,可能考虑到这个才不叫gene-cell矩阵吧~它是10xgenomics提供的官方比对定量软件,有四个子命令,我只用过cellranger count,另外三个cellranger mkfastq、cellranger aggr、cellranger reanalyze没用过,也没啥影响。
下载:https://support.10xgenomics.com/single-cell-gene-expression/software/downloads/latest
安装:https://support.10xgenomics.com/single-cell-gene-expression/software/pipelines/latest/installation
在讲Cell Ranger的使用之前,先来看一下10X的单细胞数据长什么样
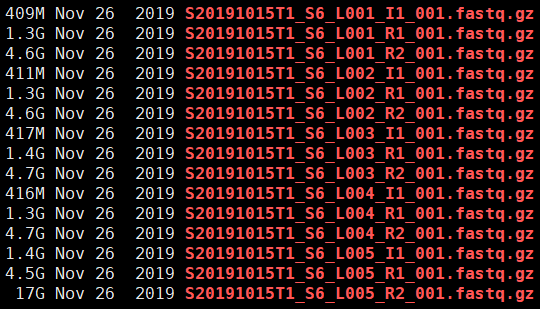
这是一个样本5个Line的测序数据,数据量足够的话可能只有一个Line。可以看出,它们的命名格式相对规范,在收到公司的数据后,尽量不要自己更改命名。此外还要注意一个细节,就是存放这些fastq文件的目录应该用第一个下划线_前面的字符串命名,否则后续cell ranger将无法识别目录里面的文件,同时报错
[error] Unable to detect the chemistry for the following dataset.
Please validate it and/or specify the chemistry
via the --chemistry argument.
其实并不是--chemistry参数的问题。
为了更清楚地理解文件内容,我们来看一下10X单细胞的测序示意图

Read1那一段序列原本是连在磁珠上面的,有cellular barcode(一个磁珠上都一样),有UMI(各不相同),还有poly-T。Read2就是来源于细胞内的RNA。它俩连上互补配对之后,还会在Read2的另一端连上sample index序列。这段sample index序列的作用是什么呢?可以参考illumina测序中index primers的作用:

简单来说就是为了在一次测序中,测多个样本,在来源于特定样本的序列后都加上特定的index,测完之后根据对应关系拆分。一个样本对应4个index:

再看每个文件里面是什么就容易理解了,我们以一个Line为例:
less -S S20191015T1_S6_L001_I1_001.fastq.gz | head -n 8
less -S S20191015T1_S6_L001_R1_001.fastq.gz | head -n 8
less -S S20191015T1_S6_L001_R2_001.fastq.gz | head -n 8
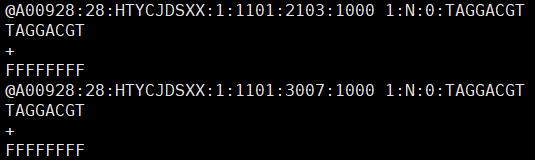
其实这个index序列就包含在文件的第1、5、9...行,有点多余,一般不太关注它。这个文件的序列最多四种,感兴趣的小伙伴可以看看。

R1文件里面就是cellular barcode信息,多余的序列已经去掉了。10X的v2试剂碱基长度是26,v3试剂碱基长度是28

最后一个文件就是真正的转录本对应的cDNA序列
上一篇讲到cell hashing测序有转录本信息,得到的文件和上面是一样的;还有一个细胞表面蛋白信息,根据这个蛋白信息区分细胞来源,如下:

从图中可以看出,和普通转录本建库差不多,就是R2那一部分换成了HTO序列,整个片段长度也改变了。


上面两张图是我在实际处理中看到的两种cell hashing测序,第一张是TotalSeqA,第二张是TotalSeqB。TotalSeqA中,R2第一个碱基开始为HTO序列(之后是polyA序列),而TotalSeqB中,R2前10个碱基为N的任意碱基,第11个碱基为HTO序列的开始位置,HTO序列长度为16。
综上,cell hashing的测序数据有两套,一套是常规的转录本fastq,一套是蛋白信息(也可以说是样本信息)的fastq。所以处理这类数据,要跟测序公司确认清楚用的是TotalSeqA还是B,以及样本和HTO序列的对应关系。
接下来说说如何用Cell Ranger处理普通10X单细胞测序数据,以及cell hashing单细胞测序数据
普通10X
indir=/project_2019_11/data/S20191015T1
outdir=/project_2019_11/cellranger/
sample=S20191015T1
ncells=5000 #预计细胞数,这个参数对最终能得到的细胞数影响并不大,所以不用纠结
threads=20
refpath=/ref/10x/human/refdata-cellranger-GRCh38-3.0.0
cellranger=/softwore/bin/cellranger
cd ${outdir}
${cellranger} count --id=${sample} \
--transcriptome=${refpath} \
--fastqs=${indir} \
--sample=${sample} \
--expect-cells=${ncells} \
--localcores=${threads}
cell hashing
total_seq_A
需要提前准备好两个文件夹,比如我用total_seq_A或total_seq_B存放HTO序列和样本来源的对应关系:
$ ls
feature.reference1.csv
$ cat feature.reference1.csv
id,name,read,pattern,sequence,feature_type
tag1,tag1,R2,^(BC),GTCAACTCTTTAGCG,Antibody Capture
tag2,tag2,R2,^(BC),TGATGGCCTATTGGG,Antibody Capture
tag1、tag2对应哪一个样本事先知道;^(BC)可以看做正则表达式,表示R2序列以barcode(也就是HTO序列)开始
total_seq_B
$ ls
feature.reference.csv
$ cat feature.reference.csv
id,name,read,pattern,sequence,feature_type
tag6,tag6,R2,5PNNNNNNNNNN(BC)NNNNNNNNN,GGTTGCCAGATGTCA,Antibody Capture
tag7,tag7,R2,5PNNNNNNNNNN(BC)NNNNNNNNN,TGTCTTTCCTGCCAG,Antibody Capture
5PNNNNNNNNNN(BC)NNNNNNNNN表示从5端开始,10个碱基之后就是HTO序列,后面的序列随意
lib_csv
第二个文件夹lib_csv,用来存放cell hashing两套数据的路径,用csv格式存储,sample这一列为文件夹名称
$ cat S20200612P1320200702N.libraries.csv
fastqs,sample,library_type
/project_2019_11/data/fastq/,S20200612P1320200702N,Gene Expression
/project_2019_11/data/antibody_barcode/,S20200612P13F20200702N,Antibody Capture
最终脚本如下
lib_dir=/script/cellranger/1/lib_csv/
#need to be changed based on your seq-tech: total_seq_A or total_seq_B
feature_ref_dir=/script/cellranger/1/total_seq_A/
outdir=/project_2019_11/cellranger/
sample=S20191017P11
ncells=5000
threads=20
refpath=/ref/10x/human/refdata-cellranger-GRCh38-3.0.0
cellranger=/softwore/bin/cellranger
cd ${outdir}
${cellranger} count --libraries=${lib_dir}${sample}.libraries.csv \
--r1-length=28 \
--feature-ref=${feature_ref_dir}feature.reference1.csv \
--transcriptome=${refpath} \
--localcores=${threads} \
--expect-cells=${ncells} \
--id=${sample}
最终的表达矩阵会输出到
${outdir}${sample_id}/outs/filtered_feature_bc_matrix
$ cd S20200619P11120200716NC/outs/filtered_feature_bc_matrix/
$ ls
barcodes.tsv.gz features.tsv.gz matrix.mtx.gz
$ less -S features.tsv.gz
ENSG00000243485 MIR1302-2HG Gene Expression
ENSG00000237613 FAM138A Gene Expression
......
ENSG00000277475 AC213203.1 Gene Expression
ENSG00000268674 FAM231C Gene Expression
tag7 tag7 Antibody Capture
tag8 tag8 Antibody Capture
features.tsv.gz存储的是基因信息,因为是cell hashing数据,矩阵最后多了几行tag信息,共33540行
$ less -S barcodes.tsv.gz | head -n 4
AAACCCAAGACTTAAG-1
AAACCCAAGCTACTGT-1
AAACCCAAGGACTGGT-1
AAACCCAAGGCCTGCT-1
barcodes.tsv.gz存放的是最后得到的cellular barcode,共10139行
$ less -S matrix.mtx.gz | head -n 8
%%MatrixMarket matrix coordinate integer general
%metadata_json: {"format_version": 2, "software_version": "3.1.0"}
33540 10139 15746600
65 1 1
103 1 1
155 1 2
179 1 2
191 1 1
matrix.mtx.gz为矩阵信息,除前三行外,余下的行数等于feature乘以CB数,第二列表示CB编号,从1到10139,1重复33540次,对应第一列的33540个feature。第三列表示UMI
下面的脚本可以将这三个文件转换为常见的矩阵形式
path1=/softwore/biosoft/cellranger-3.1.0/cellranger
path2=/project_2019_11/cellranger/
i=S20191211P71
${path1} mat2csv ${path2}${i}/outs/filtered_feature_bc_matrix ${path2}Feature_Barcode_Matrices/${i}.mat.count.csv
sed 's/,/\t/g' ${path2}Feature_Barcode_Matrices/${i}.mat.count.csv > ${path2}Feature_Barcode_Matrices/${i}.mat.count.txt
sed -i 's/^\t//g' ${path2}Feature_Barcode_Matrices/${i}.mat.count.txt
rm -f ${path2}Feature_Barcode_Matrices/${i}.mat.count.csv
单细胞分析实录(2): 使用Cell Ranger得到表达矩阵的更多相关文章
- 单细胞分析实录(1): 认识Cell Hashing
这是一个新系列 差不多是一年以前,我定导后没多久,接手了读研后的第一个课题.合作方是医院,和我对接的是一名博一的医学生,最开始两边的老师很排斥常规的单细胞文章思路,即各大类细胞分群.注释.描述,所以起 ...
- 单细胞分析实录(5): Seurat标准流程
前面我们已经学习了单细胞转录组分析的:使用Cell Ranger得到表达矩阵和doublet检测,今天我们开始Seurat标准流程的学习.这一部分的内容,网上有很多帖子,基本上都是把Seurat官网P ...
- 单细胞分析实录(3): Cell Hashing数据拆分
在之前的文章里,我主要讲了如下两个内容:(1) 认识Cell Hashing:(2): 使用Cell Ranger得到表达矩阵.相信大家已经知道了cell hashing与普通10X转录组的差异,以及 ...
- 单细胞分析实录(8): 展示marker基因的4种图形(一)
今天的内容讲讲单细胞文章中经常出现的展示细胞marker的图:tsne/umap图.热图.堆叠小提琴图.气泡图,每个图我都会用两种方法绘制. 使用的数据来自文献:Single-cell transcr ...
- 【代码更新】单细胞分析实录(20): 将多个样本的CNV定位到染色体臂,并画热图
之前写过三篇和CNV相关的帖子,如果你做肿瘤单细胞转录组,大概率看过: 单细胞分析实录(11): inferCNV的基本用法 单细胞分析实录(12): 如何推断肿瘤细胞 单细胞分析实录(13): in ...
- 【代码更新】单细胞分析实录(21): 非负矩阵分解(NMF)的R代码实现,只需两步,啥图都有
1. 起因 之前的代码(单细胞分析实录(17): 非负矩阵分解(NMF)代码演示)没有涉及到python语法,只有4个python命令行,就跟Linux下面的ls grep一样的.然鹅,有几个小伙伴不 ...
- 单细胞分析实录(4): doublet检测
最近Cell Systems杂志发表了一篇针对现有几种检测单细胞测序doublet的工具的评估文章,系统比较了常见的例如Scrublet.DoubletFinder等工具在检测准确性.计算效率等方面的 ...
- 单细胞分析实录(18): 基于CellPhoneDB的细胞通讯分析及可视化 (上篇)
细胞通讯分析可以给我们一些细胞类群之间相互调控/交流的信息,这种细胞之间的调控主要是通过受配体结合,传递信号来实现的.不同的分化.疾病过程,可能存在特异的细胞通讯关系,因此阐明这些通讯关系至关重要. ...
- 单细胞分析实录(17): 非负矩阵分解(NMF)代码演示
本次演示使用的数据来自2017年发表于Cell的头颈鳞癌单细胞文章:Single-Cell Transcriptomic Analysis of Primary and Metastatic Tumo ...
随机推荐
- java集合类(新手也能掌握)
文章目录 1.集合概述 (1)集合: (2)集合分类: 2.Collection接口 3.List接口 (1)List接口简介 (2)ArrayList集合 (3)LinkedList集合 (4)It ...
- 单调栈模板 POJ3250
上次二分st表大法失败以后的又一次尝试233333 封装,封装,封装!!!!!! #include <bits/stdc++.h> using namespace std; #define ...
- CentOS下Mysql的操作
重启Mysql的各种方法 1.通过rpm包安装的MySQL service mysqld restart /etc/inint.d/mysqld start 2.从源码包安装的MySQL // lin ...
- PyQt(Python+Qt)学习随笔:containers容器部件GroupBox分组框介绍
老猿Python博文目录 专栏:使用PyQt开发图形界面Python应用 老猿Python博客地址 1.主要属性 GroupBox分组框是一个对多个部件进行编组的框架容器,可以带有标题(title属性 ...
- PyQt(Python+Qt)学习随笔:Qt Designer中toolBar的toolButtonStyle属性
tooButtonStyle属性保存主工具栏按钮的样式设置,用来表示工具栏按钮的文字和图标怎么显示. 该属性的可设置值类型为枚举类型Qt.ToolButtonStyle,它包含如下值: 该属性的缺省值 ...
- Xray批量化自动扫描
关于Xray高级版破解: https://www.cnblogs.com/Cl0ud/p/13884206.html 不过好像新版本的Xray修复了破解的BUG,亲测Xray1.3.3高级版仍然可以破 ...
- 实验吧 Once more
0x1函数解析 ereg(): *用指定的模式搜索一个字符串中指定的字符串,如果匹配成功返回true,否则,则返回false. 搜索字母的字符是大小写敏感的. * 此函数存在两个漏洞: ①%00截断及 ...
- 【面试】java什么时候要用static
static关键字是在我们编写代码和阅读代码时碰到的常见的一个关键字,在学习java基础时就学过了,这也是各大公司的面试官喜欢在面试时问到的知识点之一.虽然大概知道是什么,但完整的表达出来还是有点难度 ...
- springboot中过滤器、拦截器、切片使用
直接贴代码:采用maven工程 pom.xml <?xml version="1.0" encoding="UTF-8"?> <project ...
- .net5+nacos+ocelot 配置中心和服务发现实现
最近一段时间 因公司业务需要,需要使用.net5做一套微服务的接口,使用nacos 做注册中心和配置中心,ocelot做网关. 因为ocelot 支持的是consol和eureka,如果使用nacos ...
