结合MATLAB、Python、R语言,在求得显著差异的边(节点对)之后,怎么画circle图
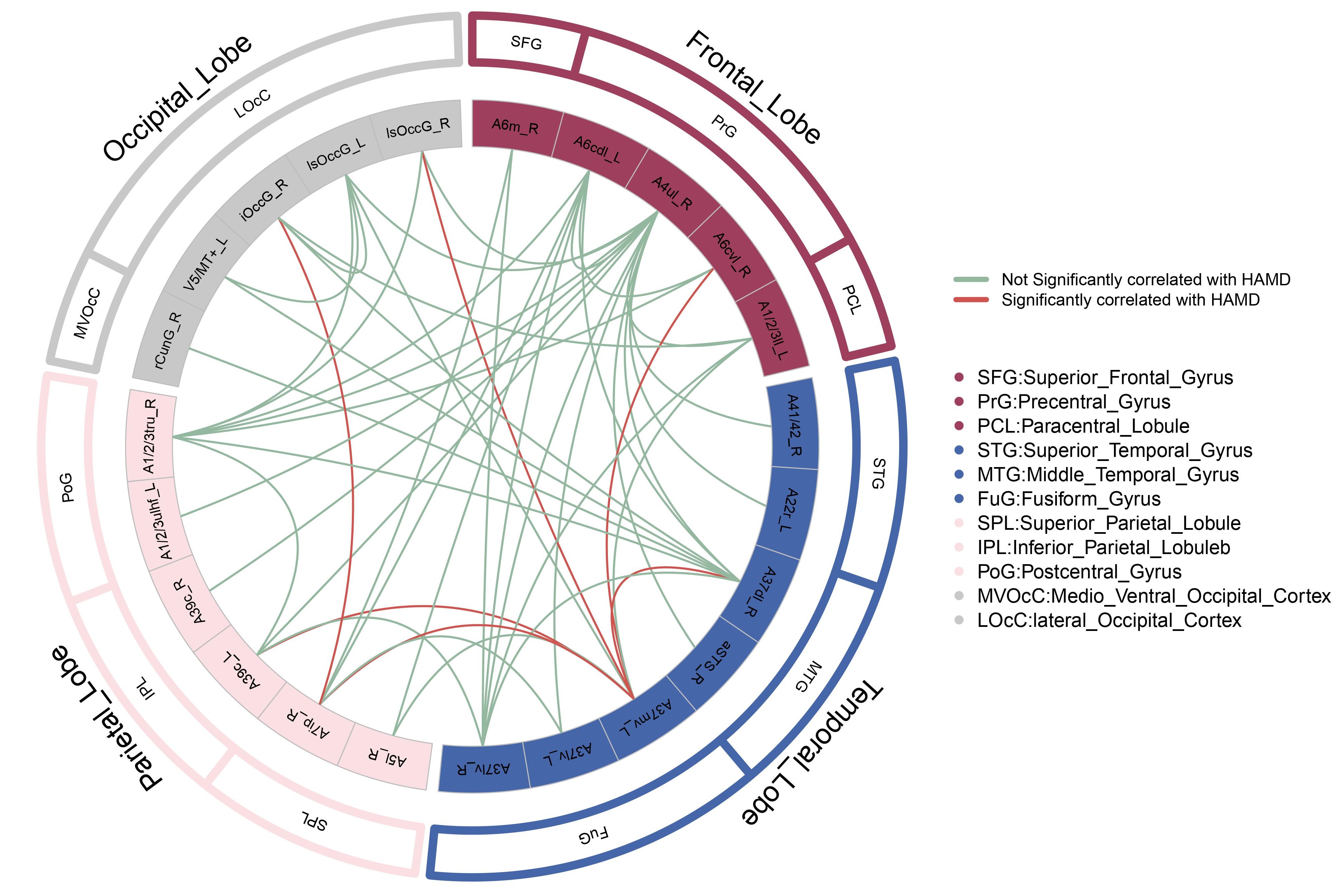
先来看看成果图:
OK,开始画图:
实验背景声明:在脑影像分析中,我们首先构建脑网络,然后使用双样本t对比两组人的连接差异,然后使用以上的图进行可视化,一般红色连接代表显著升高,绿色代表显著下降。(非必须,根据实际需求设计,如上图中红色代表相应的连接差异与HAMD抑郁量表评分显著相关,绿色表示不相关)。这里呢,我们研究了一组病人以及年龄性别匹配的健康被试的fMRI的数据,首先进行fMRI预处理,然后基于中科院246模板提取ROISignals,然后构建脑网络(细节不做介绍)。最后进行连接差异对比。然后得到病人组相对于正常被试组有哪些节点之间的连接权重出现了显著降低或显著升高。
Step1: R语言的画图代码MyCircle.R,这里我们先给出代码,根据代码的文件需求,准备材料。在以下代码中,开头即指明了需要的材料,有
LobeID.txt;
GyrusID.txt;
ROIID.txt;
Links.txt
LobeID <- read.table("./Circle edges/LobeID.txt", header = TRUE, sep = "\t")
head(LobeID)
GeneID <- read.table("./Circle edges/GyrusID.txt", header = TRUE, sep = "\t")
head(GeneID)
GeneFe <- read.table("./Circle edges/ROIID.txt", header = TRUE, sep = "\t")
head(GeneFe)
Links <- read.table("./Circle edges/Links.txt", header = TRUE, sep = "\t")
head(Links)
LobeID$LobeID <- factor(LobeID$LobeID, levels = LobeID$LobeID)
library(stringi)
library(circlize)
pdf("circlize1.pdf",width = 17.5,height = 10)
par(mar=rep(0,4))
circos.clear()
circos.par(start.degree = 90, #从哪里开始画,沿着逆时针顺序
gap.degree = 2, #每两个sectors间的间隔大小,此处是每两个组间的间隔
track.margin = c(0,0.08), #gap.degree规定了左右,track.margin规定上下
cell.padding = c(0,0,0,0) #?
)
#### 根据"LobeID.txt"画组
#图是多个组围成的一个圈,其中每个组及其亚组都画在一个单独的坐标系里
circos.initialize(factors = LobeID$LobeID,
xlim = cbind(LobeID$LobeStart, LobeID$LobeEnd))
circos.trackPlotRegion(ylim = c(0, 1), factors = LobeID$LobeID, track.height=0.1,
#panel.fun for each sector
panel.fun = function(x, y) {
#select details of current sector
name <- get.cell.meta.data("sector.index")
i <- get.cell.meta.data("sector.numeric.index")
xlim <- get.cell.meta.data("xlim")
ylim <- get.cell.meta.data("ylim")
#组ID文字的设置
circos.text(x = mean(xlim),
y = 1,
labels = name,
cex = 1.7, #组ID文字大小
#col = "darkgrey", #组ID的颜色
adj = c(0.5,-0.8)
#如果想让组ID呈放射状排列,就用下面三行代替上面那行
#niceFacing = TRUE, #组ID头朝上
#facing = "reverse.clockwise",
#adj = c(1.5, -0.8) #组ID的位置
)
#画组
circos.rect(xleft = xlim[1],
ybottom = ylim[1],
xright = xlim[2],
ytop = ylim[2],
col = "blue",
border = "black")
})
#### 根据"GyrusID.txt"画亚组
library(graphics)
#bgcol <- rainbow(sum(LobeID$LobeEnd), s = 1, v = 1, start = 0, end = max(1, sum(LobeID$LobeEnd) - 1)/sum(LobeID$LobeEnd), alpha = 1)
for (i in seq_len(nrow(GeneID))){
ylim <- c(0, 1)
circos.rect(sector.index = GeneID$LobeID[i],
track.index = 1,
xleft = GeneID$featureStart[i], # + 0.01, #让feature之间流出空隙
ybottom = ylim[1],
xright = GeneID$featureEnd[i], # - 0.01, #让feature之间流出空隙
ytop = ylim[2],
#如果想让每个方块颜色不同,用彩虹色画每一个Gene feature就运行下面这行
#col = bgcol[i],
#如果想让每个组用同一个颜色,调用输入文件中写好的颜色,就运行下面这行
col = "white",
border = paste("#", GeneID$barColor[i], sep = ""), lwd = 8)
}
# 亚组的文字
for (i in seq_len(nrow(GeneID))){
ylim <- c(0, 1)
circos.text((GeneID$featureStart[i] + GeneID$featureEnd[i])/2,
ylim[1] + 0.5,
GeneID$GyrusID[i],
sector.index = GeneID$LobeID[i],
facing = "inside",
cex = 0.9)
}
circos.trackPlotRegion(ylim = c(0, 1), factors = LobeID$LobeID, track.height=0.1,
#panel.fun for each sector
panel.fun = function(x, y) {
#select details of current sector
name <- get.cell.meta.data("sector.index")
i <- get.cell.meta.data("sector.numeric.index")
xlim <- get.cell.meta.data("xlim")
ylim <- get.cell.meta.data("ylim")
#画组
circos.rect(xleft = xlim[1],
ybottom = ylim[1],
xright = xlim[2],
ytop = ylim[2],
col = "blue",
border = "white",lwd = 6)
})
#提需求的小伙伴还想让圆圈图中每个方块颜色不同,整体显示彩虹色,就像circlize_rainbow.pdf那样的效果,就把下面这个for循环里的“col = ...”行换成“col = bgcol[i], ”
#运行下面两行,设置彩虹色
library(graphics)
bgcol <- rainbow(sum(LobeID$LobeEnd), s = 1, v = 1, start = 0, end = max(1, sum(LobeID$LobeEnd) - 1)/sum(LobeID$LobeEnd), alpha = 1)
for (i in seq_len(nrow(GeneFe))){
ylim <- c(0, 1)
circos.rect(sector.index = GeneFe$LobeID[i],
track.index = 2,
xleft = GeneFe$featureStart[i], # + 0.01, #让feature之间流出空隙
ybottom = ylim[1],
xright = GeneFe$featureEnd[i], # - 0.01, #让feature之间流出空隙
ytop = ylim[2],
#如果想让每个方块颜色不同,用彩虹色画每一个Gene feature就运行下面这行
#col = bgcol[i],
#如果想让每个组用同一个颜色,调用输入文件中写好的颜色,就运行下面这行
col = paste("#", GeneFe$barColor[i], sep = ""),
border = "gray")
}
# 亚组的文字
for (i in seq_len(nrow(GeneFe))){
ylim <- c(0, 1)
circos.text(GeneFe$featureStart[i] + 0.5,
ylim[1] + 0.5,
GeneFe$ROIID[i],
sector.index = GeneFe$LobeID[i],
facing = "inside",
cex = 0.8)
}
#### 根据"Links.txt"画连线
for(i in seq_len(nrow(Links))){
circos.link(sector.index1 = Links$ROI_1[i],
point1 = Links[i, 2],
sector.index2 = Links$ROI_2[i],
point2 = Links[i, 4],
#在输入文件中已经写好了连线的颜色
col = paste("#", Links$link_color[i], sep = ""),
#如果不想在输入文件中写颜色,还可以运行下面这行,在这里指定颜色
#col = "red",
border = FALSE,
lwd = 2, #连线的粗细
rou = 0.64, #连线起始和终止点的y值(圆半径的百分比)
h.ratio = 0.7 #连线的拱形高度,数值越大,拱形越低
#h.ratio = Links$link_height[i] #在输入文件中指定拱形高度
)
}
#Legend of brain regions
GeneCol<-data.frame(legendID=GeneID$GyrusID,legendCol=GeneID$barColor)
GeneCol_uniq<-base::unique(GeneCol)
legend(1,0.2,
bty = "n",#不要边框
pch = 16,#画成正方形,圆点是16
cex = 1.2,
legend = c("SFG:Superior_Frontal_Gyrus","PrG:Precentral_Gyrus","PCL:Paracentral_Lobule",
"STG:Superior_Temporal_Gyrus","MTG:Middle_Temporal_Gyrus","FuG:Fusiform_Gyrus",
"SPL:Superior_Parietal_Lobule","IPL:Inferior_Parietal_Lobuleb","PoG:Postcentral_Gyrus",
"MVOcC:Medio_Ventral_Occipital_Cortex","LOcC:lateral_Occipital_Cortex"),
col = paste0("#",GeneCol_uniq$legendCol),
horiz = FALSE)
#Legend of links
LinksCol<-data.frame(Links$link_color)
LinksCol_uniq<-base::unique(LinksCol)
legend(1,0.4,
bty = "n",
lty = 1,#画线
lwd = 5,#线的粗细
legend = c("Not Significantly correlated with HAMD","Significantly correlated with HAMD"),
col = paste0("#",LinksCol_uniq$Links.link_col),
horiz = FALSE)
dev.off()
Step2: 准备材料
以上提到的几个文件,内容是这个样子的:
LobeID.txt;
GyrusID.txt;

ROIID.txt;

Links.txt

根据以上的内容就可以正常使用画图了,如果你不会写代码,就自己一点一点的敲文字就好了。
如果熟悉基本的文本处理,接下来用代码辅助制作材料就可以啦,这里我使用MATLAB+python辅助实现,都是很简单的脚本,
可以提高准确性,提高效率,给大家参考。一下代码不是最好的,跟随着作者的意识流生成的,可优化空间很大,但是一定能达到目标:
Base材料:
edge_for_circles: 代表节点对,及具有显著差异的边
name_246:代表中科院246模板的子节点名字(label)---->http://atlas.brainnetome.org/

衍生材料:
根据以上矩阵,使用以下matlab代码制作衍生材料,然后将edge_all_s存储为mat文件,命名为"edge_num.mat",如下图:
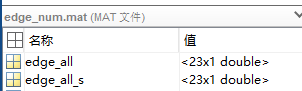
edge_all = unique(edge_for_cirles); %得到独一无二的脑区代号
edge_all_s = sort(edge_all); %对以上脑区编号排个序
edge_s = sortrows(edge_for_cirles); %对节点对进行扩展排序,按照起点的排序进行排序
name_all = name_246(edge_all_s,2); %得到节点名的mat矩阵
(1)LobeID.txt制作过程:
我们需要知道涉及的Lobe有哪些,而上面的代码可以知道所有的子区,根据子区的number,定位Lobe,这里使用了一个python的脚本把相应的名字读出来了,代码中出现的
full_nameOfROIs.csv文件,以及所有的代码文件皆可以在本人github网站下载:https://github.com/chaochaobar/circles_for_brain
import scipy.io as sio
import pandas as pd datafile = './edge_num.mat'
mat = sio.loadmat(datafile)
full_name = pd.read_csv("./full_nameOfROIs.csv")
index_global = mat['edge_all_s']
save_file_name = "for_circle.node" i = 0
for line in open('./node246.node'):
i = i + 1
a = i // 2
b = i % 2
if b == 0:
app = full_name["ROI_R"][a - 1]
elif b == 1:
app = full_name["ROI_L"][a]
if i in index_global:
if i in range(0, 69):
Lobe = "Frontal_Lobe"
if i in range(0, 15):
Gyrus = "Superior_Frontal_Gyrus"
elif i in range(15, 29):
Gyrus = "Middle_Frontal_Gyrus"
elif i in range(29, 41):
Gyrus = "Inferier_Frontal_Gyrus"
elif i in range(41, 53):
Gyrus = "Orbital_Gyrus"
elif i in range(53, 65):
Gyrus = "Precentral_Gyrus"
elif i in range(65, 69):
Gyrus = "Paracentral_Lobule"
elif i in range(69, 125):
Lobe = "Temporal_Lobe"
if i in range(69, 81):
Gyrus = "Superior_Temporal_Gyrus"
elif i in range(81, 89):
Gyrus = "Middle_Temporal_Gyrus"
elif i in range(89, 103):
Gyrus = "Inferior_Temporal_Gyrus"
elif i in range(103, 109):
Gyrus = "Fusiform_Gyrus"
elif i in range(109, 121):
Gyrus = "Parahippocampal_Gyrus"
elif i in range(121, 125):
Gyrus = "posterior_Superior_Temporal_Sulcus"
elif i in range(125, 163):
Lobe = "Parietal_Lobe"
if i in range(125, 135):
Gyrus = "Superior_Parietal_Lobule"
elif i in range(135, 147):
Gyrus = "Inferior_Parietal_Lobule"
elif i in range(147, 155):
Gyrus = "Precuneus"
elif i in range(155, 163):
Gyrus = "Postcentral_Gyrus"
elif i in range(163, 175):
Lobe = "Insular_Lobe"
Gyrus = "Insular_Gyrus"
elif i in range(175, 189):
Lobe = "Limbic_Lobe"
Gyrus = "Cingulate_Gyrus"
elif i in range(189, 211):
Lobe = "Occipital_Lobe"
if i in range(189, 199):
Gyrus = "Medio_Ventral_Occipital_Cortex"
elif i in range(199, 211):
Gyrus = "lateral_Occipital_Cortex"
elif i in range(211, 247):
Lobe = "Subcortical_Nuclei"
if i in range(211, 215):
Gyrus = "Amygdala"
elif i in range(215, 219):
Gyrus = "Hippocampus"
elif i in range(219, 231):
Gyrus = "Basal_Ganglia"
elif i in range(213, 247):
Gyrus = "Thalamus"
print(Lobe, ",", Gyrus, ",", line)
with open(save_file_name, "a+") as f:
f.write("{Lobe} {Gyrus} {line}".format(Lobe = Lobe, Gyrus = Gyrus, line = line))
f.close()
生成了“for_circle.node”文件之后,使用matlab读取,在制作数据材料ROIID.txtyiji Links.txt文件的时候,先用以下matlab代码将每一列的内容生成出来,然后粘贴到excel里面,最后粘贴到txt文件里面,有点绕是吧,这就是意识流,用最快的方法,即便有点蛮力,但是省去了过多的思考,半自动化:
%制作ROIID:先使用python读lobe和gyrus的名字,然后执行下面的语句
[lobe,gyrus,x,y,z,t,name_sub] = textread('for_circle.node','%s %s %d %d %d %f %s'); %%% 制作LobeID, GyrusID, ROIID, LinkID,使用excel
for i = 1:len_sig
index_start = find(edge_all==edge_s(i, 1));
index_end = find(edge_all==edge_s(i, 2));
edges_all_name(i,1:2) = [name_all(index_start), name_all(index_end)];
end %%% start lobe , end lobe
for i = 1:len_sig
cur_start = edges_all_name{i, 1};
index_start = find(strcmp(C,cur_start));
cur_end = edges_all_name{i,2};
index_end = find(strcmp(C, cur_end));
edges_all_name_2(i, 1) = A(index_start, 1);
edges_all_name_2(i, 2) = A(index_end, 1);
end %%% start lobe , end lobe
for i = 1:len_sig
cur_start = edges_all_name{i, 1};
index_start = find(strcmp(C,cur_start));
cur_end = edges_all_name{i,2};
index_end = find(strcmp(C, cur_end));
edges_all_name_2(i, 1) = B(index_start, 1);
edges_all_name_2(i, 2) = B(index_end, 1);
end %%% 制作LinkID的代码
point1 = zeros(len_sig, 1);
point2 = zeros(len_sig, 1);
for i = 1:len_sig
cur_start = edges_all_name{i, 1};
index_start = find(strcmp(C,cur_start));
cur_end = edges_all_name{i,2};
index_end = find(strcmp(C, cur_end));
point1(i, 1) = D(index_start, 1) + 0.5;
point2(i,1) = D(index_end, 1) + 0.5;
end
代码以及数据文件制作过程都有了,动手实践起来吧小伙伴们,有什么问题,欢迎联系:1374015299@qq.com
结合MATLAB、Python、R语言,在求得显著差异的边(节点对)之后,怎么画circle图的更多相关文章
- python R语言 入门常见指令
环境是windows R语言安装包 install.packages("magrittr")
- 第六篇:R语言数据可视化之数据分布图(直方图、密度曲线、箱线图、等高线、2D密度图)
数据分布图简介 中医上讲看病四诊法为:望闻问切.而数据分析师分析数据的过程也有点相似,我们需要望:看看数据长什么样:闻:仔细分析数据是否合理:问:针对前两步工作搜集到的问题与业务方交流:切:结合业务方 ...
- 数学类网站、代码(Matlab & Python & R)
0. math & code COME ON CODE ON | A blog about programming and more programming. 1. 中文 统计学Computa ...
- R语言与医学统计图形-【17】ggplot2几何对象之热图
ggplot2绘图系统--heatmap.geom_rect 这里不介绍更常见的pheatmap包. 1.heatmap函数 基础包. data=as.matrix(mtcars) #接受矩阵 hea ...
- R语言与医学统计图形-【15】ggplot2几何对象之线图
ggplot2绘图系统--几何对象之线图 曲线:点连线.路径曲线.时间序列曲线.模型拟合曲线...... 直线:水平直线.垂直直线.斜线. 1.曲线 对象及其参数. #路径图 geom_path(ma ...
- R语言与医学统计图形-【12】ggplot2几何对象之条图
ggplot2绘图系统--几何对象之条图(包括误差条图) 1.条图 格式: geom_bar(mapping = , data = , stat = 'count', #统计变换默认计数 positi ...
- R语言绘制空间热力图
先上图 R语言的REmap包拥有非常强大的空间热力图以及空间迁移图功能,里面内置了国内外诸多城市坐标数据,使用起来方便快捷. 开始 首先安装相关包 install_packages("dev ...
- 大数据时代的精准数据挖掘——使用R语言
老师简介: Gino老师,即将步入不惑之年,早年获得名校数学与应用数学专业学士和统计学专业硕士,有海外学习和工作的经历,近二十年来一直进行着数据分析的理论和实践,数学.统计和计算机功底强悍. 曾在某一 ...
- R语言︱SNA-社会关系网络 R语言实现专题(基础篇)(一)
每每以为攀得众山小,可.每每又切实来到起点,大牛们,缓缓脚步来俺笔记葩分享一下吧,please~ --------------------------- 笔者寄语:这里所有的应用代码都来自与igrap ...
随机推荐
- SQL Injection (Blind) Low
SQL盲注分析 盲注较普通注入难度会有所增加,根据页面响应不同大概分为以下几种:布尔型盲注:时间盲注:报错注入 普通注入与盲注的对比: 普通注入: ...
- (四)一个bug的生命周期
Bug的属性 Bug重现环境 这个应该是我们重现BUG的一个前提,如果没有这个前提,我们可能会无法重现问题,或者根本就无从下手. • 操作系统 这个是一般软件运行的一大前提,基本上所有的软件都依赖于操 ...
- 使用uniapp开发项目来的几点心得体会,供新手参考参考
先说一下提前须要会的技术 要想快速入手uniapp的话,你最好提前学会vue.微信小程序开发,因为它几乎就是这两个东西的结合体,不然,你就只有慢慢研究吧. 为什么要选择uniapp??? 开发多个平台 ...
- 聚焦LS-MIMO的四大层面,浅谈5G关键技术
摘要:本文简要讲述了5G关键技术之一的LS-MIMO,分别从导频污染.信道估计.预编码技术.信号检测四个部分入手. 导频污染 理想情况下,时分复用(TDD)系统中上行链路和下行链路之间各个导频符号之间 ...
- 容器编排系统之K8s资源标签、标签选择器、资源注解
前文我们聊到了使用k8s资源配置清单定义资源的相关话题,回顾请参考https://www.cnblogs.com/qiuhom-1874/p/14132890.html:今天我们来聊下资源标签,标签选 ...
- PHP字符串你不知道的事
PHP常见的定义字符串的方式有那些? 1.单引号 在单引号中,任何特殊字符都会按原样输出[除\.\'将会被转义输出],不是什么都不解析的,这是很多人的误解 echo 'this is a var!'. ...
- vue第十单元(动态组件 keep-alive(钩子函数) 递归组件(name) 组件命名约定)
第十单元(动态组件 keep-alive(钩子函数) 递归组件(name) 组件命名约定) #课程目标 熟练掌握动态组件的实现 掌握keep-alive缓存组件,以及相应的钩子函数 熟练掌握递归组件, ...
- [OI笔记]后缀自动机
本来没打算写的,不过想想看后缀自动机的理论看了两三天了才有点懂(我太傻了)-下周期末考的话大概要去复习一下文化课感觉回来又要忘得差不多,还是开篇blog记一下好了. 相关的资料: cls当年的课件:2 ...
- 如何push一个docker镜像到DockerHub上
在DockerHub上创建账号:https://hub.docker.com/ 这里我的账号是firewarm 本地下载镜像(这里拿alpine做示例),并为镜像打tag [root@host-30 ...
- Vue - 与后端交互
零:与后端交互 - ajax 版本1 - 出现了跨域问题 前端:index.html <!DOCTYPE html> <html lang="en"> &l ...
