MOEAD实现、基于分解的多目标进化、 切比雪夫方法-(python完整代码)
确定某点附近的点
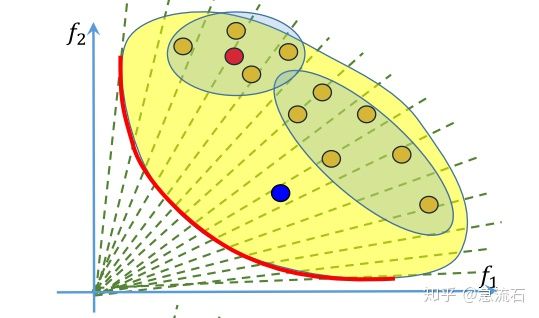
答:每个解对应的是一组权重,即子问题,红点附近的四个点,也就是它的邻居怎么确定呢?由权重来确定,算法初始化阶段就确定了每个权重对应的邻居,也就是每个子问题的邻居子问题。权重的邻居通过欧式距离来判断。取最近的几个。
取均匀分布向量
https://www.cnblogs.com/Twobox/p/16408751.html
MOEAD实现
算法理解与流程
https://www.zhihu.com/question/263555181?sort=created
其中两个回答都挺好的
1. 输入N m
# N表示取点密度 m表示问题维度
1.1 输入 T
# 表示取最近的T个作为邻居
2. 生成均匀分布权重向量
2.1 计算每个权重向量之间的欧拉距离
3. 权重向量个数即为:初始种群个数
4. 初始化种群,每个个体一一对应权重
4.1 更具权重之间距离,取前T个作为邻居person
5. EP = 空
# 维护成最优前沿
6. 计算最初的全局最优Z
# 把每个带入f1 f2中,取最小值 z1 z2
7. 开始循环N代
7.1对于每个个体,在领域中选取2个个体进行交叉变异,获得2个新个体
7.1.1更新全局解z
7.2在领域中随机选择2个个体,用新个与旧个体进行对比
# 新个体带入子目标问题,直接对比值即可
7.3如果更优,则替换旧个体dna
7.4更新EP
# 如果有别接收的新解,将新解与EP每一个进行比较,删除被新解支配的,如果新解没有被旧解支配,那么加入EP
代码实现设计
# 分析
需要维护的数据结构:
某个体最近的T位邻居: 可以考虑采用对象列表即可
均匀分布的权重向量:一个二维ndarray数组即可
权重向量与个体对应关系:个体对象,直接保存权重向量数组
权重向量之间的距离矩阵:开局初始化,不变的
EP list,里面的个体是对象的引用
z list
目标函数集合,F list domain list # 接口设计
class Person
attribute:
dns:一维ndarray
weight_vector: 一维ndarray
neighbor: list<Person>
o_func:Objective_Function 目标函数
function:
mutation
cross_get_two_new_dna:返回2段新dna
compare#与子代比较
accept_new_dna
choice_two_person:p1,p2
class Moead_Util
attribute:
N
M
T:
o_func:Objective_Function
pm:变异概率 EP:[dna1,dna2,..]
weight_vectors:二维数组
Euler_distance:二维数组
pip_size
Z:[] # 这里面元素为一维ndarray数组,即dna,即解 function:
init_mean_vector:二维数组
init_Euler_distance:二维数组
init_population:[]
init_Z:一维属猪 update_ep
update_Z class Objective_Function:
attribute:
F:[]
domain:[[0,1],[],[]]
function:
get_one_function:Objective_Function
Person.py
1 import numpy as np
2
3
4 class Person:
5 def __init__(self, dna):
6 self.dna = dna
7 self.weight_vector = None
8 self.neighbor = None
9 self.o_func = None # 目标函数
10
11 self.dns_len = len(dna)
12
13 def set_info(self, weight_vector, neighbor, o_func):
14 self.weight_vector = weight_vector
15 self.neighbor = neighbor
16 self.o_func = o_func# 目标函数
17
18 def mutation_dna(self, one_dna):
19 i = np.random.randint(0, self.dns_len)
20 low = self.o_func.domain[i][0]
21 high = self.o_func.domain[i][1]
22 new_v = np.random.rand() * (high - low) + low
23 one_dna[i] = new_v
24 return one_dna
25
26 def mutation(self):
27 i = np.random.randint(0, self.dns_len)
28 low = self.o_func.domain[i][0]
29 high = self.o_func.domain[i][1]
30 new_v = np.random.rand() * (high - low) + low
31 self.dna[i] = new_v
32
33 @staticmethod
34 def cross_get_two_new_dna(p1, p2):
35 # 单点交叉
36 cut_i = np.random.randint(1, p1.dns_len - 1)
37 dna1 = p1.dna.copy()
38 dna2 = p2.dna.copy()
39 temp = dna1[cut_i:].copy()
40 dna1[cut_i:] = dna2[cut_i:]
41 dna2[cut_i:] = temp
42 return dna1, dna2
43
44 def compare(self, son_dna):
45 F = self.o_func.f_funcs
46 f_x_son_dna = []
47 f_x_self = []
48 for f in F:
49 f_x_son_dna.append(f(son_dna))
50 f_x_self.append(f(self.dna))
51 fit_son_dna = np.array(f_x_son_dna) * self.weight_vector
52 fit_self = np.array(f_x_self) * self.weight_vector
53 return fit_son_dna.sum() - fit_self.sum()
54
55 def accept_new_dna(self, new_dna):
56 self.dna = new_dna
57
58 def choice_two_person(self):
59 neighbor = self.neighbor
60 neighbor_len = len(neighbor)
61 idx = np.random.randint(0, neighbor_len, size=2)
62 p1 = self.neighbor[idx[0]]
63 p2 = self.neighbor[idx[1]]
64 return p1, p2
Objective_Function
1 from collections import defaultdict
2
3 import numpy as np
4
5
6 def zdt4_f1(x_list):
7 return x_list[0]
8
9
10 def zdt4_gx(x_list):
11 sum = 1 + 10 * (10 - 1)
12 for i in range(1, 10):
13 sum += x_list[i] ** 2 - 10 * np.cos(4 * np.pi * x_list[i])
14 return sum
15
16
17 def zdt4_f2(x_list):
18 gx_ans = zdt4_gx(x_list)
19 if x_list[0] < 0:
20 print("????: x_list[0] < 0:", x_list[0])
21 if gx_ans < 0:
22 print("gx_ans < 0", gx_ans)
23 if (x_list[0] / gx_ans) <= 0:
24 print("x_list[0] / gx_ans<0:", x_list[0] / gx_ans)
25
26 ans = 1 - np.sqrt(x_list[0] / gx_ans)
27 return ans
28
29 def zdt3_f1(x):
30 return x[0]
31
32
33 def zdt3_gx(x):
34 if x[:].sum() < 0:
35 print(x[1:].sum(), x[1:])
36 ans = 1 + 9 / 29 * x[1:].sum()
37 return ans
38
39
40 def zdt3_f2(x):
41 g = zdt3_gx(x)
42 ans = 1 - np.sqrt(x[0] / g) - (x[0] / g) * np.sin(10 * np.pi * x[0])
43 return ans
44
45
46 class Objective_Function:
47 function_dic = defaultdict(lambda: None)
48
49 def __init__(self, f_funcs, domain):
50 self.f_funcs = f_funcs
51 self.domain = domain
52
53 @staticmethod
54 def get_one_function(name):
55 if Objective_Function.function_dic[name] is not None:
56 return Objective_Function.function_dic[name]
57
58 if name == "zdt4":
59 f_funcs = [zdt4_f1, zdt4_f2]
60 domain = [[0, 1]]
61 for i in range(9):
62 domain.append([-5, 5])
63 Objective_Function.function_dic[name] = Objective_Function(f_funcs, domain)
64 return Objective_Function.function_dic[name]
65
66 if name == "zdt3":
67 f_funcs = [zdt3_f1, zdt3_f2]
68 domain = [[0, 1] for i in range(30)]
69 Objective_Function.function_dic[name] = Objective_Function(f_funcs, domain)
70 return Objective_Function.function_dic[name]
Moead_Util.py
1 import numpy as np
2
3 from GA.MOEAD.Person import Person
4
5
6 def distribution_number(sum, m):
7 # 取m个数,数的和为N
8 if m == 1:
9 return [[sum]]
10 vectors = []
11 for i in range(1, sum - (m - 1) + 1):
12 right_vec = distribution_number(sum - i, m - 1)
13 a = [i]
14 for item in right_vec:
15 vectors.append(a + item)
16 return vectors
17
18
19 class Moead_Util:
20 def __init__(self, N, m, T, o_func, pm):
21 self.N = N
22 self.m = m
23 self.T = T # 邻居大小限制
24 self.o_func = o_func
25 self.pm = pm # 变异概率
26
27 self.Z = np.zeros(shape=m)
28
29 self.EP = [] # 前沿
30 self.EP_fx = [] # ep对应的目标值
31 self.weight_vectors = None # 均匀权重向量
32 self.Euler_distance = None # 欧拉距离矩阵
33 self.pip_size = -1
34
35 self.pop = None
36 # self.pop_dna = None
37
38 def init_mean_vector(self):
39 vectors = distribution_number(self.N + self.m, self.m)
40 vectors = (np.array(vectors) - 1) / self.N
41 self.weight_vectors = vectors
42 self.pip_size = len(vectors)
43 return vectors
44
45 def init_Euler_distance(self):
46 vectors = self.weight_vectors
47 v_len = len(vectors)
48
49 Euler_distance = np.zeros((v_len, v_len))
50 for i in range(v_len):
51 for j in range(v_len):
52 distance = ((vectors[i] - vectors[j]) ** 2).sum()
53 Euler_distance[i][j] = distance
54
55 self.Euler_distance = Euler_distance
56 return Euler_distance
57
58 def init_population(self):
59 pop_size = self.pip_size
60 dna_len = len(self.o_func.domain)
61 pop = []
62 pop_dna = np.random.random(size=(pop_size, dna_len))
63 # 初始个体的 dna
64 for i in range(pop_size):
65 pop.append(Person(pop_dna[i]))
66
67 # 初始个体的 weight_vector, neighbor, o_func
68 for i in range(pop_size):
69 # weight_vector, neighbor, o_func
70 person = pop[i]
71 distance = self.Euler_distance[i]
72 sort_arg = np.argsort(distance)
73 weight_vector = self.weight_vectors[i]
74 # neighbor = pop[sort_arg][:self.T]
75 neighbor = []
76 for i in range(self.T):
77 neighbor.append(pop[sort_arg[i]])
78
79 o_func = self.o_func
80 person.set_info(weight_vector, neighbor, o_func)
81 self.pop = pop
82 # self.pop_dna = pop_dna
83
84 return pop
85
86 def init_Z(self):
87 Z = np.full(shape=self.m, fill_value=float("inf"))
88 for person in self.pop:
89 for i in range(len(self.o_func.f_funcs)):
90 f = self.o_func.f_funcs[i]
91 # f_x_i:某个体,在第i目标上的值
92 f_x_i = f(person.dna)
93 if f_x_i < Z[i]:
94 Z[i] = f_x_i
95
96 self.Z = Z
97
98 def get_fx(self, dna):
99 fx = []
100 for f in self.o_func.f_funcs:
101 fx.append(f(dna))
102 return fx
103
104 def update_ep(self, new_dna):
105 # 将新解与EP每一个进行比较,删除被新解支配的
106 # 如果新解没有被旧解支配,则保留
107 new_dna_fx = self.get_fx(new_dna)
108 accept_new = True # 是否将新解加入EP
109 # print(f"准备开始循环: EP长度{len(self.EP)}")
110 for i in range(len(self.EP) - 1, -1, -1): # 从后往前遍历
111 old_ep_item = self.EP[i]
112 old_fx = self.EP_fx[i]
113 # old_fx = self.get_fx(old_ep_item)
114 a_b = True # 老支配行
115 b_a = True # 新支配老
116 for j in range(len(self.o_func.f_funcs)):
117 if old_fx[j] < new_dna_fx[j]:
118 b_a = False
119 if old_fx[j] > new_dna_fx[j]:
120 a_b = False
121 # T T : fx相等 直接不改变EP
122 # T F :老支配新 留老,一定不要新,结束循环.
123 # F T :新支配老 留新,一定不要这个老,继续循环
124 # F F : 非支配关系 不操作,循环下一个
125 # TF为什么结束循环,FT为什么继续循环,你们可以琢磨下
126 if a_b:
127 accept_new = False
128 break
129 if not a_b and b_a:
130 if len(self.EP) <= i:
131 print(len(self.EP), i)
132 del self.EP[i]
133 del self.EP_fx[i]
134 continue
135
136 if accept_new:
137 self.EP.append(new_dna)
138 self.EP_fx.append(new_dna_fx)
139 return self.EP, self.EP_fx
140
141 def update_Z(self, new_dna):
142 new_dna_fx = self.get_fx(new_dna)
143 Z = self.Z
144 for i in range(len(self.o_func.f_funcs)):
145 if new_dna_fx[i] < Z[i]:
146 Z[i] = new_dna_fx[i]
147 return Z
实现.py
import random import numpy as np from GA.MOEAD.Moead_Util import Moead_Util
from GA.MOEAD.Objective_Function import Objective_Function
from GA.MOEAD.Person import Person import matplotlib.pyplot as plt def draw(x, y):
plt.scatter(x, y, s=10, c="grey") # s 点的大小 c 点的颜色 alpha 透明度
plt.show() iterations = 1000 # 迭代次数
N = 400
m = 2
T = 40
o_func = Objective_Function.get_one_function("zdt3")
pm = 0.5 moead = Moead_Util(N, m, T, o_func, pm) moead.init_mean_vector()
moead.init_Euler_distance()
pop = moead.init_population()
moead.init_Z() for i in range(iterations):
print(i, len(moead.EP))
for person in pop:
p1, p2 = person.choice_two_person()
d1, d2 = Person.cross_get_two_new_dna(p1, p2) if np.random.rand() < pm:
p1.mutation_dna(d1)
if np.random.rand() < pm:
p1.mutation_dna(d2) moead.update_Z(d1)
moead.update_Z(d2)
t1, t2 = person.choice_two_person()
if t1.compare(d1) < 0:
t1.accept_new_dna(d1)
moead.update_ep(d1)
if t2.compare(d1) < 0:
t2.accept_new_dna(d2)
moead.update_ep(d1) # 输出结果画图
EP_fx = np.array(moead.EP_fx) x = EP_fx[:, 0]
y = EP_fx[:, 1]
draw(x, y)
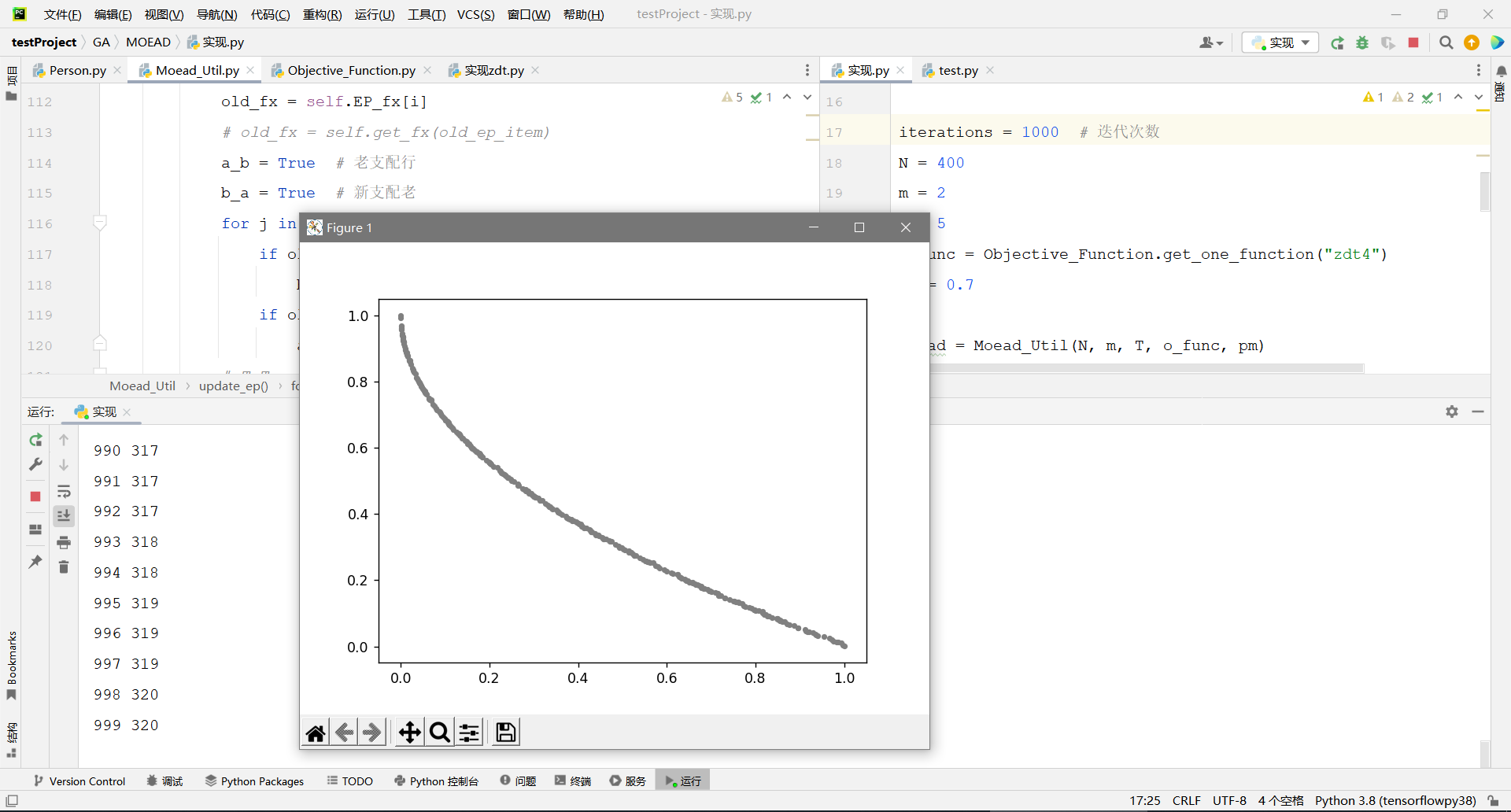
效果-ZDT4
本文原创作者:湘潭大学-魏雄,未经许可禁止转载
MOEAD实现、基于分解的多目标进化、 切比雪夫方法-(python完整代码)的更多相关文章
- 【毕业设计】基于Android的家校互动平台开发(内含完整代码和所有文档)——爱吖校推(你关注的,我们才推)
☆ 写在前面 之前答应大家的毕业答辩之后把所有文档贡献出来,现在答辩已过,LZ信守承诺,把所有文档开源到了GitHub(这个地址包含所有的代码和文档以及PPT,外层为简单的代码).还望喜欢的朋友们,不 ...
- AAAI2019 | 基于区域分解集成的目标检测 论文解读
Object Detection based on Region Decomposition and Assembly AAAI2019 | 基于区域分解集成的目标检测 论文解读 作者 | 文永亮 学 ...
- 基于深度学习的目标检测技术演进:R-CNN、Fast R-CNN,Faster R-CNN
基于深度学习的目标检测技术演进:R-CNN.Fast R-CNN,Faster R-CNN object detection我的理解,就是在给定的图片中精确找到物体所在位置,并标注出物体的类别.obj ...
- #Deep Learning回顾#之基于深度学习的目标检测(阅读小结)
原文链接:https://www.52ml.net/20287.html 这篇博文主要讲了深度学习在目标检测中的发展. 博文首先介绍了传统的目标检测算法过程: 传统的目标检测一般使用滑动窗口的框架,主 ...
- 第十九节、基于传统图像处理的目标检测与识别(词袋模型BOW+SVM附代码)
在上一节.我们已经介绍了使用HOG和SVM实现目标检测和识别,这一节我们将介绍使用词袋模型BOW和SVM实现目标检测和识别. 一 词袋介绍 词袋模型(Bag-Of-Word)的概念最初不是针对计算机视 ...
- 开源项目(9-0)综述--基于深度学习的目标跟踪sort与deep-sort
基于深度学习的目标跟踪sort与deep-sort https://github.com/Ewenwan/MVision/tree/master/3D_Object_Detection/Object_ ...
- 基于层级表达的高效网络搜索方法 | ICLR 2018
论文基于层级表达提出高效的进化算法来进行神经网络结构搜索,通过层层堆叠来构建强大的卷积结构.论文的搜索方法简单,从实验结果看来,达到很不错的准确率,值得学习 来源:[晓飞的算法工程笔记] 公众号 ...
- [信安Presentation]一种基于GPU并行计算的MD5密码解密方法
-------------------paper--------------------- 一种基于GPU并行计算的MD5密码解密方法 0.abstract1.md5算法概述2.md5安全性分析3.基 ...
- 一种基于RBAC模型的动态访问控制改进方法
本发明涉及一种基于RBAC模型的动态访问控制改进方法,属于访问控制领域.对原有RBAC模型进行了权限的改进和约束条件的改进,具体为将权限分为静态权限和动态权限,其中静态权限是非工作流的权限,动态权限是 ...
随机推荐
- 纯css 实现动画的暂停和运动
<template> <div> <input id="stop" type="radio" name="p ...
- 从零开始,开发一个 Web Office 套件(15):拖动边框,平移编辑器
这是一个系列博客,最终目的是要做一个基于 HTML Canvas 的.类似于微软 Office 的 Web Office 套件(包括:文档.表格.幻灯片--等等). 博客园:<从零开始, 开发一 ...
- C++逆向 可变参数Hook
目录 C++逆向 可变参数Hook 0x00 前言: 0x01 C++可变参数: 可变参数简介 可变参数代码实战 0x02 逆向分析C++可变参数原理 0x03 printf Hook实战 Pwn菜鸡 ...
- 接口测试实战| GET/POST 请求区别详解
1.请求行的 method 不同:2.POST 可以附加 body,可以支持 form.json.xml.binary等各种数据格式:3.从行业通用规范的角度来说,无状态变化的建议使用 GET 请求, ...
- 从0开始用webpack开发antd,react组件库npm包并发布
一.初始化一个npm包 1.新建一个文件夹(名称随意,建议和报名一致),输入命令 :npm init -y 会自动生成一个包的说明文件 package.json如下(本文以scroll-antd-ta ...
- Go学习-基本语法(一)
前言 一直对Service Mesh相关内容比较感兴趣,后面一路学习了Dcoker.Kubernetes等相关内容,可以说是对基本概念和使用有一定了解,随着开始学习一些相关的组件的时候,发现基本上全部 ...
- 【面试普通人VS高手系列】死锁的发生原因和怎么避免
一个去阿里面试的小伙伴私信我说:今天被一个死锁的问题难到了. 平常我都特意看了死锁这块的内容,但是回答的时候就想不起来. 这里可能存在一个误区,认为技术是要靠记的. 大家可以想想,平时写代码的时候,这 ...
- XCTF练习题---MISC---something_in_image
XCTF练习题---MISC---something_in_image flag:Flag{yc4pl0fvjs2k1t7T} 解题步骤: 1.观察题目,下载附件,这是一道2019湖湘杯的题目 2.下 ...
- Oracle查看表空间大小
遇到报错 java.sql.SQLException: ORA-01653: 表 MESHIS.HIS_RET_LOT_FQC 无法通过 8 (在表空间 MESHIS_DATA_TBS 中) 扩展 a ...
- Citus 11(分布式 PostgreSQL) 文档贡献与本地运行
Citus 可以使用分片跨多台计算机来水平缩放查询. 其查询引擎会将这些服务器的传入 SQL 查询并行化,加快大型数据集上的响应. 它为需要比其他部署选项更大规模和更高性能的应用程序提供服务:通常,工 ...
