机器学习---用python实现朴素贝叶斯算法(Machine Learning Naive Bayes Algorithm Application)
在《机器学习---朴素贝叶斯分类器(Machine Learning Naive Bayes Classifier)》一文中,我们介绍了朴素贝叶斯分类器的原理。现在,让我们来实践一下。
在这里,我们使用一份皮马印第安女性的医学数据,用来预测其是否会得糖尿病。文件一共有768个样本,我们先剔除缺失值,然后选出20%的样本作为测试样本。
文件下载地址:https://raw.githubusercontent.com/jbrownlee/Datasets/master/pima-indians-diabetes.data.csv
特征分别是:
怀孕次数
口服葡萄糖耐量试验中血浆葡萄糖浓度
舒张压(mm Hg)
三头肌组织褶厚度(mm)
2小时血清胰岛素(μU/ ml)
体重指数(kg/(身高(m))^ 2)
糖尿病系统功能
年龄(岁)
(注:特征值为0表示缺失值)
标签是:
是否患有糖尿病,0代表没有糖尿病,1代表患有糖尿病
在开始之前,先回顾一下朴素贝叶斯算法:
假设训练集特征数为i,记作xi,目标为k个类别,记为Ck,样本数为n,新样本特征记作xnew_sample。
1,计算出先验概率P(Ck),即每个类别在训练集中的概率;
2,分别计算出训练集中每个特征在每个类别下的条件概率P(xi|Ck),具体分为以下三种情况;
a)如果特征数据是离散值,那么我们假设其符合多项式分布,代入公式
b)如果特征数据是布尔类型的离散值,那么我们假设其符合伯努利分布,代入公式
c)如果特征数据是连续值,那么我们假设其符合高斯分布。只需要求出这个特征在每个类别下的平均值μk和方差σk2,然后代入公式
3,对于需要预测类别的新样本,分别计算每个类别下的极大后验概率: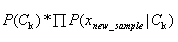 (我们在实际计算中为防止数据下溢,将连乘运算取对数变成相加运算);
(我们在实际计算中为防止数据下溢,将连乘运算取对数变成相加运算);
4,对上述极大后验概率进行比较,最大的那个对应的类别即为结果;
首先导入pandas和numpy库,读取csv文件,然后去除缺失值:
import pandas as pd
import numpy as np data=pd.read_csv(r"C:\Users\ccav\pima-indians-diabetes.data.csv",header=None,\
names=["怀孕次数","口服葡萄糖耐量试验中血浆葡萄糖浓度",\
"舒张压(mm Hg)","三头肌组织褶厚度(mm)",\
"2小时血清胰岛素(μU/ ml)","体重指数(kg/(身高(m))^ 2)",\
"糖尿病系统功能","年龄(岁)","是否患有糖尿病"]) data.iloc[:,0:8]=data.iloc[:,0:8].applymap(lambda x:np.NaN if x==0 else x) #把属性值为0的地方转换成NaN data=data.dropna(how="any",axis=0) #去除有缺失值的行
经过处理还只剩336个有效样本:
print(len(data))
336
通过查看,我们发现在这336个有效样本中,225个是没有糖尿病人的样本,111个是患有糖尿病人的样本,显然原数据里两个类别的样本是不均衡的。这个问题可以通过不同的采样方法解决,但是鉴于简便起见,我们随机选取大约20%的样本用于测试:
#随机选取80%的样本作为训练样本
data_train=data.sample(frac=0.8,random_state=4,axis=0)
#剩下的作为测试样本
test_idx=[i for i in data.index.values if i not in data_train.index.values]
data_test=data.loc[test_idx,:]
由于我们一共有8个特征,2个类别,因此,我们一共需要计算16个条件概率以及2个先验概率,然后根据这些数据计算出2个极大后验概率。该如何计算这些数据,又该如何合并,如果不事先想清楚怎么做,很容易被搞晕。此外,考虑到数据是连续值,我们假设其符合正态分布,因此在训练时需要计算出每个特征的平均值和方差。
让我们先来理清一下思路:
1,按类别分隔数据
2,获取类别总数和类别名称
3,训练数据:计算每个类别的先验概率,计算每个类别每个特征的平均值和方差
4,预测:计算每个类别每个特征的条件概率,计算每个类别的极大后验概率并合并,得出最大可能的类别名称
按照这个步骤,我们发现以下几项都是需要复用的:按类别分隔数据,计算每个类别的先验概率,计算每个类别每个特征的平均值和方差,计算每个类别每个特征的条件概率。因此,我们把这几项分别做成function。
按类别分隔数据:
def SepByClass(X, y):
###按类别分隔数据###
###输入未分类的特征和目标,输出分类完成的数据(字典形式)###
num_of_samples=len(y) #总样本数 y=y.reshape(X.shape[0],1)
data=np.hstack((X,y)) #把特征和目标合并成完整数据 data_byclass={} #初始化分类数据,为一个空字典
#提取各类别数据,字典的键为类别名,值为对应的分类数据
for i in range(len(data[:,-1])):
if i in data[:,-1]:
data_byclass[i]=data[data[:,-1]==i] class_name=list(data_byclass.keys()) #类别名
num_of_class=len(data_byclass.keys()) #类别总数 return data_byclass
计算每个类别的先验概率:
def CalPriorProb(y_byclass):
###计算y的先验概率(使用拉普拉斯平滑)###
###输入当前类别下的目标,输出该目标的先验概率###
#计算公式:(当前类别下的样本数+1)/(总样本数+类别总数)
return (len(y_byclass)+1)/(num_of_samples+num_of_class)
计算每个类别每个特征的平均值和方差:
def CalXMean(X_byclass):
###计算各类别特征各维度的平均值###
###输入当前类别下的特征,输出该特征各个维度的平均值###
X_mean=[]
for i in range(X_byclass.shape[1]):
X_mean.append(np.mean(X_byclass[:,i]))
return X_mean def CalXVar(X_byclass):
###计算各类别特征各维度的方差###
###输入当前类别下的特征,输出该特征各个维度的方差###
X_var=[]
for i in range(X_byclass.shape[1]):
X_var.append(np.var(X_byclass[:,i]))
return X_var
计算每个类别每个特征的条件概率:
def CalGaussianProb(X_new, mean, var):
###计算训练集特征(符合正态分布)在各类别下的条件概率###
###输入新样本的特征,训练集特征的平均值和方差,输出新样本的特征在相应训练集中的分布概率###
#计算公式:(np.exp(-(X_new-mean)**2/(2*var)))*(1/np.sqrt(2*np.pi*var))
gaussian_prob=[]
for a,b,c in zip(X_new, mean, var):
formula1=np.exp(-(a-b)**2/(2*c))
formula2=1/np.sqrt(2*np.pi*c)
gaussian_prob.append(formula2*formula1)
return gaussian_prob
接下来,我们开始训练数据。首先按类别分隔数据,然后遍历每一个类别,分别计算每个类别的先验概率和每个类别每个特征的平均值和方差,并储存在列表中。
def fit(X, y):
###训练数据###
###输入训练集特征和目标,输出目标的先验概率,特征的平均值和方差###
#将输入的X,y转换为numpy数组
X, y = np.asarray(X, np.float32), np.asarray(y, np.float32) data_byclass=Gaussian_NB.SepByClass(X,y) #将数据分类
#计算各类别数据的目标先验概率,特征平均值和方差
for data in data_byclass.values():
X_byclass=data[:,:-1]
y_byclass=data[:,-1]
prior_prob.append(Gaussian_NB.CalPriorProb(y_byclass))
X_mean.append(Gaussian_NB.CalXMean(X_byclass))
X_var.append(Gaussian_NB.CalXVar(X_byclass))
return prior_prob, X_mean, X_var
最后,输入一个新样本的特征,预测其所属的类别。首先,遍历之前在训练数据时计算出的每个类别的先验概率和每个类别每个特征的平均值和方差,再以此计算条件概率和极大后验概率。选出最大的极大后验概率所对应的索引,最后提取其对应的类别名称。
def predict(X_new):
###预测数据###
###输入新样本的特征,输出新样本最有可能的目标###
#将输入的x_new转换为numpy数组
X_new=np.asarray(X_new, np.float32) posteriori_prob=[] #初始化极大后验概率 for i,j,o in zip(prior_prob, X_mean, X_var):
gaussian=Gaussian_NB.CalGaussianProb(X_new,j,o)
posteriori_prob.append(np.log(i)+sum(np.log(gaussian)))
idx=np.argmax(posteriori_prob) return class_name[idx]
整理一下以上代码,我们把这个高斯朴素贝叶斯分类器做成一个类,完整代码如下:
import pandas as pd
import numpy as np data=pd.read_csv(r"C:\Users\ccav\pima-indians-diabetes.data.csv",header=None,\
names=["怀孕次数","口服葡萄糖耐量试验中血浆葡萄糖浓度",\
"舒张压(mm Hg)","三头肌组织褶厚度(mm)",\
"2小时血清胰岛素(μU/ ml)","体重指数(kg/(身高(m))^ 2)",\
"糖尿病系统功能","年龄(岁)","是否患有糖尿病"]) data.iloc[:,0:8]=data.iloc[:,0:8].applymap(lambda x:np.NaN if x==0 else x) #把属性值为0的地方转换成NaN data=data.dropna(how="any",axis=0) #去除有缺失值的行 #随机选取80%的样本作为训练样本
data_train=data.sample(frac=0.8,random_state=4,axis=0)
#剩下的作为测试样本
test_idx=[i for i in data.index.values if i not in data_train.index.values]
data_test=data.loc[test_idx,:] #提取训练集和测试集的特征和目标
X_train=data_train.iloc[:,:-1]
y_train=data_train.iloc[:,-1] X_test=data_test.iloc[:,:-1]
y_test=data_test.iloc[:,-1] class Gaussian_NB:
def __init__(self):
self.num_of_samples = None
self.num_of_class = None
self.class_name = []
self.prior_prob = []
self.X_mean = []
self.X_var = [] def SepByClass(self, X, y):
###按类别分隔数据###
###输入未分类的特征和目标,输出分类完成的数据(字典形式)###
self.num_of_samples=len(y) #总样本数 y=y.reshape(X.shape[0],1)
data=np.hstack((X,y)) #把特征和目标合并成完整数据 data_byclass={} #初始化分类数据,为一个空字典
#提取各类别数据,字典的键为类别名,值为对应的分类数据
for i in range(len(data[:,-1])):
if i in data[:,-1]:
data_byclass[i]=data[data[:,-1]==i] self.class_name=list(data_byclass.keys()) #类别名
self.num_of_class=len(data_byclass.keys()) #类别总数 return data_byclass def CalPriorProb(self, y_byclass):
###计算y的先验概率(使用拉普拉斯平滑)###
###输入当前类别下的目标,输出该目标的先验概率###
#计算公式:(当前类别下的样本数+1)/(总样本数+类别总数)
return (len(y_byclass)+1)/(self.num_of_samples+self.num_of_class) def CalXMean(self, X_byclass):
###计算各类别特征各维度的平均值###
###输入当前类别下的特征,输出该特征各个维度的平均值###
X_mean=[]
for i in range(X_byclass.shape[1]):
X_mean.append(np.mean(X_byclass[:,i]))
return X_mean def CalXVar(self, X_byclass):
###计算各类别特征各维度的方差###
###输入当前类别下的特征,输出该特征各个维度的方差###
X_var=[]
for i in range(X_byclass.shape[1]):
X_var.append(np.var(X_byclass[:,i]))
return X_var def CalGaussianProb(self, X_new, mean, var):
###计算训练集特征(符合正态分布)在各类别下的条件概率###
###输入新样本的特征,训练集特征的平均值和方差,输出新样本的特征在相应训练集中的分布概率###
#计算公式:(np.exp(-(X_new-mean)**2/(2*var)))*(1/np.sqrt(2*np.pi*var))
gaussian_prob=[]
for a,b,c in zip(X_new, mean, var):
formula1=np.exp(-(a-b)**2/(2*c))
formula2=1/np.sqrt(2*np.pi*c)
gaussian_prob.append(formula2*formula1)
return gaussian_prob def fit(self, X, y):
###训练数据###
###输入训练集特征和目标,输出目标的先验概率,特征的平均值和方差###
#将输入的X,y转换为numpy数组
X, y = np.asarray(X, np.float32), np.asarray(y, np.float32) data_byclass=Gaussian_NB.SepByClass(X,y) #将数据分类
#计算各类别数据的目标先验概率,特征平均值和方差
for data in data_byclass.values():
X_byclass=data[:,:-1]
y_byclass=data[:,-1]
self.prior_prob.append(Gaussian_NB.CalPriorProb(y_byclass))
self.X_mean.append(Gaussian_NB.CalXMean(X_byclass))
self.X_var.append(Gaussian_NB.CalXVar(X_byclass)) return self.prior_prob, self.X_mean, self.X_var def predict(self,X_new):
###预测数据###
###输入新样本的特征,输出新样本最有可能的目标###
#将输入的x_new转换为numpy数组
X_new=np.asarray(X_new, np.float32) posteriori_prob=[] #初始化极大后验概率 for i,j,o in zip(self.prior_prob, self.X_mean, self.X_var):
gaussian=Gaussian_NB.CalGaussianProb(X_new,j,o)
posteriori_prob.append(np.log(i)+sum(np.log(gaussian)))
idx=np.argmax(posteriori_prob) return self.class_name[idx] if __name__=="__main__":
Gaussian_NB=Gaussian_NB() #实例化Gaussian_NB
Gaussian_NB.fit(X_train,y_train) #使用Gaussian_NB模型训练数据
acc=0
TP=0
FP=0
FN=0
for i in range(len(X_test)):
predict=Gaussian_NB.predict(X_test.iloc[i,:])
target=np.array(y_test)[i]
if predict==1 and target==1:
TP+=1
if predict==0 and target==1:
FP+=1
if predict==target:
acc+=1
if predict==1 and target==0:
FN+=1
print("准确率:",acc/len(X_test))
print("查准率:",TP/(TP+FP))
print("查全率:",TP/(TP+FN))
print("F1:",2*TP/(2*TP+FP+FN))
结果如下:
准确率: 0.7611940298507462
查准率: 0.7391304347826086
查全率: 0.6296296296296297
F1: 0.68
由于样本不均衡,因此光看准确率是不够的,在这里一并计算了查准率和查全率以及F1值。可以看出这个分类器的效果不是很好,一方面是因为样本数量较少,另一方面朴素贝叶斯的预测能力确实比不上复杂模型,只能提供一个粗略的判断。
机器学习---用python实现朴素贝叶斯算法(Machine Learning Naive Bayes Algorithm Application)的更多相关文章
- 机器学习---朴素贝叶斯分类器(Machine Learning Naive Bayes Classifier)
朴素贝叶斯分类器是一组简单快速的分类算法.网上已经有很多文章介绍,比如这篇写得比较好:https://blog.csdn.net/sinat_36246371/article/details/6014 ...
- Andrew Ng机器学习公开课笔记 -- 朴素贝叶斯算法
网易公开课,第5,6课 notes,http://cs229.stanford.edu/notes/cs229-notes2.pdf 前面讨论了高斯判别分析,是一种生成学习算法,其中x是连续值 这里要 ...
- Python机器学习笔记:朴素贝叶斯算法
朴素贝叶斯是经典的机器学习算法之一,也是为数不多的基于概率论的分类算法.对于大多数的分类算法,在所有的机器学习分类算法中,朴素贝叶斯和其他绝大多数的分类算法都不同.比如决策树,KNN,逻辑回归,支持向 ...
- Python机器学习算法 — 朴素贝叶斯算法(Naive Bayes)
朴素贝叶斯算法 -- 简介 朴素贝叶斯法是基于贝叶斯定理与特征条件独立假设的分类方法.最为广泛的两种分类模型是决策树模型(Decision Tree Model)和朴素贝叶斯模型(Naive Baye ...
- 机器学习:python中如何使用朴素贝叶斯算法
这里再重复一下标题为什么是"使用"而不是"实现": 首先,专业人士提供的算法比我们自己写的算法无论是效率还是正确率上都要高. 其次,对于数学不好的人来说,为了实 ...
- 朴素贝叶斯算法的python实现
朴素贝叶斯 算法优缺点 优点:在数据较少的情况下依然有效,可以处理多类别问题 缺点:对输入数据的准备方式敏感 适用数据类型:标称型数据 算法思想: 朴素贝叶斯比如我们想判断一个邮件是不是垃圾邮件,那么 ...
- 朴素贝叶斯算法--python实现
朴素贝叶斯算法要理解一下基础: [朴素:特征条件独立 贝叶斯:基于贝叶斯定理] 1朴素贝叶斯的概念[联合概率分布.先验概率.条件概率**.全概率公式][条件独立性假设.] 极大似然估计 ...
- 朴素贝叶斯算法原理及Spark MLlib实例(Scala/Java/Python)
朴素贝叶斯 算法介绍: 朴素贝叶斯法是基于贝叶斯定理与特征条件独立假设的分类方法. 朴素贝叶斯的思想基础是这样的:对于给出的待分类项,求解在此项出现的条件下各个类别出现的概率,在没有其它可用信息下,我 ...
- 朴素贝叶斯算法的python实现方法
朴素贝叶斯算法的python实现方法 本文实例讲述了朴素贝叶斯算法的python实现方法.分享给大家供大家参考.具体实现方法如下: 朴素贝叶斯算法优缺点 优点:在数据较少的情况下依然有效,可以处理多类 ...
随机推荐
- kafka服务端实验记录
kafka单机实验: 环境准备: 1.下载kafka,zookeeper,并解压 wget http://mirror.bit.edu.cn/apache/kafka/2.3.0/kafka_2.11 ...
- C++使用代码创建一个Windows桌面应用程序
WinMain函数 Windows应用程序的唯一程序入口. 函数原型 int WINAPI WinMain { HINSTANCE hInstancem HINSTANCE hPreInstance, ...
- vs2012配置gitHub管理代码详细步骤
http://www.bitscn.com/pdb/otherdb/201411/411244.html
- 提高QPS
常用方案 1.异步化+MQ 即非阻塞,化繁为简,拿到你需要处理的资源后尽快回复.适用于事务处理场景,且无需对上游返回数据场景. 2.无锁设计 本质上是要降低锁冲突,基于数据版本的乐观锁 有效的减少了互 ...
- CSS 实现盒子水平居中、垂直居中和水平垂直居中的方法
CSS 实现盒子模型水平居中 水平居中效果图如下: HTML: CSS 全局样式: 方法一:使用margin: 0 auto;(只适用于子盒子有宽的时候) 方法二:text-align + disp ...
- ORACLE主键ID的生成
转自:https://blog.csdn.net/yh_zeng2/article/details/83477880 一般常用的方法有两种,使用Sequence和使用SYS_GUID(); 方法一 ...
- Jquery简单闭包
<html> <body> <script src="Js/Index.js"></script> <script type= ...
- PLC编程的基础知识的总结
1.Programmable Logic Controller PLC 代替计算机实现复杂的逻辑控制系统 ,可编程控制器是一种数字运算操作电子系统,转门在工业控制系统中使用而设计的,采用一种可以 ...
- python中的嵌套类
python中的嵌套类 在.NET和JAVA语言中看到过嵌套类的实现,作为外部类一个局部工具还是很有用的,今天在python也看到了很不错支持一下.动态语言中很好的嵌套类的实现,应该说嵌套类解决设计问 ...
- 使用BERT预训练模型+微调进行文本分类
本文记录使用BERT预训练模型,修改最顶层softmax层,微调几个epoch,进行文本分类任务. BERT源码 首先BERT源码来自谷歌官方tensorflow版:https://github.co ...
