maftools | 从头开始绘制发表级oncoplot(瀑布图)
本文首发于微信公众号 **“ 生信补给站 ”** ,期待您的关注!!!
原文链接:https://mp.weixin.qq.com/s/G-0PtaoO6bYuhx_D_Rlrlw
对于组学数据的分析和展示来说,maftools算是一个宝藏“R包”,可用于MAF格式的组学数据的汇总,分析和可视化展示。
随着癌症基因组学的进步, 突变注释格式+Specification) (MAF) 被广泛用于存储检测到的somatic variants。The Cancer Genome Atlas 项目对30多种不同的癌症进行了测序,每种癌症类型的样本量超过200种。maftools-R包能够有效的汇总,分析和可视化MAF格式的文件。
maftools函数主要分为可视化和分析两个模块,其主要功能及简短的描述如下所示,使用时只需读取MAF文件然后降MAF对象传递给所需要的绘图或分析功能即可。
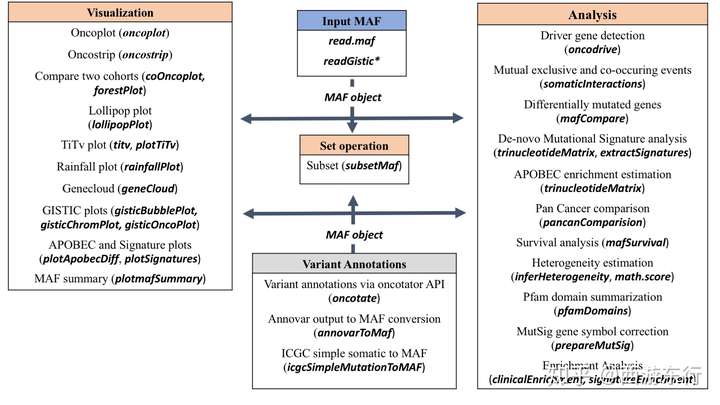
本次主要使用R-maftools包绘制组学突变结果(MAF)的oncoplot或者叫“瀑布图”。
一、 载入R包,数据
1) 载入maftools包
if (!require("BiocManager"))
install.packages("BiocManager")
BiocManager::install("maftools")
2) 载入数据
通过read.maf函数读入MAF文件,将各种数据(组学基因突变,拷贝数变异,临床数据,表达数据等)汇总并将其存储为MAF对象。
#TCGA-LAML MAF file (gz)
laml.maf = system.file('extdata', 'tcga_laml.maf.gz', package = 'maftools')
#clinical information (optional)
laml.clin = system.file('extdata', 'tcga_laml_annot.tsv', package = 'maftools')
laml = read.maf(maf = laml.maf, clinicalData = laml.clin)
MAF对象中除了上面基因突变数据和对应的临床数据外,还可以加入拷贝数变异,表达数据等其他数据类型,后面需要的时候会添加。
二、 MAF对象汇总
1) 展示MAF重点变量的summary信息
#Shows sample summry.
getSampleSummary(laml)
#Shows gene summary.
getGeneSummary(laml)
#Shows all fields in MAF
getFields(laml)
#shows clinical data associated with samples
getClinicalData(laml)
#Writes maf summary to an output file with basename laml.
write.mafSummary(maf = laml, basename = 'laml')
上图为临床数据(getClinicalData)的summary结果,其余可自行输入查看结果。
2) 绘制MAF-summary图
使用 plotmafSummary 绘制 maf 文件的summary信息,如下:
#plotmafSummary
plotmafSummary(maf = laml, rmOutlier = TRUE, addStat = 'median', dashboard = TRUE, titvRaw = FALSE)
堆叠的 barplot展示maf 文件中每个样本中的变异数量,并添加中位线,以显示队列间的中位数变异数量。箱线图展示variant_Classification的变异类型。
三、 绘制oncoplot(瀑布)图
1 绘制基础oncoplots(瀑布图)
oncoplots或者瀑布图可以很好的展示maf文件中的变异信息,侧面条形图和顶部条形图可分别由drawRowBar和drawColBar参数控制。
#展示top20的变异genes.
oncoplot(maf = laml, top = 20)
注:变异注释为Multi_Hit表示同一样本中突变多次的基因。
2 更改变异类型的颜色
#此处使用RColorBrewer的颜色,当然也可以使用任意颜色
vc_cols = RColorBrewer::brewer.pal(n = 8, name = 'Paired')
names(vc_cols) = c(
'Frame_Shift_Del',
'Missense_Mutation',
'Nonsense_Mutation',
'Multi_Hit',
'Frame_Shift_Ins',
'In_Frame_Ins',
'Splice_Site',
'In_Frame_Del'
)
#查看变异类型对应的颜色
print(vc_cols)
#> Frame_Shift_Del Missense_Mutation Nonsense_Mutation Multi_Hit
#> "#A6CEE3" "#1F78B4" "#B2DF8A" "#33A02C"
#> Frame_Shift_Ins In_Frame_Ins Splice_Site In_Frame_Del
#> "#FB9A99" "#E31A1C" "#FDBF6F" "#FF7F00"
oncoplot(maf = laml, colors = vc_cols, top = 20)
3 添加copy number 信息
可以使用两种方式向maf文件中添加SCNA信息
GISTIC result
Custom copy number table
3.1 GISTIC results
GISTIC会得到很多结果,此处只需要四个文件即可: all_lesions.conf_XX.txt, amp_genes.conf_XX.txt, del_genes.conf_XX.txt, scores.gistic:
#GISTIC results
all.lesions =
system.file("extdata", "all_lesions.conf_99.txt", package = "maftools")
amp.genes =
system.file("extdata", "amp_genes.conf_99.txt", package = "maftools")
del.genes =
system.file("extdata", "del_genes.conf_99.txt", package = "maftools")
scores.gis =
system.file("extdata", "scores.gistic", package = "maftools")
#Read GISTIC results along with MAF
laml.plus.gistic = read.maf(
maf = laml.maf,
gisticAllLesionsFile = all.lesions,
gisticAmpGenesFile = amp.genes,
gisticDelGenesFile = del.genes,
gisticScoresFile = scores.gis,
isTCGA = TRUE,
verbose = FALSE,
clinicalData = laml.clin
)
#绘制含有SCNA信息的oncolpot
oncoplot(maf = laml.plus.gistic, top = 20)
3.2 Custom copy-number table
可以自定义一个copy-number结果,例如,让我们在随机的 20 个样本中创建 DNMT3A 的假 SCNA变异。
set.seed(seed = 1234)
barcodes = as.character(getSampleSummary(x = laml)[,Tumor_Sample_Barcode])
#Random 20 samples
dummy.samples = sample(x = barcodes,
size = 20,
replace = FALSE)
#Genarate random CN status for above samples
cn.status = sample(
x = c('Amp', 'Del'),
size = length(dummy.samples),
replace = TRUE
)
custom.cn.data = data.frame(
Gene = "DNMT3A",
Sample_name = dummy.samples,
CN = cn.status,
stringsAsFactors = FALSE
)
head(custom.cn.data)
#> Gene Sample_name CN
#> 1 DNMT3A TCGA-AB-2898 Amp
#> 2 DNMT3A TCGA-AB-2879 Amp
#> 3 DNMT3A TCGA-AB-2920 Del
#> 4 DNMT3A TCGA-AB-2866 Amp
#> 5 DNMT3A TCGA-AB-2892 Amp
#> 6 DNMT3A TCGA-AB-2863 Amp
#添加SCNA信息
laml.plus.cn = read.maf(maf = laml.maf,
cnTable = custom.cn.data,
verbose = FALSE)
oncoplot(maf = laml.plus.cn, top = 10)
4 添加 significance values
输入data.frame格式的数据,包括gene and q值两列即可:
#MutSig results
laml.mutsig = system.file("extdata", "LAML_sig_genes.txt.gz", package = "maftools")
oncoplot(
maf = laml,
mutsig = laml.mutsig,
mutsigQval = 0.01,
)
5 临床信息 annotations
1)使用存储在 clinical.data 中的变量进行注释
#使用FAB_classification注释
oncoplot(maf = laml, clinicalFeatures = 'FAB_classification')
2)临床信息注释的变量同样可以自定义颜色:
#更改FAB classification的颜色设置
fabcolors = RColorBrewer::brewer.pal(n = 8,name = 'Spectral')
#颜色和变量的种类要一样多
names(fabcolors) = c("M0", "M1", "M2", "M3", "M4", "M5", "M6", "M7")
fabcolors = list(FAB_classification = fabcolors)
oncoplot(
maf = laml,
clinicalFeatures = 'FAB_classification',
sortByAnnotation = TRUE,
annotationColor = fabcolors
)
3)注释多个临床数据
oncoplot(
maf = laml,
clinicalFeatures = c('FAB_classification','SEX','Overall_Survival_Status'),
annotationColor = fabcolors)
以上就是如何使用R-maftools包完成瀑布图绘制的简单介绍,然后基本上也就完成了文献的Fig1
maftools | 从头开始绘制发表级oncoplot(瀑布图)的更多相关文章
- Tableau绘制漏斗图、甘特图、瀑布图、镶边面积图、阴影坡度图
Tableau绘制漏斗图.甘特图.瀑布图.镶边面积图.阴影坡度图 本文首发于博客冰山一树Sankey,去博客浏览效果更好.直接右上角搜索该标题即可 一. 漏斗图 数据源 1.1 分色直条漏斗图 (1) ...
- Excel催化剂图表系列之品味IBCS瀑布图观察企业利润构成
IBCS图表,每个细节都值得反复琢磨参悟,此篇给大家送上详尽的瀑布图方式下的利润数据观察.请不要拿Excel2016版提供的瀑布图与IBCS版的瀑布图作对比,那完全不是一个级别的,可以类比为拿一辆经济 ...
- 教你用Python创建瀑布图
介绍 对于绘制某些类型的数据来说,瀑布图是一种十分有用的工具.不足为奇的是,我们可以使用Pandas和matplotlib创建一个可重复的瀑布图. 在往下进行之前,我想先告诉大家我指代的是哪种类型的图 ...
- 使用highcharts绘制美观的燃尽图
使用highcharts绘制美观的燃尽图 助教在博客中介绍了两种绘制燃尽图的方法,但是我们组在使用时发现有些任务不适合写进issue,而且网站生成的燃尽图不是很美观,因此我们打算使用其他方法自己绘制燃 ...
- C#绘制立体三维饼状图
转载自原文 C#绘制立体三维饼状图(超酷) 内容原文地址:LINK [翻译]Julijan Sribar著3D Pie Chart一个用于绘制立体三维饼状图的C#类库[简介]本文的想法就是创建一个独立 ...
- 绘制3D的托卡马克位形图的matlab脚本文件 ThreeD.m
% 绘制3D的托卡马克位形图, (V 0.1 by Jiale Chan for Y. H. Huang) % Dee Formula % 特征参数 rzero = 2.0; rmax ...
- [math] 绘制空间几何体的直观图
这么多年,一直凭着从天而降的神来之灵感画着立体图. 而今才知道在二维平面上绘制空间几何体的直观图也是有方法的.叫做“画法几何” 1. 斜二测图 就是倾斜y轴,使y轴与x轴成45度的夹角.见: http ...
- 看懂 Fiddler 的瀑布图
最近准备给组内的新同学们分享下 Fiddler 这枚神器,可以讲的地方太多,我打算把一节课讲不完的内容写在博客上,大家可以随便看看.今天先介绍下 Fiddler 的瀑布图. 每个网络请求都会经历域名解 ...
- Qt绘制简单的风向玫瑰图代码
1.绘制简单的风向玫瑰图代码2.主要使用QPainter3.在子widget上绘制需要使用widget监视事件 eventfilter update();//更新界面 //镜头12 QPainter ...
随机推荐
- Microsoft Visual C++ Runtime library not enough space for thread data
Microsoft Visual C++ Runtime library not enough space for thread data 电脑最近一直在运行的时候,弹出提示框,如下: 解决办 ...
- 【Oracle/Java】给十六张表各插入十万条数据 单线程耗时半小时 多线程耗时一刻钟
测试机Oracle版本: SQL> select * from v$version; BANNER ----------------------------------------------- ...
- python模块之request模块的理解
首先还是老生长谈,说说定义和作用,request模块是一个用于访问网络的模块,其实类似的模块还有很多,不在一一在这里解释.这么多的相似的模块为什么都说只有这个好用呢.因为他人性化.如果你学过urlli ...
- 30 Flutter自定义Dialog
MyDialog.dart import 'dart:async'; import 'package:flutter/material.dart'; class MyDialog extends Di ...
- SpringCloud学习成长之路二 服务客户端(rest+ribbon)
在微服务架构中,业务都会被拆分成一个独立的服务,服务与服务的通讯是基于http restful的. Spring cloud有两种服务调用方式,一种是ribbon+restTemplate,另一种是f ...
- ALV显示金额字段值扩大100倍
内表数据 物料 库位 期末庫存金額F0D7004DSA 1PYF 701410.944F0D7004DSA 1SNT ...
- 重新学习微信小程序
基础学习: 传送门:http://www.jianshu.com/p/1cec15a81722 这个简书博客介绍的很详细,今天思思重新学习了一下. 一路到最后只遇到一个坑,还是自己不仔细.这里记录下: ...
- 【VS开发】TCP服务端如何判断客户端断开连接
原文出自:http://www.cnblogs.com/youxin/p/4056041.html 一篇文章: 最近在做一个服务器端程序,C/S结构.功能方面比较简单就是client端与serve ...
- JWT(JSON Web Tokens)操作帮助类
载荷实体: /// <summary> /// JWT载荷实体 /// </summary> public sealed class JWTPlayloadInfo { /// ...
- 阿里云主机如何设置swap
简介:swap交换空间实际上是一个磁盘分区,在安装操作系统时,默认划分出物理内存的1~2倍空间用于交换分区,它类似于 Windows 的虚拟内存.系统会把一部分硬盘空间虚拟成内存使用,将系统内非活动内 ...
