R语言-聚类与分类
一.聚类:
一般步骤:
1.选择合适的变量
2.缩放数据
3.寻找异常点
4.计算距离
5.选择聚类算法
6.采用一种或多种聚类方法
7.确定类的数目
8.获得最终聚类的解决方案
9.结果可视化
10.解读类
11.验证结果
1.层次聚类分析
案例:采用flexclust的营养数据集作为参考
1.基于5种营养标准的27类鱼,禽,肉的相同点和不同点是什么
2.是否有一种办法把这些食物分成若干各类
1.1计算距离
data(nutrient,package = 'flexclust')
head(nutrient,4)
d <- dist(nutrient)
as.matrix(d)[1:4,1:4]
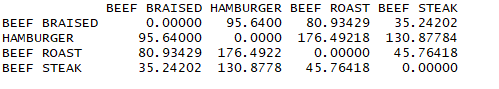
结论:观测的距离越大,异质性越大
1.2平均联动聚类
row.names(nutrient) <- tolower(row.names(nutrient))
nutrient.scaled <- scale(nutrient)
d2 <- dist(nutrient.scaled)
fit.average <- hclust(d2,method = 'average')
plot(fit.average,hang=-1,cex=.8,main='Average Linkage Clustering')
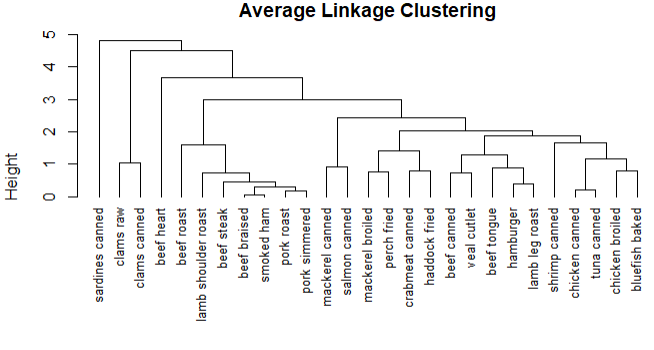
结论:只能提供食物营养成分的相似性和相异性
1.3获取聚类的个数
library('NbClust')
devAskNewPage(ask = T)
nc <- NbClust(nutrient.scaled, distance="euclidean",
min.nc=2, max.nc=15, method="average")
table(nc$Best.n[1,])
barplot(table(nc$Best.n[1,]),
xlab = 'Number of Clusters',ylab = 'Number of Criteria',
main='Number of Clusters chosen by 26 criteria')

结论:分别有4个投票数最多的聚类(2,3,5,15),从中选择一个更适合的聚类数
1.4获取聚类的最终方案
# 聚类分配情况
clusters <- cutree(fit.average,k=5)
table(clusters)
# 描述聚类
aggregate(nutrient,by=list(clusters=clusters),median)
aggregate(as.data.frame(nutrient.scaled),by=list(clusters=clusters),median)
plot(fit.average,hang=-1,cex=.8,main='Average Linkpage Clustering\n 5 Cluster Solution')
rect.hclust(fit.average,k=5)
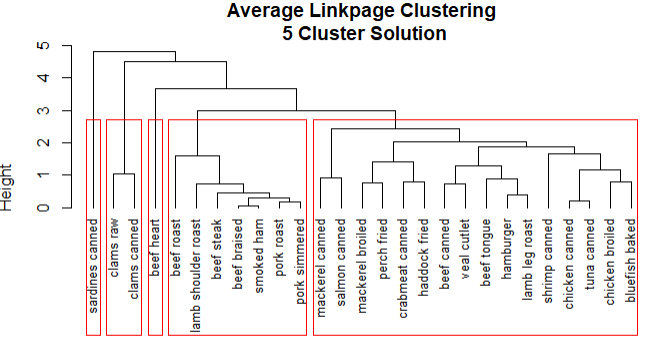
结论:
1.sardines canned形成自己的类,因为钙含量比较高
2.beef heart也是单独的类,富含蛋白质和铁
3.beef roast到pork simmered含有高能量和脂肪
4.clams raw到clams canned含有较高的维生素
5.mackerel canned到bluefish baked含有较低的铁
2.划分聚类分析
案例:采用rattle.data中的wine数据集进行分析
1.葡萄酒数据的K均值聚类
# 使用卵石图确定类的数量
wssplot <- function(data,nc=15,seed=1234){
wss <- (nrow(data)-1) * sum(apply(data,2,var))
for (i in 2:nc) {
set.seed(seed)
wss[i] <- sum(kmeans(data,centers = i)$withinss)
}
plot(1:nc,wss,type = 'b',xlab = 'Number of Clusters',ylab = 'Within groups sum of squares')
}
data(wine,package = 'rattle.data') head(wine)
df <- scale(wine[-1])
wssplot(df)
library(NbClust)
set.seed(1234)
# 确定聚类的数量
nc <- NbClust(df, min.nc=2, max.nc=15, method="kmeans")
table(nc$Best.n[1,])
barplot(table(nc$Best.n[1,]),
xlab="Numer of Clusters", ylab="Number of Criteria",
main="Number of Clusters Chosen by 26 Criteria")
set.seed(1234)
# 进行k值聚类分析
fit.km <- kmeans(df, 3, nstart=25)
fit.km$size
fit.km$centers
aggregate(wine[-1], by=list(cluster=fit.km$cluster), mean)

结论:分3个聚类对数据有很好的拟合
# 使用兰德系数来量化类型变量和类之间的协议
ct.km <- table(wine$Type,fit.km$cluster)
library(flexclust)
randIndex(ct.km)

结论:拟合结果优秀
围绕中心点的分类:因为K均值聚类方法是基于均值的,所以对异常值较为敏感,更为稳健的方法是围绕中心点的划分,
k均值聚类一般使用欧几里得距离,而PAM可以使用任意的距离来计算
library(cluster)
set.seed(1234)
fit.pam <- pam(wine[-1],k=3,stand = T)
fit.pam$medoids
clusplot(fit.pam,main = 'Bivariate Cluster Plot')
ct.pam <- table(wine$Type,fit.pam$clustering)
randIndex(ct.pam)
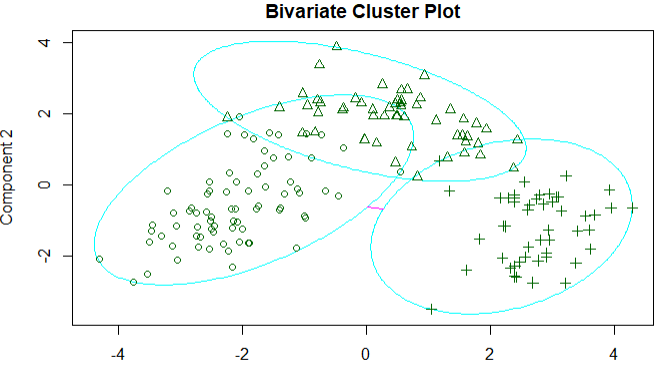
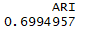
结论:调整后的兰德指数从之前的0.9下降到0.7
3.避免不存在的聚类
3.1查看数据集
library(fMultivar)
set.seed(1234)
df <- rnorm2d(1000,rho=.5)
df <- as.data.frame(df)
plot(df,main='Bivariate Normal Distribution with rho=0.5')

结论:没有存在的类
3.2计算聚类的个数
library(NbClust)
nc <- NbClust(df,min.nc = 2,max.nc = 15,method = 'kmeans')
dev.new()
barplot(table(nc$Best.n[1,]),xlab="Numer of Clusters", ylab="Number of Criteria",
main="Number of Clusters Chosen by 26 Criteria")

结论:一共可分为3各类
3.3聚类图像
library(ggplot2)
fit2 <- pam(df,k=2)
df$clustering <- factor(fit2$clustering)
ggplot(data = df,aes(x=V1,y=V2,color=clustering,shape=clustering))+
geom_point()+
ggtitle('Clustering of Bivariate Normal Data')
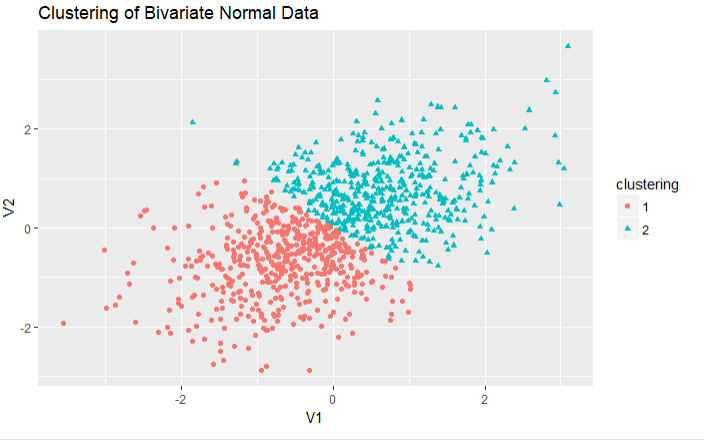
结论:对于二元数据的PAM聚类分析,提取出2类
3.4分析聚类
plot(nc$All.index[,4],type='o',ylab='ccc',xlab='Number of Clusters',col='blue')
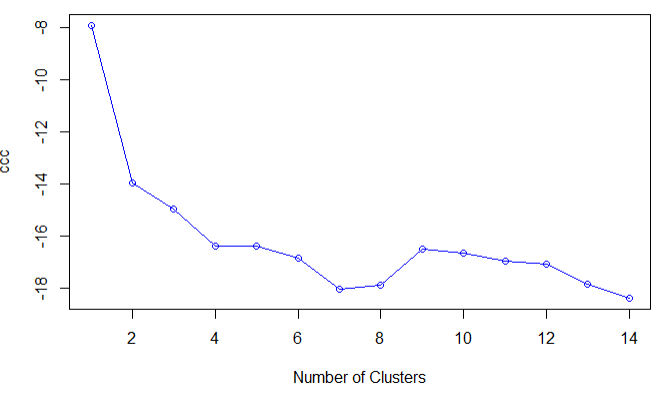
结论:二元正态数据的CCC图,表明没有类存在,当CCC为负数并且对于两类或者是更多的类的递减
二.分类
使用机器学习来预测二分类结果
案例分析:使用乳腺癌数据作为测试,训练集建立逻辑回归,决策时,条件推断树,随机森林,支持向量机等分类模型,测试集用于评估各个模型的有效性
1.准备数据
loc <- "http://archive.ics.uci.edu/ml/machine-learning-databases/"
ds <- "breast-cancer-wisconsin/breast-cancer-wisconsin.data"
url <- paste(loc, ds, sep="") breast <- read.table(url, sep=",", header=FALSE, na.strings="?")
names(breast) <- c("ID", "clumpThickness", "sizeUniformity",
"shapeUniformity", "maginalAdhesion",
"singleEpithelialCellSize", "bareNuclei",
"blandChromatin", "normalNucleoli", "mitosis", "class")
df <- breast[-1]
df$class <-factor(df$class,levels = c(2,4),labels = c('begign','malignant'))
set.seed(1234)
train <- sample(nrow(df),0.7*nrow(df))
df.train <- df[train,]
df.validate <- df[-train,]
table(df.train$class)
table(df.validate$class)
2.逻辑回归
# 拟合逻辑回归
fit.logit <- glm(class~.,data=df.train,family = binomial())
prob <- predict(fit.logit,df.validate,type='response')
# 对训练集外的样本进行分类
logit.pred <- factor(prob>.5,levels = c(F,T),labels = c('benign','malignant'))
# 评估预测的准确性
logit.pref <- table(df.validate$class,logit.pred,dnn = c('Actual','Predicted'))
logit.pref

结论:正确分类的模型是97%
3.决策树
library(rpart)
set.seed(1234)
# 生成树
dtree <- rpart(class~.,data = df.train,method = 'class',parms = list(split='information'))
plotcp(dtree)
# 剪枝
dtree.pruned <- prune(dtree,cp=.0125)
library(rpart.plot)
prp(dtree.pruned,type = 2,extra = 104,fallen.leaves = T,main='decision tree')
# 对训练集外的样本单元分类
dtree.pred <- predict(dtree.pruned,df.validate,type='class')
dtree.pref <- table(df.validate$class,dtree.pred,dnn = c('Actual','Predict'))
dtree.pref
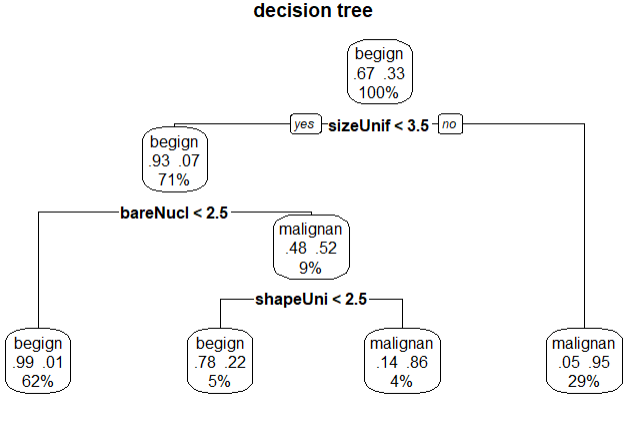
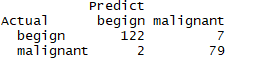
结论:验证的准确率96%
4.条件推断树
library(party)
library(partykit)
fit.tree <- ctree(class~.,data=df.train)
plot(fit.tree,main='Condition Inference Tree')
ctree.pred <- predict(fit.tree,df.validate,type='response')
ctree.pref <- table(df.validate$class,ctree.pred,dnn = c('Actual','Predicted'))
ctree.pref
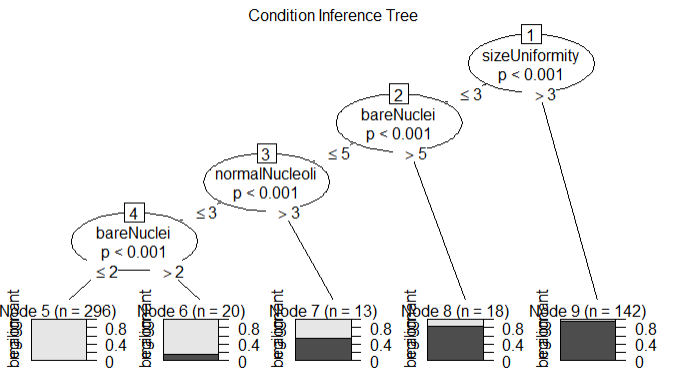
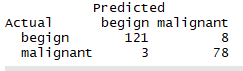
结论:验证的准确率97%
5.随机森林
library(randomForest)
set.seed(1234)
# 生成森林
fit.forest <- randomForest(class~.,data=df.train,na.action=na.roughfix,importance=T)
importance(fit.forest,type=2)
forest.pred <- predict(fit.forest,df.validate)
# 对训练集外的样本点分类
forest.pref <- table(df.validate$class,forest.pred,dnn = c('Actual','Predicted'))
forest.pref

结论:验证准确率在98%
6.支持向量机
library(e1071)
set.seed(1234)
fit.svm <- svm(class~.,data=df.train)
svm.pred <- predict(fit.svm,na.omit(df.validate))
svm.pref <- table(na.omit(df.validate)$class,svm.pred,dnn = c('Actual','Predicted'))
svm.pref

结论:验证准确率在96%
7.带有RBF内核的支持向量机
set.seed(1234)
# 通过调整gamma和c来拟合模型
tuned <- tune.svm(class~.,data=df.train,gamma = 10^(-6:1),cost = 10^(-10:10))
tuned
fit.svm <- svm(class~.,data=df.train,gamma=.01,cost=1)
svm.pred <- predict(fit.svm,na.omit(df.validate))
svm.pref <- table(na.omit(df.validate)$class,svm.pred,dnn = c('Actual','Predicted'))
svm.pref

结论验证的成功率有97%
8.编写函数选择预测效果最好的解
performance <- function(table,n=2){
if(!all(dim(table) == c(2,2))){
stop('Must be a 2 * 2 table')
}
tn = table[1,1]
fp = table[1,2]
fn = table[2,1]
tp = table[2,2]
sensitivity = tp/(tp+fn)
specificity = tn/(tn+fp)
ppp = tp/(tp+fp)
npp = tn/(tn+fn)
hitrate = (tp+tn)/(tp+tn+fn+fp)
result <- paste("Sensitivity = ", round(sensitivity, n) ,
"\nSpecificity = ", round(specificity, n),
"\nPositive Predictive Value = ", round(ppp, n),
"\nNegative Predictive Value = ", round(npp, n),
"\nAccuracy = ", round(hitrate, n), "\n", sep="")
cat(result)
}
performance(logit.pref)
performance(dtree.pref)
performance(ctree.pref)
performance(forest.pref)
performance(svm.pref)

结论:从以上的分类器中,本案例随机森林的拟合度最优
三.使用rattle进行数据挖掘
案例:预测糖尿病
library(rattle)
rattle()

结论:设定好这些变量点击执行

选择model选项卡,然后选择条件推断树作为预测模型,点击Draw生成图片

通过Evalute选项卡来评估模型

结论:只有35%的病人被成功鉴别,我们可以试试随机森林和支持向量机的匹配度是否更高
R语言-聚类与分类的更多相关文章
- 15、R语言聚类树的绘图原理
聚类广泛用于数据分析.去年研究了一下R语言聚类树的绘图原理.以芯片分析为例,我们来给一些样品做聚类分析.聚类的方法有很多种,我们选择Pearson距离.ward方法. 选择的样品有: "GS ...
- R语言聚类方法&主要软件包-K-means
主要4中软件包 stas:主要包含基本统计函数. cluster:用于聚类分析. fpc:含聚类算法函数(固定聚类.线性回归聚类等). mclust:处理高斯分布混合模型,通过EM算法实现聚类.分类及 ...
- R语言常用包分类总结
常用包: ——数据处理:lubridata ,plyr ,reshape2,stringr,formatR,mcmc: ——机器学习:nnet,rpart,tree,party,lars,boost, ...
- 大数据基础--R语言(刘鹏《大数据》课后习题答案)
1.R语言是解释性语言还是编译性语言? 解释性语言 2.简述R语言的基本功能. R语言是一套完整的数据处理.计算和制图软件系统,主要包括以下功能: (1)数据存储和处理功能,丰富的数据读取与存 ...
- R语言
什么是R语言编程? R语言是一种用于统计分析和为此目的创建图形的编程语言.不是数据类型,它具有用于计算的数据对象.它用于数据挖掘,回归分析,概率估计等领域,使用其中可用的许多软件包. R语言中的不同数 ...
- 分类算法的R语言实现案例
最近在读<R语言与网站分析>,书中对分类.聚类算法的讲解通俗易懂,和数据挖掘理论一起看的话,有很好的参照效果. 然而,这么好的讲解,作者居然没提供对应的数据集.手痒之余,我自己动手整理了一 ...
- R语言学习笔记—决策树分类
一.简介 决策树分类算法(decision tree)通过树状结构对具有某特征属性的样本进行分类.其典型算法包括ID3算法.C4.5算法.C5.0算法.CART算法等.每一个决策树包括根节点(root ...
- 机器学习-K-means聚类及算法实现(基于R语言)
K-means聚类 将n个观测点,按一定标准(数据点的相似度),划归到k个聚类(用户划分.产品类别划分等)中. 重要概念:质心 K-means聚类要求的变量是数值变量,方便计算距离. 算法实现 R语言 ...
- 分类-回归树模型(CART)在R语言中的实现
分类-回归树模型(CART)在R语言中的实现 CART模型 ,即Classification And Regression Trees.它和一般回归分析类似,是用来对变量进行解释和预测的工具,也是数据 ...
随机推荐
- c指针作为参数传递以及指针的指针
指针作为函数参数传递 函数参数传递的只能是数值,所以当指针作为函数参数传递时,传递的是指针的值,而不是地址. #include "stdio.h" void pointer(int ...
- RAC某节点v$asm_disk查询hang分析处理
主题:RAC某节点v$asm_disk查询hang分析处理 环境:Oracle 11.2.0.3 RAC 故障描述:RAC环境2个节点,节点1查询v$asm_disk正常返回结果,节点2查询v$asm ...
- SpringMvc4.x---快捷的ViewController
@RequestMapping("/index") public String hello(){ return "index"; } 此处无任何的业务处理,只是 ...
- [转]sysctl -P 报错解决办法
问题症状 修改 linux 内核文件 #vi /etc/sysctl.conf后执行sysctl -P 报错 error: "net.bridge.bridge-nf-call-ip6ta ...
- python_协程方式操作数据库
# !/usr/bin/python3 # -*- coding: utf-8 -*- import requests import gevent import pymysql from gevent ...
- grails项目中(DB的相关操作)
grails项目中(DB的相关操作) save:保存Domain对象的数据到对应的库表中(可能是insert也可能是update) findBy: 动态方法,查找并返回第一条记录,方法名可以变化 eg ...
- ansible-playbook相关
获取目标主机的信息 ansible all -m setup -a "filter=ansible_os_family" 不执行仅测试 ```sh 安装一个zabbix-agent ...
- Sql Server的艺术(一) 视图的增删查改
视图是从一个或者多个表中查询数据的另一种方式.利用视图可以集中.简化定制数据库,同时还能保障安全. 视图其结构和数据是建立在对应的查询基础上的.和表一样,视图也是包括几个被定义的数据列和多个数据行,但 ...
- Python算法——二叉树
一.二叉树 from collections import deque class BiTreeNode: def __init__(self, data): self.data = data sel ...
- easyUI---datagrid合并单元格代码实现
1.html部分: <div id="table1"></div> 2.js部分: $('#table1').datagrid({ data : data, ...
