(数据科学学习手札17)线性判别分析的原理简介&Python与R实现
之前数篇博客我们比较了几种具有代表性的聚类算法,但现实工作中,最多的问题是分类与定性预测,即通过基于已标注类型的数据的各显著特征值,通过大量样本训练出的模型,来对新出现的样本进行分类,这也是机器学习中最多的问题,而本文便要介绍分类算法中比较古老的线性判别分析:
线性判别
最早提出合理的判别分析法者是R.A.Fisher(1936),Fisher提出将线性判别函数用于花卉分类上,将花卉的各种特征利用线性组合方法变成单变量值,即将高维数据利用线性判别函数进行线性变化投影到一条直线上,再利用单值比较方法来对新样本进行分类,主要步骤如下:
Step1:求线性判别函数;
Step2:计算判别界值;
Step3:建立判别标准(这里与模糊分类中的隶属度有些相似,即离哪一类的投影中心最近,就将样本判别为哪一类)
下面分别利用Python,R,基于著名的花卉分类数据集iris进行演示:
Python
我们利用sklearn包中封装的LinearDiscriminantAnalysis对iris构建线性判别模型,因为LDA实际上是将高维数据尽可能分开的投影到一条直线上,因此LDA也可以对特定数据进行降维转换:
'''Fisher线性判别分析'''
import numpy as np
import matplotlib.pyplot as plt
from sklearn import datasets
from sklearn.discriminant_analysis import LinearDiscriminantAnalysis
from matplotlib.pyplot import style
from sklearn.model_selection import train_test_split style.use('ggplot') iris = datasets.load_iris() X = iris.data
y = iris.target '''展示LDA的降维功能'''
target_names = iris.target_names '''设置压缩到1维'''
lda = LinearDiscriminantAnalysis(n_components=1) '''利用线性判别函数将四维的样本数据压缩到一条直线上'''
X_r2 = lda.fit(X,y).transform(X)
X_Zero = np.zeros(X_r2.shape)
'''绘制降维效果图'''
for c,i,target_names in zip('ryb',[0,1,2],target_names):
plt.scatter(X_r2[y == i],X_Zero[y == i],c=c,label=target_names,s=5) plt.legend()
plt.grid()
降维后的效果图如下:
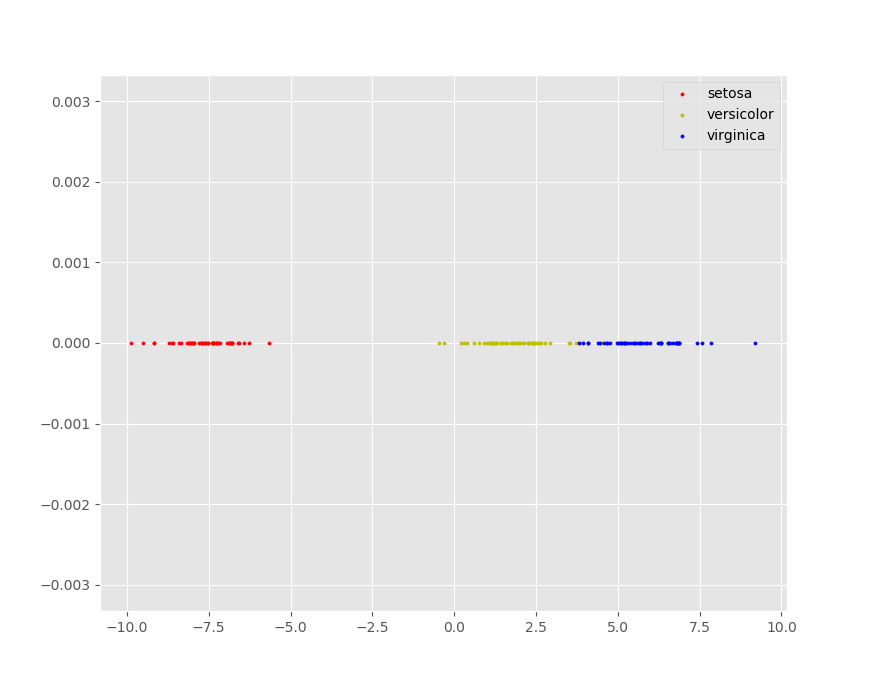
下面正式对iris数据集进行LDA分类,这里用到一个新的方法,是sklearn.model_selection.train_test_split,它的作用是根据设置的训练集与测试集的比例进行随机分割,我们利用从样本集中分割的7成数据作为训练集,3成数据进行测试,过程及结果如下:
'''利用sklearn自带的样本集划分方法进行分类,这里选择训练集测试集73开'''
X_train,X_test,y_train,y_test = train_test_split(X,y,test_size=0.3)
'''搭建LDA模型'''
lda = LinearDiscriminantAnalysis(n_components=1)
'''利用分割好的训练集进行模型训练并对测试集进行预测'''
ld = lda.fit(X_train,y_train).predict(X_test)
'''比较预测结果与真实分类结果'''
print(np.array([ld,y_test]))
'''打印正确率'''
print('正确率:',str(lda.score(X_test,y_test)))
结果如下:

可以看出,在iris上取得了非常高的准确率。
R
在R中做LDA需要用到MASS包中的lda(formula~feature1+feature2+...+featuren,data=df),其中formula表示数据集中表示分类标注的列,右边的各种feature表示将要使用到的分类特征值,也即是构建线性判别函数要用到的基础变量,data指保存全部数据的数据框,具体过程如下:
> #Fisher线性判别
> rm(list=ls())
> library(MASS)
> data(iris)
> data <- iris
> data$Species = as.character(data$Species)
> #创造类别变量
> data$type[data$Species == 'setosa'] = 1
> data$type[data$Species == 'versicolor'] = 2
> data$type[data$Species == 'virginica'] = 3
> #利用简单随机抽样将样本集划分为训练集与验证集
> sam <- sample(1:length(data[,1]),105)
> train <- data[sam,]
> test <- data[-sam,]
> #根据样本数据创建线性判别分析模型
> ld <- lda(type~Sepal.Length+Sepal.Width+Petal.Length+Petal.Width,data=train)
> #将样本集作为验证集求分类结果
> Z <- predict(ld)
> #保存预测类别
> newType <- Z$class
> #与真实分类结果进行比较
> cbind(train$type,Z$x,newType)
LD1 LD2 newType
74 2 -2.0419740 -1.16551052 2
93 2 -1.2286978 -1.34868982 2
124 3 -4.2056288 -0.39378496 3
43 1 7.0499296 -0.38479505 1
141 3 -6.3297144 1.43240686 3
112 3 -5.2009805 -0.39665336 3
75 2 -1.2076317 -0.53028083 2
83 2 -0.8607055 -1.04255122 2
128 3 -3.7958024 0.38283281 3
134 3 -3.5521695 -0.79338125 2
84 2 -4.1930475 -0.86304028 3
80 2 0.2455854 -1.48598565 2
16 1 8.9754828 2.99336222 1
35 1 6.7862663 -0.74053169 1
64 2 -2.4399568 -0.53150309 2
79 2 -2.4024680 -0.24830622 2
142 3 -4.9647448 1.48158101 3
54 2 -2.2217845 -1.93554530 2
14 1 7.3092968 -0.99420385 1
119 3 -8.8253015 -0.67042900 3
71 2 -3.3701925 0.94839271 3
44 1 6.2098650 0.99672773 1
29 1 7.6841973 0.08156876 1
62 2 -1.7099434 0.15332464 2
59 2 -1.6648086 -0.67372500 2
77 2 -2.3932456 -0.84052244 2
27 1 6.6078477 0.36272030 1
101 3 -7.2690741 1.94879260 3
91 2 -2.1739793 -1.53761667 2
40 1 7.4330661 0.03443993 1
103 3 -5.9833783 0.46642759 3
121 3 -5.9399677 1.45493705 3
51 2 -1.3784535 0.24058778 2
88 2 -2.5153892 -2.12899905 2
6 1 7.5334551 1.60652494 1
148 3 -4.7085798 0.61397476 3
76 2 -1.3919721 -0.13234144 2
10 1 7.0733099 -0.92823629 1
7 1 7.0416936 0.27174257 1
104 3 -5.1966351 -0.20937105 3
24 1 6.0120553 0.24387476 1
137 3 -6.0383747 2.20984770 3
61 2 -1.2257443 -3.03469429 2
41 1 7.6466593 0.57623503 1
26 1 6.4775174 -1.04708182 1
70 2 -1.0443574 -1.74662921 2
31 1 6.5351351 -0.78766052 1
85 2 -2.5818237 0.01276122 2
36 1 7.5972776 -0.33972390 1
4 1 6.6085362 -0.73929708 1
130 3 -4.2970292 -0.42496657 3
95 2 -1.8418994 -0.99664464 2
111 3 -4.1644831 1.17871159 3
117 3 -4.7101562 0.09594447 3
48 1 6.9765285 -0.43315849 1
106 3 -7.0303766 0.13158736 3
69 2 -3.5167119 -2.05931691 2
131 3 -5.9675456 -0.52249341 3
92 2 -2.0719645 -0.22536449 2
22 1 7.3872921 1.18564382 1
39 1 6.6977209 -0.90199151 1
125 3 -5.3082628 1.33894916 3
47 1 7.9455957 1.02129249 1
108 3 -5.9474167 -0.54626897 3
123 3 -7.2281863 -0.62126561 3
25 1 6.4877846 -0.15448692 1
96 2 -0.9672993 -0.40896608 2
149 3 -5.4267988 2.11763536 3
58 2 -0.1967945 -1.90480909 2
100 2 -1.4146638 -0.69091758 2
107 3 -4.3326495 -0.90276305 3
89 2 -1.1216984 -0.17330958 2
67 2 -2.4633370 0.01193815 2
45 1 6.7958450 1.25408059 1
138 3 -4.5932952 0.35495424 3
30 1 6.6519961 -0.52865075 1
17 1 8.3010066 1.79668640 1
9 1 6.3297286 -1.20813011 1
135 3 -4.6952607 -1.73516107 3
18 1 7.5140148 0.52828312 1
145 3 -6.4564371 2.08976755 3
68 2 -0.6703941 -1.51304115 2
12 1 7.0634482 -0.01186583 1
23 1 8.4484974 0.79139590 1
20 1 7.8504400 1.25653745 1
15 1 9.4800594 1.72576966 1
147 3 -5.0367690 -0.77081719 3
46 1 6.4557628 -0.76347342 1
60 2 -1.7902528 -0.66467281 2
19 1 7.8221244 1.15898751 1
144 3 -6.3829867 1.36026786 3
1 1 7.8010583 0.34057853 1
90 2 -1.8695758 -1.41834884 2
53 2 -2.2846205 0.07502495 2
115 3 -6.4317784 0.89801781 3
146 3 -5.4512238 1.17626549 3
78 2 -3.3451866 0.14511863 2
99 2 0.3864166 -1.31670824 2
118 3 -6.0412297 2.34012590 3
38 1 8.1457196 0.41229523 1
139 3 -3.6631579 0.43078471 3
105 3 -6.4339941 0.70414177 3
132 3 -4.7729922 2.10651473 3
110 3 -6.3994587 2.67334311 3
72 2 -0.9858025 -0.64502336 2
> #打印混淆矩阵
> (tab <- table(newType,train$type)) newType 1 2 3
1 35 0 0
2 0 33 1
3 0 2 34
> #显示正确率
> cat('Accuracy:',sum(diag(tab))/length(train[,1]))
Accuracy: 0.9714286
> #将验证集代入训练好的模型中计算近似泛化误差
> T <-predict(ld,test)
> #与真实分类结果进行比较
> cbind(test$type,T$x,T$class)
LD1 LD2
2 1 6.8020498 -0.95158956 1
3 1 7.2276597 -0.38602966 1
5 1 7.9179194 0.59958829 1
8 1 7.3738228 0.03485147 1
11 1 8.1391093 0.80900002 1
13 1 7.0298500 -1.13888262 1
21 1 7.2270204 -0.06187541 1
28 1 7.6684138 0.29262662 1
32 1 7.0367090 0.40861452 1
33 1 9.0120836 1.65651141 1
34 1 9.2707624 2.14912000 1
37 1 8.2299196 0.38647275 1
42 1 5.2371900 -2.52488607 1
49 1 8.0798659 0.80941155 1
50 1 7.3896062 -0.17620640 1
52 2 -1.6371815 0.52584233 2
55 2 -2.4742435 -0.55650250 2
56 2 -2.1822152 -0.88107904 2
57 2 -2.1911397 0.87747596 2
63 2 -1.2405414 -2.75931501 2
65 2 -0.3383635 -0.19420599 2
66 2 -1.1566243 0.12584525 2
73 2 -3.6967069 -1.47409522 2
81 2 -1.0878173 -1.95727554 2
82 2 -0.6088858 -2.09743977 2
86 2 -1.8089897 1.23238953 2
87 2 -2.0193315 0.17092876 2
94 2 -0.3136555 -2.16381886 2
97 2 -1.4304473 -0.47985972 2
98 2 -1.3261184 -0.52945776 2
102 3 -5.1726649 -0.29910341 3
109 3 -6.0478550 -1.34049085 3
113 3 -5.3935569 0.65782366 3
114 3 -5.6792727 -0.58064337 3
116 3 -5.4686329 1.64715619 3
120 3 -4.5946379 -2.29619567 3
122 3 -5.0183151 0.24310322 3
126 3 -4.9026833 0.37255836 3
127 3 -3.8968800 -0.08723483 3
129 3 -6.1746269 0.09473298 3
133 3 -6.4616705 0.28243757 3
136 3 -6.5857811 0.74428684 3
140 3 -4.9663213 0.96355072 3
143 3 -5.1726649 -0.29910341 3
150 3 -4.2980648 0.28857515 3
> #打印混淆矩阵
> (tab <- table(T$class,test$type)) 1 2 3
1 15 0 0
2 0 15 0
3 0 0 15
> #显示正确率
> cat('Accuracy:',sum(diag(tab))/length(test[,1]))
Accuracy: 1
> #Fisher线性判别
> rm(list=ls())
> library(MASS)
> data(iris)
> data <- iris
> data$Species = as.character(data$Species)
> #创造类别变量
> data$type[data$Species == 'setosa'] = 1
> data$type[data$Species == 'versicolor'] = 2
> data$type[data$Species == 'virginica'] = 3
> #利用简单随机抽样将样本集划分为训练集与验证集
> sam <- sample(1:length(data[,1]),105)
> train <- data[sam,]
> test <- data[-sam,]
> #根据样本数据创建线性判别分析模型
> ld <- lda(type~Sepal.Length+Sepal.Width+Petal.Length+Petal.Width,data=train)
> #将样本集作为验证集求分类结果
> Z <- predict(ld)
> #保存预测类别
> newType <- Z$class
> #与真实分类结果进行比较
> cbind(train$type,Z$x,newType)
LD1 LD2 newType
44 1 6.5356943 1.380095755 1
150 3 -5.0777607 0.353229454 3
48 1 7.2092979 -0.233679182 1
17 1 8.7810490 1.832128789 1
11 1 8.5106371 0.591364581 1
13 1 7.4261015 -0.949875862 1
82 2 -0.7168056 -1.805629506 2
19 1 8.2170070 0.870266056 1
123 3 -7.9020752 -1.233817765 3
23 1 8.7554448 0.980676590 1
59 2 -1.8788410 -0.915972039 2
70 2 -1.2367635 -1.516788339 2
146 3 -5.6852127 1.788579457 3
142 3 -5.0723482 2.082228707 3
115 3 -6.9597642 1.869494439 3
7 1 7.2188221 0.472577805 1
112 3 -5.6667730 -0.156736416 3
117 3 -5.4232144 -0.112017744 3
26 1 6.8888800 -0.860736454 1
111 3 -4.6405840 1.346460523 3
29 1 8.1491362 0.125965565 1
81 2 -1.2242456 -1.597693375 2
14 1 7.6286098 -0.598455540 1
110 3 -7.1444731 2.585994755 3
40 1 7.7798631 0.022078382 1
39 1 6.9782244 -0.504346286 1
68 2 -1.0104685 -1.671771426 2
20 1 8.0502422 1.117799195 1
31 1 6.7929797 -0.656064848 1
134 3 -4.1319678 -1.006493240 2
73 2 -4.0110318 -1.372974920 2
4 1 6.8227612 -0.537268125 1
108 3 -6.6972047 -1.217364668 3
54 2 -2.3695531 -1.376553027 2
148 3 -5.1575482 0.848139667 3
141 3 -6.8305530 1.864676411 3
109 3 -6.6173517 -1.422188092 3
50 1 7.7923809 -0.058826655 1
91 2 -2.7513769 -1.544578568 2
97 2 -1.8728790 -0.435815301 2
96 2 -1.4911207 -0.557876549 2
77 2 -2.5464899 -1.021564544 2
66 2 -1.2439344 0.003029162 2
145 3 -7.0771380 2.462076368 3
38 1 8.3218303 0.213545770 1
45 1 6.7744745 0.999164236 1
58 2 -0.3713519 -1.340382308 2
35 1 7.1622529 -0.552177665 1
36 1 8.1741719 -0.035844508 1
57 2 -2.7067243 0.719387605 2
27 1 6.9079284 0.551777520 1
132 3 -5.6031449 1.030132762 3
128 3 -4.3392145 0.561003821 3
87 2 -2.2635929 -0.006748767 2
88 2 -2.4886790 -1.851739918 2
118 3 -7.1004619 1.347087687 3
119 3 -9.4291409 -0.890616169 3
46 1 6.9234400 -0.316289540 1
33 1 9.0573501 1.063438766 1
10 1 7.4135836 -0.868970825 1
78 2 -3.6650096 0.105534552 2
32 1 7.6166932 0.640755164 1
139 3 -4.1962691 0.674830697 3
103 3 -6.5226603 0.373114540 3
43 1 7.2390794 -0.114882460 1
121 3 -6.4785762 1.623818440 3
15 1 10.1229028 1.482252025 1
125 3 -6.0718150 1.194903725 3
92 2 -2.5655637 -0.379597733 2
21 1 7.6071362 -0.210545218 1
116 3 -6.0199587 2.084095792 3
95 2 -2.2468978 -0.820309281 2
93 2 -1.3874483 -1.124059988 2
135 3 -5.6483660 -2.247095684 3
52 2 -1.9604386 0.420606745 2
37 1 8.8751646 0.414644969 1
127 3 -4.2307963 0.275427178 3
137 3 -6.8919261 2.468751536 3
98 2 -1.5631688 -0.569521563 2
72 2 -1.0384324 -0.432712540 2
80 2 0.2825956 -1.208391313 2
67 2 -3.1266046 0.070901690 2
120 3 -4.9979151 -2.051118149 3
114 3 -6.2027782 0.131952933 3
69 2 -3.4910411 -1.516772693 2
130 3 -4.8967334 -1.106964081 3
42 1 5.9270652 -1.555646363 1
64 2 -2.9521004 -0.683186676 2
86 2 -2.4035699 1.146743118 2
75 2 -1.3368410 -0.579461256 2
143 3 -5.8335378 0.090796722 3
140 3 -5.3380144 1.122071295 3
1 1 8.1663998 0.325667325 1
83 2 -1.0009115 -0.820471045 2
28 1 8.0234544 0.211840448 1
85 2 -3.3529324 0.080841383 2
89 2 -1.5995061 -0.127256512 2
18 1 7.9150690 0.642460485 1
147 3 -5.2724641 -0.214659306 3
105 3 -7.1968652 0.828583809 3
41 1 8.0580144 0.756287362 1
63 2 -1.1801689 -2.546513654 2
122 3 -5.6685504 0.829975549 3
107 3 -5.0735507 -0.234382628 3
47 1 8.0454637 0.692149004 1
> #打印混淆矩阵
> (tab <- table(newType,train$type)) newType 1 2 3
1 36 0 0
2 0 33 1
3 0 0 35
> #显示正确率
> cat('Accuracy:',sum(diag(tab))/length(train[,1]))
Accuracy: 0.9904762> #将验证集代入训练好的模型中计算近似泛化误差
> T <-predict(ld,test)
> #与真实分类结果进行比较
> cbind(test$type,T$x,T$class)
LD1 LD2
2 1 7.2879346 -0.63805255 1
3 1 7.5785711 -0.12979200 1
5 1 8.1836634 0.52536908 1
6 1 7.7566121 1.39670067 1
8 1 7.6666992 0.02704823 1
9 1 6.5916877 -0.80793523 1
12 1 7.1842622 -0.07186911 1
16 1 9.2604596 2.57316476 1
22 1 7.6684840 1.23986044 1
24 1 6.3832248 0.56001189 1
25 1 6.4159345 -0.39844020 1
30 1 6.8102433 -0.45636309 1
34 1 9.5320477 1.66891133 1
49 1 8.3974733 0.59633443 1
51 2 -1.5423430 -0.14371955 2
53 2 -2.5494836 -0.23440252 2
55 2 -2.6250939 -0.47214778 2
56 2 -2.7716342 -0.95711830 2
60 2 -2.1825564 -0.15706564 2
61 2 -1.2921165 -2.34199387 2
62 2 -2.0187853 0.38256324 2
65 2 -0.4493874 0.22229672 2
71 2 -4.0485780 1.06926437 3
74 2 -2.5798663 -1.51150491 2
76 2 -1.4875258 -0.18673290 2
79 2 -2.8043766 -0.14370961 2
84 2 -4.8532177 -0.86952245 3
90 2 -2.1086982 -0.98708920 2
94 2 -0.3886155 -1.54008407 2
99 2 0.5024002 -0.51222584 2
100 2 -1.7471972 -0.52169018 2
101 3 -8.2981213 2.15538466 3
102 3 -5.8335378 0.09079672 3
104 3 -6.0360789 -0.40566699 3
106 3 -7.7496056 -0.41373390 3
113 3 -5.8377150 0.82345220 3
124 3 -4.5041691 -0.03313161 3
126 3 -5.6507584 -0.30162799 3
129 3 -6.8073348 0.34501073 3
131 3 -6.4535806 -0.88255921 3
133 3 -7.0586655 0.66180389 3
136 3 -6.8585571 0.75916772 3
138 3 -5.4059508 0.08768402 3
144 3 -7.1039586 1.41107423 3
149 3 -6.2415407 2.37464228 3
> #打印混淆矩阵
> (tab <- table(T$class,test$type)) 1 2 3
1 14 0 0
2 0 15 0
3 0 2 14
> #显示正确率
> cat('Accuracy:',sum(diag(tab))/length(test[,1]))
Accuracy: 0.9555556
可以看出,和Python中的效果相差无几。
以上就是关于线性判别的基本内容,如有意见望提出。
(数据科学学习手札17)线性判别分析的原理简介&Python与R实现的更多相关文章
- (数据科学学习手札18)二次判别分析的原理简介&Python与R实现
上一篇我们介绍了Fisher线性判别分析的原理及实现,而在判别分析中还有一个很重要的分支叫做二次判别,本文就对二次判别进行介绍: 二次判别属于距离判别法中的内容,以两总体距离判别法为例,对总体G1,, ...
- (数据科学学习手札23)决策树分类原理详解&Python与R实现
作为机器学习中可解释性非常好的一种算法,决策树(Decision Tree)是在已知各种情况发生概率的基础上,通过构成决策树来求取净现值的期望值大于等于零的概率,评价项目风险,判断其可行性的决策分析方 ...
- (数据科学学习手札08)系统聚类法的Python源码实现(与Python,R自带方法进行比较)
聚类分析是数据挖掘方法中应用非常广泛的一项,而聚类分析根据其大体方法的不同又分为系统聚类和快速聚类,其中系统聚类的优点是可以很直观的得到聚类数不同时具体类中包括了哪些样本,而Python和R中都有直接 ...
- (数据科学学习手札34)多层感知机原理详解&Python与R实现
一.简介 机器学习分为很多个领域,其中的连接主义指的就是以神经元(neuron)为基本结构的各式各样的神经网络,规范的定义是:由具有适应性的简单单元组成的广泛并行互连的网络,它的组织能够模拟生物神经系 ...
- (数据科学学习手札44)在Keras中训练多层感知机
一.简介 Keras是有着自主的一套前端控制语法,后端基于tensorflow和theano的深度学习框架,因为其搭建神经网络简单快捷明了的语法风格,可以帮助使用者更快捷的搭建自己的神经网络,堪称深度 ...
- (数据科学学习手札72)用pdpipe搭建pandas数据分析流水线
1 简介 在数据分析任务中,从原始数据读入,到最后分析结果出炉,中间绝大部分时间都是在对数据进行一步又一步的加工规整,以流水线(pipeline)的方式完成此过程更有利于梳理分析脉络,也更有利于查错改 ...
- (数据科学学习手札75)基于geopandas的空间数据分析——坐标参考系篇
本文对应代码已上传至我的Github仓库https://github.com/CNFeffery/DataScienceStudyNotes 1 简介 在上一篇文章中我们对geopandas中的数据结 ...
- (数据科学学习手札55)利用ggthemr来美化ggplot2图像
一.简介 R中的ggplot2是一个非常强大灵活的数据可视化包,熟悉其绘图规则后便可以自由地生成各种可视化图像,但其默认的色彩和样式在很多时候难免有些过于朴素,本文将要介绍的ggthemr包专门针对原 ...
- (数据科学学习手札50)基于Python的网络数据采集-selenium篇(上)
一.简介 接着几个月之前的(数据科学学习手札31)基于Python的网络数据采集(初级篇),在那篇文章中,我们介绍了关于网络爬虫的基础知识(基本的请求库,基本的解析库,CSS,正则表达式等),在那篇文 ...
随机推荐
- 扫描FTP,保存文件
1.需求:某公司ftp服务器中一个文件夹中有30个文件(文件名字是不同的),每五分钟产生一个新的文件,同时删除这三十个文件中最早产生的文件,该文件夹中始终保持30个文件. 现在需要采集一周的数据做研究 ...
- 管道(Pipelines)模型
Pipeline模型最早被使用在Unix操作系统中.据称,假设说Unix是计算机文明中最伟大的发明,那么,Unix下的Pipe管道就是尾随Unix所带来的还有一个伟大的发明[1].我觉得管道的出现,所 ...
- LA 4254 贪心
题意:有 n 个工作,他的允许的工作时间是 [l,r] ,工作量是 v ,求CPU最速度的最小值. 分析: 可能太久没有做题了,竟然脑子反应好慢的.还是很容易想到二分,但是二分怎么转移呢? 可以看出, ...
- csu 1947 三分
题意: 长者对小明施加了膜法,使得小明每天起床就像马丁的早晨一样. 今天小明早上6点40醒来后发现自己变成了一名高中生,这时马上就要做早操了,小明连忙爬起来 他看到操场密密麻麻的人,突然灵光一闪想到了 ...
- 【[JLOI2014]松鼠的新家】
//第一次A掉紫题就来写题解,我是不是疯了 //说实话这道题还是比较裸的树上差分 //对于树上的一条路径(s,t),我们只需要把ch[s]++,ch[t]++,ch[LCA(S,T)]--,再把lca ...
- 【洛谷P1039】侦探推理
侦探推理 题目链接 这是一道恶心至极的模拟题 我们可以枚举罪犯是谁,今天是星期几,从而判断每个人说的话是真是假 若每个人说的话的真假一致,且说谎话的人数<=k且说真话的人数<=m-k,就是 ...
- 【luogu P1314 聪明的质监员】 题解
题目链接:https://www.luogu.org/problemnew/show/P1314 二分答案 但是计算区间贡献的时候 直接暴力会挂 前缀和加速 #include <cstdio&g ...
- 学习Node.js知识小结
什么是Node.js 官方解释:Node.js 是一个基于 Chrome V8 引擎的 JavaScript 运行环境. Node.js使用了一个事件驱动.非阻塞式I/O的模型( Node.js的特性 ...
- vue使用v-for循环,动态修改element-ui的el-switch
在使用element-ui的el-switch中,因为要用v-for循环,一直没有成功,后来仔细查看文档,发现可以这样写 <el-switch v-for="(item, key) i ...
- Mysql jar包
密码cngb https://pan.baidu.com/share/init?surl=bSGA6T-LTwjx-qaNAiipCA
