UVALive 3716 DNA Regions
题目大意:给定两个长度相等的字符串A和B,与一个百分比p%,求最长的、失配不超过p%的区间长度。O(nlogn)。
题目比较简单套路,推推式子就好了。
记S[i]表示到下标i一共有多少个失配,就相当于前缀和。那么对于一段区间[l,r],有以下式子成立:

然后转化一下得到:
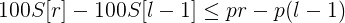
把变量相同的项放在一边:
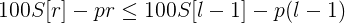
两边形式是一样的,不妨设:
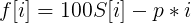
则有:
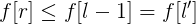
枚举右端点r,找到左边最左边的、f值大于等于它的值,即为对于r的答案。
怎么找呢?一种方法是按f值sort一下,把下标扔进堆里,或者随便搞搞就可以了。
还有一种方法是记录前缀最大值,二分查找即可,复杂度O(nlogn),可以过去。
还有一种复杂度相同,但常数更小的方法:
看上面那个前缀最大值,答案一定在出现变化的点上。
我们把变化的点记录下来,在这上面二分就好了。
因为数组长度会变小,所以可以大力降常。
#include <iostream>
#include <cstdio>
#include <cstdlib>
#include <algorithm>
#include <vector>
#include <cstring>
#include <queue>
#include <complex>
#include <stack>
#define LL long long int
#define dob double
#define FILE "3716"
using namespace std; const int N = ;
int n,p,S[N],f[N],bin[N],tot,Mx,Ans;
char A[N],B[N]; inline int gi(){
int x=,res=;char ch=getchar();
while(ch>''||ch<''){if(ch=='-')res*=-;ch=getchar();}
while(ch<=''&&ch>='')x=x*+ch-,ch=getchar();
return x*res;
} int main()
{
freopen(FILE".in","r",stdin);
freopen(FILE".out","w",stdout);
while((n=gi())&&(p=gi())){
scanf("%s",A+);scanf("%s",B+);
for(int i=;i<=n;++i){
S[i]=S[i-]+(A[i]!=B[i]);
f[i]=*S[i]-p*i;
}
bin[tot=]=Mx=;bin[n+]=n;Ans=-S[];
for(int i=;i<=n;++i){
if(f[i]>f[Mx]){bin[++tot]=Mx=i;continue;}
register int l=,r=tot,ans=n+;
while(l<=r){
int mid=(l+r)>>;
if(f[bin[mid]]<f[i])l=mid+;
else ans=mid,r=mid-;
}
Ans=max(Ans,i-bin[ans]);
}
Ans?printf("%d\n",Ans):printf("No solution.\n");
}
fclose(stdin);fclose(stdout);
return ;
}
DNA Regions
UVALive 3716 DNA Regions的更多相关文章
- UVALive 3716 DNA Regions ——(扫描法)
乍一看这个问题似乎是很复杂,但其实很好解决. 先处理出每个点到原点的距离和到x正半轴的角度(从x正半轴逆时针旋转的角度).然后以后者进行排序. 枚举每一个点到圆心的距离,作为半径,并找出其他到圆心距离 ...
- UVALive 3716 DNA Regions ——(式子变形)
一开始直接想到了二分,写了一发然后过了全部样例就交了,果断WA.因为这个问题显然是不满足单调性的. 然后想之前刚做的斜率优化DP,但是那个是求斜率最大值,不是求满足斜率大于一定值的最大长度的.也构造不 ...
- uvalive 3602 DNA Consensus String
https://vjudge.net/problem/UVALive-3602 题意: 给定m个长度均为n的DNA序列,求一个DNA序列,使得它到所有的DNA序列的汉明距离最短,若有多个解则输出字典序 ...
- UVALive 6663 Count the Regions --离散化+DFS染色
题意:给你n(n<=50)个矩形(左上角坐标和右下角坐标),问这些矩形总共将平面分成多少个部分.坐标值可能有1e9. 分析:看到n和坐标的范围,容易想到离散化,当时就没想到离散化以后怎么判断区域 ...
- [UVALive 6663 Count the Regions] (dfs + 离散化)
链接:https://icpcarchive.ecs.baylor.edu/index.php? option=com_onlinejudge&Itemid=8&page=show_p ...
- UVALive 6663 Count the Regions 离散+bfs染色_(:зゝ∠)_
题目链接:option=com_onlinejudge&Itemid=8&page=show_problem&problem=4675">点击打开链接 gg.. ...
- UvaLive 6663 Count the Regions 离散化+DFS
链接:http://vjudge.net/problem/viewProblem.action?id=49408 题意:在平面内给出若干个矩形,求出它们能将整个平面分成多少份. 思路:刚開始一眼看到认 ...
- DNA拷贝数变异CNV检测——基础概念篇
DNA拷贝数变异CNV检测——基础概念篇 一.CNV 简介 拷贝数异常(copy number variations, CNVs)是属于基因组结构变异(structural variation), ...
- UVALive 4262——Trip Planning——————【Tarjan 求强连通分量个数】
Road Networks Time Limit:3000MS Memory Limit:0KB 64bit IO Format:%lld & %llu Submit Stat ...
随机推荐
- 苹果快速的修复了Mac OS High Sierra 上出现了root的漏洞
最近苹果因为Mac最新系统 Mac OS High Sierra 上出现了root的漏洞走上了风口浪尖,不过还好,在一封苹果给科技媒体'9to5 Mac'的回复中得知,苹果在接收到报告之后,立即展开修 ...
- 十一、Hadoop学习笔记————数据库与数据仓库
数据仓库是集成的面向主题的数据库的集合 面向主题主要是宏观上解决某一类问题,集合性指数据集 数据库主要处理用于事务处理,数据仓库用于分析处理,数据库适用于操作型数据,便于增删改查, 数据仓库则用于挖掘 ...
- JAVA基础3——常见关键字解读(1)
常见的JAVA中的关键字 static static静态变量 静态变量:使用static关键字定义的变量.static可以修饰变量和方法,也有static静态代码块.被static修饰的成员变量和成员 ...
- 如何滚动更新 Service?- 每天5分钟玩转 Docker 容器技术(102)
在前面的实验中,我们部署了多个副本的服务,本节将讨论如何滚动更新每一个副本. 滚动更新降低了应用更新的风险,如果某个副本更新失败,整个更新将暂停,其他副本则可以继续提供服务.同时,在更新的过程中,总是 ...
- JavaScript正则表达式(Regular Expression):RegExp对象
第一部分:新建正则表达式 JavaScript中正则表达式是参照Perl 5(一门历史很悠久的语言,现在tiobe编程语言排行依然在10名左右)建立的. 新建正则表达式的方法有两种: 1.使用字面量( ...
- powershell 监控, 重启网卡
#powershell 监控, 重启网卡 一台Windows服务器,由于负荷较大,偶尔会网络中断,就写了个powershell脚本,监控ip是否通,不通就重启网卡 cls $host.UI.RawUI ...
- Python将纳入高考?
最近,"Python将纳入高考"的消息,狠狠地刷了朋友圈. 尽管这则消息目前还未得到官方的确认,但人们对于Python的火热关注度,还是引来众程序员热议. 虽然小编资历尚浅,但还是 ...
- Yii2如何添加sql日志记录的配置信息
在使用Yii2框架的时候,常常会出现没有sql日志记录的问题.在代码里一句一句的打印sql语句也不现实.所以就要用文件记录起来. 在 config/web.php 里面的 log配置中增加如下配置 [ ...
- 网页设计——4.html基本标签链接,图片,表格
今天主要认识一个有趣的html标签 1.超链接标签a 基本结构:<a href="url" >内容 </a> url就是网页地址. 但直接用这种超链接,会自 ...
- Web前端学习——HTML
HTML其实还是蛮容易学习的,无非就是一些标签.格式的填写,大学的时候也做过网站设计,所以这里主要记录一些常用的HTML标签.属性以及书写方法等. 一.常见HTML格式 主要包含文件type,html ...
