寻找与疾病相关的SNP位点——R语言从SNPedia批量提取搜索数据
SNP是单核苷酸多态性,人的基因是相似的,有些位点上存在差异,这种某个位点的核苷酸差异就做单核苷酸多态性,它影响着生物的性状,影响着对某些疾病的易感性。SNPedia是一个SNP调査百科,它引用各种已经发布的文章,或者数据库信息对SNP位点进行描述,共享着人类基因组变异的信息。我们可以搜索某个SNP位点来寻找与之相关的信息,也可以根据相关疾病,症状来寻找相关的SNP。
初次使用SNPedia
SNPedia主页网址为http://snpedia.com/index.php/SNPedia,比如我想查找与crouzon综合症相关的SNP,只需要在SNPedia中搜索crouzon syndrome,即会出现许多相关的SNP搜索结果

如果这时候我想看每个SNP的相关信息,我就要每个链接分别点进去
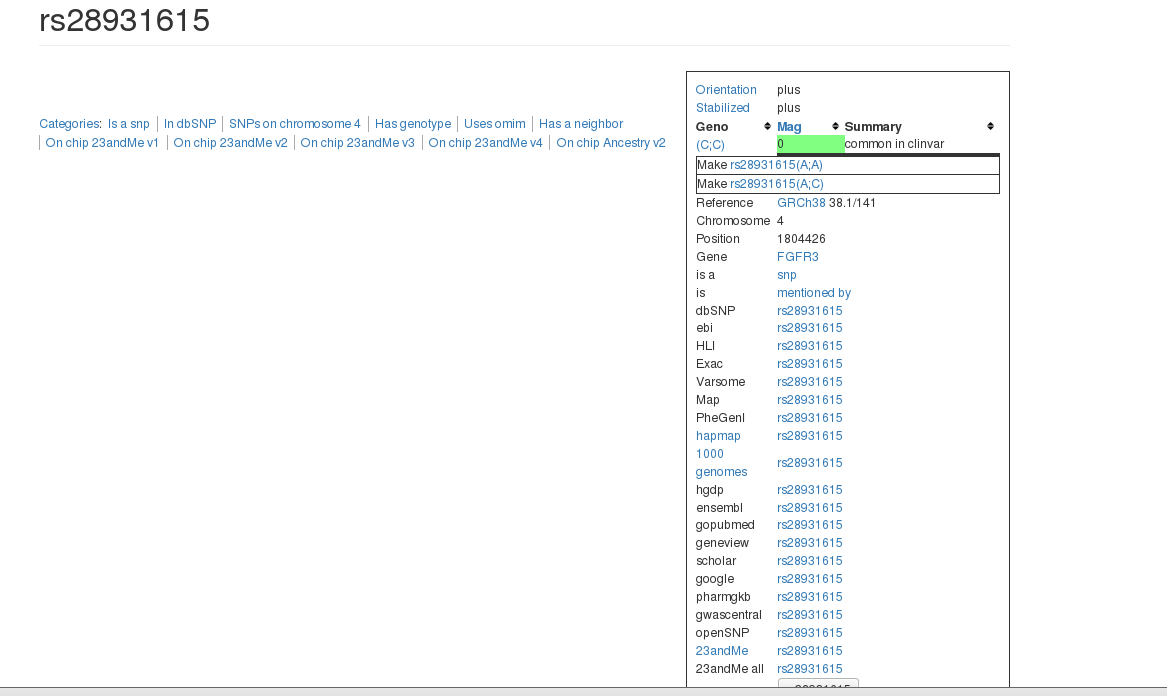
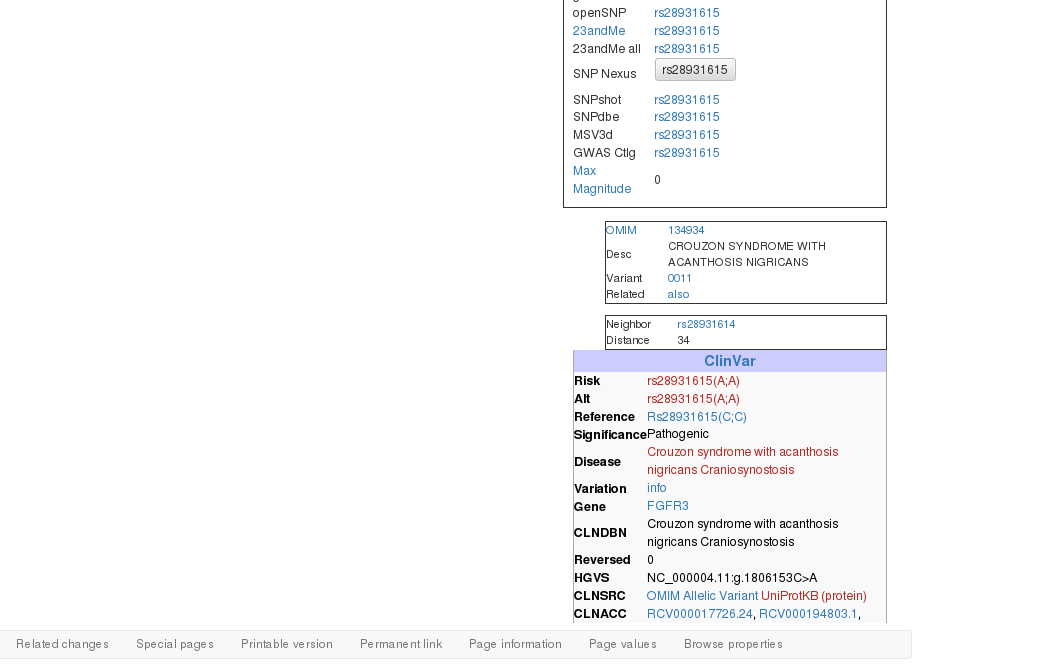
后来发现我们只需要提取里面的部分信息,Orientation,Stabilized,Reference,Chromosome,Position,Gene,还有clinvar表格信息,这时候我们就可以从网页中利用RCurl包,XML包,正则表达是把所需要的内容提取出来,有效抓取有用信息。
知识准备
RCurl包和XML包
在前一篇博文R语言从小木虫网页批量提取考研调剂信息 http://www.cnblogs.com/ywliao/p/6420501.html中已经提过,这里再提一个XML包中之前没有介绍的函数。
readHTMLTable(doc) #doc 是XML或者HTML格式文本,可以是文件名,也可以是刚刚parse的html对象,该函数返回XML或HTML中的表格
正则表达式
这里阐述基本的正则表达式使用 **
[ ]中括号,匹配中括号里面的任意字符,例如[a]匹配"a"
[a-z]表示匹配a到z任意字母,[A-Z]匹配大写A到Z,[0-9]匹配0-9任意数字
[ ]*中括号加*表示匹配任意次,[ ]+表示匹配至少一次,例如[a-zA-z,;: ]+表示匹配小写和大写字母,;:和空格至少一次
[ a|b ] 匹配a或者b
直接输入字符,实现精确定位。比如"apple[a-zA-z,;: ]+",定位到apple开头的后面匹配小写和大写字母,;:和空格至少一次的内容
[\u4E00-\u9FA5]匹配汉字
**
R语言gregexpr函数
使用方法:```gregexpr(pattern,istring, fixed = FALSE) #pattern就是要匹配正则表达是,istring是待匹配的字符串矢量,比如c("abc","cdf"),fixed,
如果设置为true,默认pattern是真正的字符串,不会作为其它使用,相当于转义,
函数返回列表,包括每个字符串的匹配长度和是否匹配)
#实例
 这里直接上代码,代码里面有着详细解释,许多函数以后可以直接复制使用,或者放进一个自己做的R包
!/usr/bin/env Rscript
download <- function(strURL){
输入网址返回html树格式文件
strURL:网页链接地址 return: html树文件
h <- basicTextGatherer()# 查看服务器返回的头信息
txt <- getURL(strURL, headerfunction = h$update,.encoding="gbk") ## 字符串形式
htmlParse(txt,asText=T,encoding="gbk") #选择gbk进行网页的解析
}
getinf <- function(strURL){
主要提取网页信息函数
strURL:网页链接网址 return:包括所要的所有信息的data.frame
doc<- download(strURL)
写如标题
info<- data.frame("Title"=strsplit(xmlValue(getNodeSet(doc,'//title')[[1]])," -")[[1]][1]) #"rs... - SNPedia"进行split
写入"Geno Mag Summary "table
GMS_table <- readHTMLTable(doc)
GMS_index <- 0
if (length(GMS_table)>2){
for (p in 1:6){
if (length(GMS_table[[p]])3){
GMS_index <- p
}
}
}
if (GMS_index!=0){
for (i in 1:length(GMS_table[[GMS_index]])){
tmp <- ""
for (t in 1:nrow(GMS_table[[GMS_index]][i])){
if(tmp""){tmp <-as.vector(GMS_table[[GMS_index]][i][t,1])}else{
tmp <- paste(tmp,as.vector(GMS_table[[GMS_index]][i][t,1]),sep=";")
}
}
if (i1){info$Geno <-tmp}
else if (i2){info$Mag <-tmp}
else if (i==3){info$Summary <- tmp
tmp <- ""
}
}
}else{
info$Geno <-" "
info$Mag <-" "
info$Summary <- " "
}
写入剩下table信息
mes <- getNodeSet(doc,'//td')
mes2 <- list()
for (c in mes){
d <- xmlValue(c)
if (d""){
}else{
mes2=c(mes2,d)
}
}
tmp <- greg_return_string("Make[-A-Za-z0-9_.%;\(\), ]+",mes2)
if (length(tmp)2){info$"Make"=paste(strsplit(tmp[[1]]," ")[[1]][2],strsplit(tmp[[2]]," ")[[1]][2],sep=";")}else{info$"Make"=" "}
for (i in (1:length(pattlistMainTable))){
tmp <- greg_return_index(pattlistMainTable[[i]],mes2)
if (i1 && length(tmp)1){info$"Orientation"=strsplit(mes2[[tmp+1]],"\n")[[1]]}else if (i1 && length(tmp)!=1){info$"Orientation"=" "}
else if (i2 && length(tmp)1){info$"Stabilized"=strsplit(mes2[[tmp+1]],"\n")[[1]]}else if (i2 && length(tmp)!=1){info$"Stabilized"=" "}
else if (i3 && length(tmp)1){info$"Reference"=strsplit(mes2[[tmp+1]],"\n")[[1]]}else if (i3 && length(tmp)!=1){info$"Reference"=" "}
else if (i4 && length(tmp)1){info$"Chromosome"=strsplit(mes2[[tmp+1]],"\n")[[1]]}else if (i4 && length(tmp)!=1){info$"Chromosome"=" "}
else if (i5 &&length(tmp)1){info$"Position"=strsplit(mes2[[tmp+1]],"\n")[[1]]}else if (i5 && length(tmp)!=1){info$"Position"=" "}
else if (i6&&length(tmp)1){info$"Gene"=strsplit(mes2[[tmp+1]],"\n")[[1]]}else if (i6 && length(tmp)!=1){info$"Gene"=" "}
}
写入clivar
mes <- getNodeSet(doc,'//tr')
mes2 <- list()
for (c in mes){
d <- xmlValue(c)
if (d""){
}else{
mes2=c(mes2,d)
}
}
for (i in (1:length(pattlistClinvar))){
tmp <- greg_return_string(pattlistClinvar[i],mes2)
if (length(tmp)!=0){tmp <- tmp[[1]]}
if (i1 && length(tmp)!=0){info$"Risk"=strsplit(tmp,"\n")[[1]][3]}else if (i1 && length(tmp)0){info$"Risk"=" "}
else if (i2 && length(tmp)!=0){info$"Alt"=strsplit(tmp,"\n")[[1]][3]}else if (i2 && length(tmp)0){info$"Alt"=" "}
else if (i3 && length(tmp)!=0){info$"ReferenceBase"=strsplit(tmp,"\n")[[1]][3]}else if (i3&& length(tmp)0){info$"ReferenceBase"=" "}
else if (i4 && length(tmp)!=0){info$"Significance"=strsplit(tmp,"\n")[[1]][2]}else if (i4 && length(tmp)0){info$"Significance"=" "}
else if (i5&& length(tmp)!=0){info$"Disease "=strsplit(tmp,"\n")[[1]][3]}else if (i5 && length(tmp)0){info$"Disease "=" "}
else if (i6 && length(tmp)!=0){info$"CLNDBN"=strsplit(tmp,"\n")[[1]][3]}else if (i6 && length(tmp)0){info$"CLNDBN"=" "}
else if (i7 && length(tmp)!=0){info$"Reversed"=strsplit(tmp,"\n")[[1]][3]}else if (i7 && length(tmp)0){info$"Reversed"=" "}
else if (i8 && length(tmp)!=0){info$"HGVS"=strsplit(tmp,"\n")[[1]][3]}else if (i8 && length(tmp)0){info$"HGVS"=" "}
else if (i9 && length(tmp)!=0){info$"CLNSRC"=strsplit(tmp,"\n")[[1]][3]}else if (i9 && length(tmp)0){info$"CLNSRC"=" "}
else if (i10 && length(tmp)!=0){info$"CLNACC "=strsplit(tmp,"\n")[[1]][3]}else if (i10 && length(tmp)==0){info$"CLNACC "=" "}
}
info
}
greg_return_string <- function(pattern,stringlist){
greg_return_stirng 指定匹配全部字符串列表,返回匹配的字符串
pattern:匹配模式,比如"abc[a-z]*" stringlist:字符串列表,list("abc","abcde","cdfe") return : 列表里字符串匹配结果,"abc""abcde"
findlist <- gregexpr(pattern,stringlist)
needlist <- list()
for (i in which(unlist(findlist)>0)){
preadress <- substr(stringlist[i],findlist[[i]],findlist[[i]]+attr(findlist[[i]],'match.length')-1)
needlist<- c(needlist,list(preadress))
}
return(needlist)
}
greg_return_index <- function(pattern,stringlist){
greg_return_stirng 指定匹配全部字符串列表,返回存在匹配的字符串列表index
pattern:匹配模式 stringlst:待匹配字符串列表 return:存在返回匹配的字符串在列表中的index
findlist <- gregexpr(pattern,stringlist)
needlist <- list()
which(unlist(findlist)>0)
}
extradress <- function(strURL){
将strURL网页里面我们所需要链接提取出来并加工
strURL:网页链接网址 return:网址列表,包括所有提取加工后的网址链接
pattern <- "/index.php/Rs[0-9]+"
prefix <- "https://snpedia.com" #网址改为https起始
links <- getHTMLLinks(download(strURL)) # getHTMLLinks不能解析https的网址,因此先用download解析网址
needlinks <- gregexpr(pattern,links)
needlinkslist <- list()
for (i in which(unlist(needlinks)>0)){
preadress <- substr(links[i],needlinks[[i]],needlinks[[i]]+attr(needlinks[[i]],'match.length')-1)
needlinkslist<- c(needlinkslist,list(preadress))
adresses <- lapply(needlinkslist,function(x)paste(prefix,x,sep=""))
}
adresses
}
greg <- function(pattern,istring){
greg函数查看单个字符串istring,并且返回匹配的部分,不匹配返回空
gregout <- gregexpr(pattern,istring)
substr(istring,gregout[[1]],gregout[[1]]+attr(gregout[[1]],'match.length')-1)
}
library(RCurl)
library(XML)
自定义部分
strURL <- "https://snpedia.com/index.php?title=Special%3ASearch&profile=default&fulltext=Search&search=Congenital+adrenal+hyperplasia" #snpedia网址都已改为https开头
output <- "ouput.txt"
message(paste("[prog]",strURL,output,sep=" "))
strURLs <- extradress(strURL)
pattlistMainTable <- list("Orientation$","Stabilized$","Reference$","Chromosome$","Position$","Gene$")
此匹配模式列表用于返回该字符串所在index,而对应的值是index是该index+1
pattlistClinvar <- list("Risk\n\n[-A-Za-z0-9_.%;\(\), ]+","Alt\n\n[-A-Za-z0-9_.%;\(\), ]+",
"Reference\n\n[-A-Za-z0-9_.%;\(\) ]+","Significance \n[A-Za-z ]+","Disease \n\n[A-Za-z ]+",
"CLNDBN \n\n[-A-Za-z0-9_.% ]+","Reversed \n\n[0-9]+", "HGVS \n\n[-A-Za-z0-9_.%:> ]+","CLNSRC \n\n[-A-Za-z0-9_.% ]+","CLNACC \n\n[-A-Za-z0-9_.%, ]+")
此匹配模式列表用于返回相应clinvar
inf <- NULL
for ( strURL in strURLs){
dat <- getinf(strURL)
if (length(inf) == 0){
inf <- dat
}else{
inf <- rbind(inf,dat)
}
}
write.table(inf, file = output, row.names = F, col.names=T,quote = F, sep="\t") # tab 分隔的文件
message("完成!")
结果可以直接打开,也可以用excel的自文本打开,方便查看
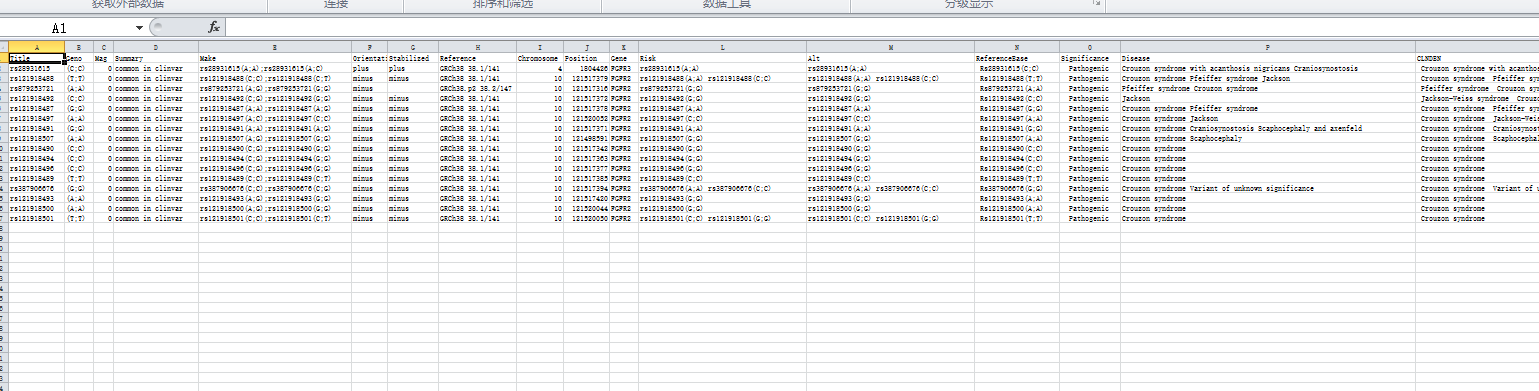寻找与疾病相关的SNP位点——R语言从SNPedia批量提取搜索数据的更多相关文章
- 利用R语言制作出漂亮的交互数据可视化
利用R语言制作出漂亮的交互数据可视化 利用R语言也可以制作出漂亮的交互数据可视化,下面和大家分享一些常用的交互可视化的R包. rCharts包 说起R语言的交互包,第一个想到的应该就是rCharts包 ...
- R语言数据分析利器data.table包—数据框结构处理精讲
R语言数据分析利器data.table包-数据框结构处理精讲 R语言data.table包是自带包data.frame的升级版,用于数据框格式数据的处理,最大的特点快.包括两个方面,一方面是写的快,代 ...
- 使用R语言的RTCGA包获取TCGA数据--转载
转载生信技能树 https://mp.weixin.qq.com/s/JB_329LCWqo5dY6MLawfEA TCGA数据源 - R包RTCGA的简单介绍 - 首先安装及加载包 - 指定任意基因 ...
- R语言基础入门之二:数据导入和描述统计
by 写长城的诗 • October 30, 2011 • Comments Off This post was kindly contributed by 数据科学与R语言 - go there t ...
- 用R语言分析我的fitbit计步数据
目标:把fitbit的每日运动记录导入到R语言中进行分析,画出统计图表来 已有原始数据:fitbit2014年每日的记录电子表格文件,全部数据点此下载,示例如下: 日期 消耗卡路里数 步 距离 攀爬楼 ...
- R语言数据分析利器data.table包 —— 数据框结构处理精讲
R语言data.table包是自带包data.frame的升级版,用于数据框格式数据的处理,最大的特点快.包括两个方面,一方面是写的快,代码简洁,只要一行命令就可以完成诸多任务,另一方面是处理 ...
- R语言data.table包fread读取数据
R语言处理大规模数据速度不算快,通过安装其他包比如data.table可以提升读取处理速度. 案例,分别用read.csv和data.table包的fread函数读取一个1.67万行.230列的表格数 ...
- R语言学习 第三篇:数据框
数据框(data.frame)是最常用的数据结构,用于存储二维表(即关系表)的数据,每一列存储的数据类型必须相同,不同数据列的数据类型可以相同,也可以不同,但是每列的行数(长度)必须相同.数据框的每列 ...
- R语言笔记001——读取csv格式数据
读取csv格式数据 数据来源是西南财经大学 司亚卿 老师的课程作业 方法一:read.csv()函数 file.choose() read.csv("C:\\Users\\Administr ...
随机推荐
- 解决mysql 1032 主从错误
1032错误----现在生产库中好多数据,在从库误删了,生产库更新后找不到了,现在主从不同步了,再跳过错误也没用,因为没这条,再更新还会报错 临时解决方案 mysql> stop slave; ...
- 3. 托管对象模型的迁移(Core Data 应用程序实践指南)
本章介绍如何添加模型版本及模型映射,演示几种迁移技术,供在升级模型时使用. 3.1. 修改托管对象模型 3.2. 添加模型版本 3.3. 轻量级迁移方式 3.4. 默认的迁移方式 3.5. 通过迁移管 ...
- EF Code First:实体映射
二.实体映射 实体与数据库的映射可以通过DataAnnotation与FluentAPI两种方式来进行映射: (一) DataAnnotation DataAnnotation 特性由.NET 3.5 ...
- Ninja介绍
什么是Ninja 在Unix/Linux下通常使用Makefile来控制代码的编译,但是Makefile对于比较大的项目有时候会比较慢,看看上面那副漫画,代码在编译都变成了程序员放松的借口了.所以这个 ...
- MVC伪一个12306图片验证码
本文的来由主要是满足自己的好奇心,而不是证明什么东西,如果涉及到什么官方性的事情,麻烦通知我谢谢:本篇将要和大家分享的是一个看起来通12306图片验证码相似的效果,这篇应该是今年农历最后一篇分享文章了 ...
- 通过Spring Data Neo4J操作您的图形数据库
在前面的一篇文章<图形数据库Neo4J简介>中,我们已经对其内部所使用的各种机制进行了简单地介绍.而在我们尝试对Neo4J进行大版本升级时,我发现网络上并没有任何成型的样例代码以及简介,而 ...
- 解决使用Idea/Eclipse编写Hadoop程序包依赖问题
解决使用Idea/Eclipse编写Hadoop程序包依赖问题 解决包依赖的一种简单粗暴方法就是, 把下载下来的Hadoop压缩包解压, 搜索里面所有的额jar包文件,然后复制到一个目录,在使用Ide ...
- ADO.NET高级应用
ADO.NET事务处理(4个步骤) 1.调用SqlConnection对象的BeginTransaction()方法,创建一个SqlTransaction对象,标志事务开始. 2.将创建的SqlTra ...
- 《JAVASCRIPT高级程序设计》错误处理与调试
一.错误处理 错误处理在程序设计中的重要性是毋庸置疑的,任何有影响力的web应用程序都需要一套完善的错误处理机制.良好的错误机制可以让用户得到提醒,知道发生了什么事. 1.try-catch语句 tr ...
- 《JAVASCRIPT高级程序设计》原生拖放和媒体元素
一.原生拖放 最早在网页中引入javascript拖放功能的是IE4,当时,网页中只有两种对象可以拖放:图像和某些文本.而现在,几乎网页中的任何元素都可以拖放以及作为放置目标.下面介绍一些与拖放相关的 ...
