Kmeans算法实现


下面的demo是根据kmeans算法原理实现的demo,使用到的数据是kmeans.txt
1.658985 4.285136
-3.453687 3.424321
4.838138 -1.151539
-5.379713 -3.362104
0.972564 2.924086
-3.567919 1.531611
0.450614 -3.302219
-3.487105 -1.724432
2.668759 1.594842
-3.156485 3.191137
3.165506 -3.999838
-2.786837 -3.099354
4.208187 2.984927
-2.123337 2.943366
0.704199 -0.479481
-0.392370 -3.963704
2.831667 1.574018
-0.790153 3.343144
2.943496 -3.357075
-3.195883 -2.283926
2.336445 2.875106
-1.786345 2.554248
2.190101 -1.906020
-3.403367 -2.778288
1.778124 3.880832
-1.688346 2.230267
2.592976 -2.054368
-4.007257 -3.207066
2.257734 3.387564
-2.679011 0.785119
0.939512 -4.023563
-3.674424 -2.261084
2.046259 2.735279
-3.189470 1.780269
4.372646 -0.822248
-2.579316 -3.497576
1.889034 5.190400
-0.798747 2.185588
2.836520 -2.658556
-3.837877 -3.253815
2.096701 3.886007
-2.709034 2.923887
3.367037 -3.184789
-2.121479 -4.232586
2.329546 3.179764
-3.284816 3.273099
3.091414 -3.815232
-3.762093 -2.432191
3.542056 2.778832
-1.736822 4.241041
2.127073 -2.983680
-4.323818 -3.938116
3.792121 5.135768
-4.786473 3.358547
2.624081 -3.260715
-4.009299 -2.978115
2.493525 1.963710
-2.513661 2.642162
1.864375 -3.176309
-3.171184 -3.572452
2.894220 2.489128
-2.562539 2.884438
3.491078 -3.947487
-2.565729 -2.012114
3.332948 3.983102
-1.616805 3.573188
2.280615 -2.559444
-2.651229 -3.103198
2.321395 3.154987
-1.685703 2.939697
3.031012 -3.620252
-4.599622 -2.185829
4.196223 1.126677
-2.133863 3.093686
4.668892 -2.562705
-2.793241 -2.149706
2.884105 3.043438
-2.967647 2.848696
4.479332 -1.764772
-4.905566 -2.911070
import numpy as np
import matplotlib.pyplot as plt
# 载入数据
data = np.genfromtxt("kmeans.txt", delimiter=" ") plt.scatter(data[:,],data[:,])
plt.show()
print(data.shape)
#训练模型
# 计算距离
def euclDistance(vector1, vector2):
return np.sqrt(sum((vector2 - vector1) ** )) # 初始化质心
def initCentroids(data, k):
numSamples, dim = data.shape
# k个质心,列数跟样本的列数一样
centroids = np.zeros((k, dim))
# 随机选出k个质心
for i in range(k):
# 随机选取一个样本的索引
index = int(np.random.uniform(, numSamples))#从一个均匀分布[low,high)中随机采样
# 作为初始化的质心
centroids[i, :] = data[index, :]
return centroids # 传入数据集和k的值
def kmeans(data, k):
# 计算样本个数
numSamples = data.shape[]
# 样本的属性,第一列保存该样本属于哪个簇,第二列保存该样本跟它所属簇的误差
clusterData = np.array(np.zeros((numSamples, )))
# 决定质心是否要改变的变量
clusterChanged = True # 初始化质心
centroids = initCentroids(data, k) while clusterChanged:
clusterChanged = False
# 循环每一个样本
for i in range(numSamples):
# 最小距离
minDist = 100000.0
# 定义样本所属的簇
minIndex =
# 循环计算每一个质心与该样本的距离
for j in range(k):
# 循环每一个质心和样本,计算距离
distance = euclDistance(centroids[j, :], data[i, :])
# 如果计算的距离小于最小距离,则更新最小距离
if distance < minDist:
minDist = distance
# 更新最小距离
clusterData[i, ] = minDist
# 更新样本所属的簇
minIndex = j # 如果样本的所属的簇发生了变化
if clusterData[i, ] != minIndex:
# 质心要重新计算
clusterChanged = True
# 更新样本的簇
clusterData[i, ] = minIndex # 更新质心
for j in range(k):
# 获取第j个簇所有的样本所在的索引
cluster_index = np.nonzero(clusterData[:, ] == j)
# 第j个簇所有的样本点
pointsInCluster = data[cluster_index]
# 计算质心
centroids[j, :] = np.mean(pointsInCluster, axis=)
# showCluster(data, k, centroids, clusterData) return centroids, clusterData # 显示结果
def showCluster(data, k, centroids, clusterData):
numSamples, dim = data.shape
if dim != :
print("dimension of your data is not 2!")
return # 用不同颜色形状来表示各个类别
mark = ['or', 'ob', 'og', 'ok', '^r', '+r', 'sr', 'dr', '<r', 'pr']
if k > len(mark):
print("Your k is too large!")
return # 画样本点
for i in range(numSamples):
markIndex = int(clusterData[i, ])
plt.plot(data[i, ], data[i, ], mark[markIndex]) # 用不同颜色形状来表示各个类别
mark = ['*r', '*b', '*g', '*k', '^b', '+b', 'sb', 'db', '<b', 'pb']
# 画质心点
for i in range(k):
plt.plot(centroids[i, ], centroids[i, ], mark[i], markersize=) plt.show()
# 设置k值
k =
# centroids 簇的中心点
# cluster Data样本的属性,第一列保存该样本属于哪个簇,第二列保存该样本跟它所属簇的误差
centroids, clusterData = kmeans(data, k)
if np.isnan(centroids).any():
print('Error')
else:
print('cluster complete!')
# 显示结果
showCluster(data, k, centroids, clusterData)
print(centroids)
# 做预测
x_test = [,]
np.tile(x_test,(k,))
# 误差
np.tile(x_test,(k,))-centroids
# 误差平方
(np.tile(x_test,(k,))-centroids)**
# 误差平方和
((np.tile(x_test,(k,))-centroids)**).sum(axis=)
# 最小值所在的索引号
np.argmin(((np.tile(x_test,(k,))-centroids)**).sum(axis=))
def predict(datas):
return np.array([np.argmin(((np.tile(data,(k,))-centroids)**).sum(axis=)) for data in datas])
#画出簇的作用区域
# 获取数据值所在的范围
x_min, x_max = data[:, ].min() - , data[:, ].max() +
y_min, y_max = data[:, ].min() - , data[:, ].max() + # 生成网格矩阵
xx, yy = np.meshgrid(np.arange(x_min, x_max, 0.02),
np.arange(y_min, y_max, 0.02)) z = predict(np.c_[xx.ravel(), yy.ravel()])# ravel与flatten类似,多维数据转一维。flatten不会改变原始数据,ravel会改变原始数据
z = z.reshape(xx.shape)
# 等高线图
cs = plt.contourf(xx, yy, z)
# 显示结果
showCluster(data, k, centroids, clusterData)
下面这个demo是使用sklearn库实现聚类
from sklearn.cluster import KMeans
import numpy as np
from matplotlib import pyplot as plt
data = np.genfromtxt('kmeans.txt',delimiter=" ")# 载入数据
k = # 设置k值
model = KMeans(n_clusters=)# 训练模型
model.fit(data)
print(model.labels_)# 结果
centers = model.cluster_centers_# 分类中心点坐标
print(centers)
result = model.labels_
mark = ['or','ob','og','oy']# 画出各个数据点,用不同颜色表示分类
for i,d in enumerate(data):
plt.plot(d[],d[],mark[result[i]])
mark = ['*r','*b','*g','*y']# 画出各个分类的中心点
for i,center in enumerate(centers):
plt.plot(center[],center[],mark[i],markersize = )
plt.show()
x_min, x_max = data[:,].min() - ,data[:,].max() +# 获取数据值所在的范围
y_min, y_max = data[:,].min() - ,data[:,].max() +
xx ,yy= np.meshgrid(np.arange(x_min,x_max,0.02),np.arange(y_min,y_max,0.02))# 生成网格矩阵
z = model.predict(np.c_[xx.ravel(),yy.ravel()])
z = z.reshape(xx.shape)
cs = plt.contourf(xx,yy,z)# 等高线图
mark = ['or','ob','og','oy']
for i,d in enumerate(centers):
plt.plot(d[],d[],mark[result[i]])
mark = ['*r','*b','*g','*y']
for i,center in enumerate(centers):
plt.plot(center[],center[],mark[i],markersize = )
plt.show()
当数据量很大的时候,会出现原始聚类算法效率很低,计算速度很慢,因此可以使用mini batch k-means在数据量很大的时候提高速度

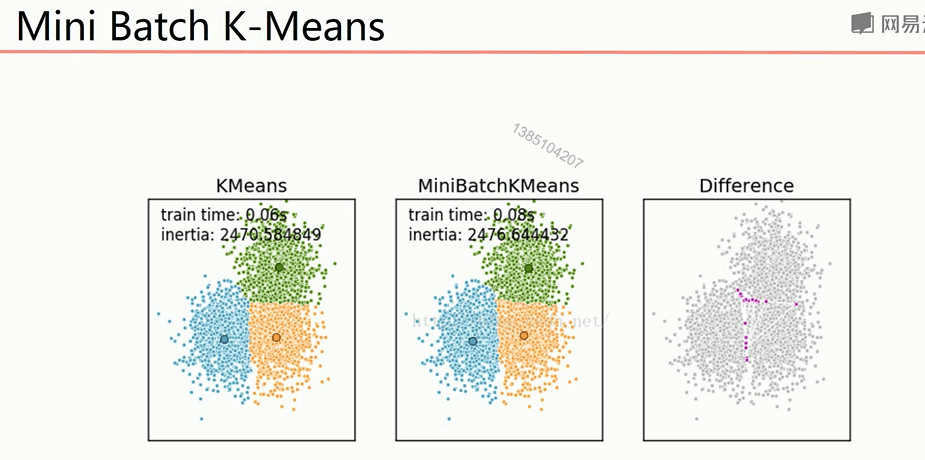
from sklearn.cluster import MiniBatchKMeans
import numpy as np
from matplotlib import pyplot as plt
data = np.genfromtxt('kmeans.txt',delimiter=" ")# 载入数据
k = # 设置k值
model = MiniBatchKMeans(n_clusters=)# 训练模型
model.fit(data)
print(model.labels_)# 结果
centers = model.cluster_centers_# 分类中心点坐标
print(centers)
result = model.labels_
mark = ['or','ob','og','oy']# 画出各个数据点,用不同颜色表示分类
for i,d in enumerate(data):
plt.plot(d[],d[],mark[result[i]])
mark = ['*r','*b','*g','*y']# 画出各个分类的中心点
for i,center in enumerate(centers):
plt.plot(center[],center[],mark[i],markersize = )
plt.show()
x_min, x_max = data[:,].min() - ,data[:,].max() +# 获取数据值所在的范围
y_min, y_max = data[:,].min() - ,data[:,].max() +
xx ,yy= np.meshgrid(np.arange(x_min,x_max,0.02),np.arange(y_min,y_max,0.02))# 生成网格矩阵
z = model.predict(np.c_[xx.ravel(),yy.ravel()])
z = z.reshape(xx.shape)
cs = plt.contourf(xx,yy,z)# 等高线图
mark = ['or','ob','og','oy']
for i,d in enumerate(centers):
plt.plot(d[],d[],mark[result[i]])
mark = ['*r','*b','*g','*y']
for i,center in enumerate(centers):
plt.plot(center[],center[],mark[i],markersize = )
plt.show()
当然传统聚类算法有一些弊端(sklearn提供的Kmeans已经解决了下面两个问题),如下
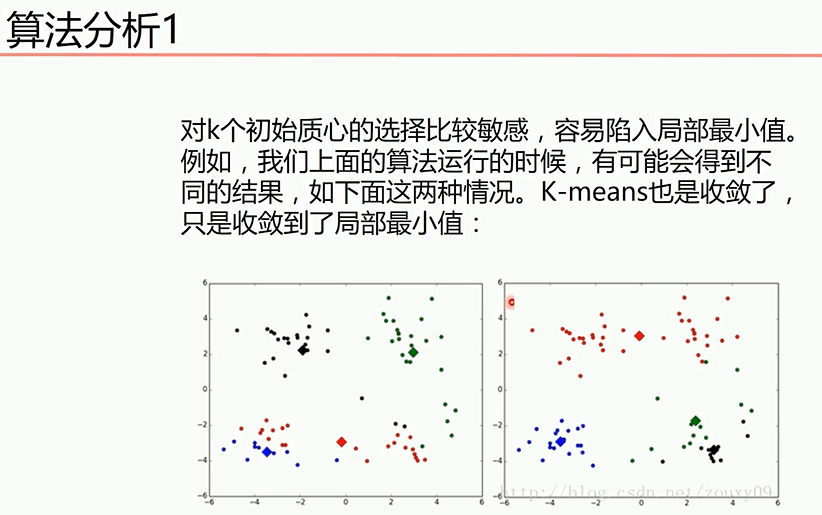
该问题可以通过多次初始化随机点的选取,然后选择代价函数最小的那个

下面的demo实现了优化
import numpy as np
import matplotlib.pyplot as plt
# 载入数据
data = np.genfromtxt("kmeans.txt", delimiter=" ") # 计算距离
def euclDistance(vector1, vector2):
return np.sqrt(sum((vector2 - vector1) ** )) # 初始化质心
def initCentroids(data, k):
numSamples, dim = data.shape
# k个质心,列数跟样本的列数一样
centroids = np.zeros((k, dim))
# 随机选出k个质心
for i in range(k):
# 随机选取一个样本的索引
index = int(np.random.uniform(, numSamples))
# 作为初始化的质心
centroids[i, :] = data[index, :]
return centroids # 传入数据集和k的值
def kmeans(data, k):
# 计算样本个数
numSamples = data.shape[]
# 样本的属性,第一列保存该样本属于哪个簇,第二列保存该样本跟它所属簇的误差
clusterData = np.array(np.zeros((numSamples, )))
# 决定质心是否要改变的变量
clusterChanged = True # 初始化质心
centroids = initCentroids(data, k) while clusterChanged:
clusterChanged = False
# 循环每一个样本
for i in range(numSamples):
# 最小距离
minDist = 100000.0
# 定义样本所属的簇
minIndex =
# 循环计算每一个质心与该样本的距离
for j in range(k):
# 循环每一个质心和样本,计算距离
distance = euclDistance(centroids[j, :], data[i, :])
# 如果计算的距离小于最小距离,则更新最小距离
if distance < minDist:
minDist = distance
# 更新样本所属的簇
minIndex = j
# 更新最小距离
clusterData[i, ] = distance # 如果样本的所属的簇发生了变化
if clusterData[i, ] != minIndex:
# 质心要重新计算
clusterChanged = True
# 更新样本的簇
clusterData[i, ] = minIndex # 更新质心
for j in range(k):
# 获取第j个簇所有的样本所在的索引
cluster_index = np.nonzero(clusterData[:, ] == j)
# 第j个簇所有的样本点
pointsInCluster = data[cluster_index]
# 计算质心
centroids[j, :] = np.mean(pointsInCluster, axis=)
# showCluster(data, k, centroids, clusterData) return centroids, clusterData # 显示结果
def showCluster(data, k, centroids, clusterData):
numSamples, dim = data.shape
if dim != :
print("dimension of your data is not 2!")
return # 用不同颜色形状来表示各个类别
mark = ['or', 'ob', 'og', 'ok', '^r', '+r', 'sr', 'dr', '<r', 'pr']
if k > len(mark):
print("Your k is too large!")
return # 画样本点
for i in range(numSamples):
markIndex = int(clusterData[i, ])
plt.plot(data[i, ], data[i, ], mark[markIndex]) # 用不同颜色形状来表示各个类别
mark = ['*r', '*b', '*g', '*k', '^b', '+b', 'sb', 'db', '<b', 'pb']
# 画质心点
for i in range(k):
plt.plot(centroids[i, ], centroids[i, ], mark[i], markersize=) plt.show() # 设置k值
k = min_loss =
min_loss_centroids = np.array([])
min_loss_clusterData = np.array([]) for i in range():
# centroids 簇的中心点
# cluster Data样本的属性,第一列保存该样本属于哪个簇,第二列保存该样本跟它所属簇的误差
centroids, clusterData = kmeans(data, k)
loss = sum(clusterData[:, ]) / data.shape[]
if loss < min_loss:
min_loss = loss
min_loss_centroids = centroids
min_loss_clusterData = clusterData # print('loss',min_loss)
print('cluster complete!')
centroids = min_loss_centroids
clusterData = min_loss_clusterData # 显示结果
showCluster(data, k, centroids, clusterData)
# 做预测
x_test = [,]
np.tile(x_test,(k,))
# 误差
np.tile(x_test,(k,))-centroids
# 误差平方
(np.tile(x_test,(k,))-centroids)**
# 误差平方和
((np.tile(x_test,(k,))-centroids)**).sum(axis=)
# 最小值所在的索引号
np.argmin(((np.tile(x_test,(k,))-centroids)**).sum(axis=))
def predict(datas):
return np.array([np.argmin(((np.tile(data,(k,))-centroids)**).sum(axis=)) for data in datas])
# 获取数据值所在的范围
x_min, x_max = data[:, ].min() - , data[:, ].max() +
y_min, y_max = data[:, ].min() - , data[:, ].max() + # 生成网格矩阵
xx, yy = np.meshgrid(np.arange(x_min, x_max, 0.02),
np.arange(y_min, y_max, 0.02)) z = predict(np.c_[xx.ravel(), yy.ravel()])# ravel与flatten类似,多维数据转一维。flatten不会改变原始数据,ravel会改变原始数据
z = z.reshape(xx.shape)
# 等高线图
cs = plt.contourf(xx, yy, z)
# 显示结果
showCluster(data, k, centroids, clusterData)
关于k值选择,一个方法是肘部法,另一个方法是按照自己的需要选择分为几类,下面的demo是根据肘部法选择K值
import numpy as np
import matplotlib.pyplot as plt
# 载入数据
data = np.genfromtxt("kmeans.txt", delimiter=" ") # 计算距离
def euclDistance(vector1, vector2):
return np.sqrt(sum((vector2 - vector1) ** )) # 初始化质心
def initCentroids(data, k):
numSamples, dim = data.shape
# k个质心,列数跟样本的列数一样
centroids = np.zeros((k, dim))
# 随机选出k个质心
for i in range(k):
# 随机选取一个样本的索引
index = int(np.random.uniform(, numSamples))
# 作为初始化的质心
centroids[i, :] = data[index, :]
return centroids # 传入数据集和k的值
def kmeans(data, k):
# 计算样本个数
numSamples = data.shape[]
# 样本的属性,第一列保存该样本属于哪个簇,第二列保存该样本跟它所属簇的误差
clusterData = np.array(np.zeros((numSamples, )))
# 决定质心是否要改变的变量
clusterChanged = True # 初始化质心
centroids = initCentroids(data, k) while clusterChanged:
clusterChanged = False
# 循环每一个样本
for i in range(numSamples):
# 最小距离
minDist = 100000.0
# 定义样本所属的簇
minIndex =
# 循环计算每一个质心与该样本的距离
for j in range(k):
# 循环每一个质心和样本,计算距离
distance = euclDistance(centroids[j, :], data[i, :])
# 如果计算的距离小于最小距离,则更新最小距离
if distance < minDist:
minDist = distance
# 更新样本所属的簇
minIndex = j
# 更新最小距离
clusterData[i, ] = distance # 如果样本的所属的簇发生了变化
if clusterData[i, ] != minIndex:
# 质心要重新计算
clusterChanged = True
# 更新样本的簇
clusterData[i, ] = minIndex # 更新质心
for j in range(k):
# 获取第j个簇所有的样本所在的索引
cluster_index = np.nonzero(clusterData[:, ] == j)
# 第j个簇所有的样本点
pointsInCluster = data[cluster_index]
# 计算质心
centroids[j, :] = np.mean(pointsInCluster, axis=)
# showCluster(data, k, centroids, clusterData) return centroids, clusterData # 显示结果
def showCluster(data, k, centroids, clusterData):
numSamples, dim = data.shape
if dim != :
print("dimension of your data is not 2!")
return # 用不同颜色形状来表示各个类别
mark = ['or', 'ob', 'og', 'ok', '^r', '+r', 'sr', 'dr', '<r', 'pr']
if k > len(mark):
print("Your k is too large!")
return # 画样本点
for i in range(numSamples):
markIndex = int(clusterData[i, ])
plt.plot(data[i, ], data[i, ], mark[markIndex]) # 用不同颜色形状来表示各个类别
mark = ['*r', '*b', '*g', '*k', '^b', '+b', 'sb', 'db', '<b', 'pb']
# 画质心点
for i in range(k):
plt.plot(centroids[i, ], centroids[i, ], mark[i], markersize=) plt.show() list_lost = []
for k in range(, ):
min_loss =
min_loss_centroids = np.array([])
min_loss_clusterData = np.array([])
for i in range():
# centroids 簇的中心点
# cluster Data样本的属性,第一列保存该样本属于哪个簇,第二列保存该样本跟它所属簇的误差
centroids, clusterData = kmeans(data, k)
loss = sum(clusterData[:, ]) / data.shape[]
if loss < min_loss:
min_loss = loss
min_loss_centroids = centroids
min_loss_clusterData = clusterData
list_lost.append(min_loss) # print('loss',min_loss)
# print('cluster complete!')
# centroids = min_loss_centroids
# clusterData = min_loss_clusterData # 显示结果
# showCluster(data, k, centroids, clusterData)
print(list_lost)
plt.plot(range(,),list_lost)
plt.xlabel('k')
plt.ylabel('loss')
plt.show()
# 做预测
x_test = [,]
np.tile(x_test,(k,))
# 误差
np.tile(x_test,(k,))-centroids
# 误差平方
(np.tile(x_test,(k,))-centroids)**
# 误差平方和
((np.tile(x_test,(k,))-centroids)**).sum(axis=)
# 最小值所在的索引号
np.argmin(((np.tile(x_test,(k,))-centroids)**).sum(axis=))
def predict(datas):
return np.array([np.argmin(((np.tile(data,(k,))-centroids)**).sum(axis=)) for data in datas])
# 获取数据值所在的范围
x_min, x_max = data[:, ].min() - , data[:, ].max() +
y_min, y_max = data[:, ].min() - , data[:, ].max() + # 生成网格矩阵
xx, yy = np.meshgrid(np.arange(x_min, x_max, 0.02),
np.arange(y_min, y_max, 0.02)) z = predict(np.c_[xx.ravel(), yy.ravel()])# ravel与flatten类似,多维数据转一维。flatten不会改变原始数据,ravel会改变原始数据
z = z.reshape(xx.shape)
# 等高线图
cs = plt.contourf(xx, yy, z)
# 显示结果
showCluster(data, k, centroids, clusterData)





from sklearn.cluster import DBSCAN
import numpy as np
from matplotlib import pyplot as plt
data = np.genfromtxt("kmeans.txt",delimiter=' ')
model = DBSCAN(eps=1.5,min_samples=)
model.fit(data)
result = model.fit_predict(data)
print(result)
mark = ['or', 'ob', 'og', 'oy', 'ok', 'om']
for i,d in enumerate(data):
plt.plot(d[],d[],mark[result[i]])
plt.show()
import numpy as np
import matplotlib.pyplot as plt
from sklearn import datasets
x1, y1 = datasets.make_circles(n_samples=, factor=0.5, noise=0.05)
x2, y2 = datasets.make_blobs(n_samples=, centers=[[1.2,1.2]], cluster_std=[[.]]) x = np.concatenate((x1, x2))
plt.scatter(x[:, ], x[:, ], marker='o')
plt.show()
from sklearn.cluster import KMeans
y_pred = KMeans(n_clusters=).fit_predict(x)
plt.scatter(x[:, ], x[:, ], c=y_pred)
plt.show()
from sklearn.cluster import DBSCAN
y_pred = DBSCAN().fit_predict(x)
plt.scatter(x[:, ], x[:, ], c=y_pred)
plt.show()
y_pred = DBSCAN(eps = 0.2).fit_predict(x)
plt.scatter(x[:, ], x[:, ], c=y_pred)
plt.show()
y_pred = DBSCAN(eps = 0.2, min_samples=).fit_predict(x)
plt.scatter(x[:, ], x[:, ], c=y_pred)
plt.show()
Kmeans算法实现的更多相关文章
- kmeans算法并行化的mpi程序
用c语言写了kmeans算法的串行程序,再用mpi来写并行版的,貌似参照着串行版来写并行版,效果不是很赏心悦目~ 并行化思路: 使用主从模式.由一个节点充当主节点负责数据的划分与分配,其他节点完成本地 ...
- 【原创】数据挖掘案例——ReliefF和K-means算法的医学应用
数据挖掘方法的提出,让人们有能力最终认识数据的真正价值,即蕴藏在数据中的信息和知识.数据挖掘 (DataMiriing),指的是从大型数据库或数据仓库中提取人们感兴趣的知识,这些知识是隐含的.事先未知 ...
- kmeans算法c语言实现,能对不同维度的数据进行聚类
最近在苦于思考kmeans算法的MPI并行化,花了两天的时间把该算法看懂和实现了串行版. 聚类问题就是给定一个元素集合V,其中每个元素具有d个可观察属性,使用某种算法将V划分成k个子集,要求每个子集内 ...
- kmeans算法实践
这几天学习了无监督学习聚类算法Kmeans,这是聚类中非常简单的一个算法,它的算法思想与监督学习算法KNN(K近邻算法)的理论基础一样都是利用了节点之间的距离度量,不同之处在于KNN是利用了有标签的数 ...
- 二分K-means算法
二分K-means聚类(bisecting K-means) 算法优缺点: 由于这个是K-means的改进算法,所以优缺点与之相同. 算法思想: 1.要了解这个首先应该了解K-means算法,可以看这 ...
- 视觉机器学习------K-means算法
K-means(K均值)是基于数据划分的无监督聚类算法. 一.基本原理 聚类算法可以理解为无监督的分类方法,即样本集预先不知所属类别或标签,需要根据样本之间的距离或相似程度自动进行分类.聚 ...
- EM算法(1):K-means 算法
目录 EM算法(1):K-means 算法 EM算法(2):GMM训练算法 EM算法(3):EM算法运用 EM算法(4):EM算法证明 EM算法(1) : K-means算法 1. 简介 K-mean ...
- K-means算法及文本聚类实践
K-Means是常用的聚类算法,与其他聚类算法相比,其时间复杂度低,聚类的效果也还不错,这里简单介绍一下k-means算法,下图是一个手写体数据集聚类的结果. 基本思想 k-means算法需要事先指定 ...
- K-means算法和矢量量化
语音信号的数字处理课程作业——矢量量化.这里采用了K-means算法,即假设量化种类是已知的,当然也可以采用LBG算法等,不过K-means比较简单.矢量是二维的,可以在平面上清楚的表示出来. 1. ...
- [聚类算法] K-means 算法
聚类 和 k-means简单概括. 聚类是一种 无监督学习 问题,它的目标就是基于 相似度 将相似的子集聚合在一起. k-means算法是聚类分析中使用最广泛的算法之一.它把n个对象根据它们的属性分为 ...
随机推荐
- linux 下无法输入# 显示为£
在键盘布局里面,(Keyboard Layout)设置为中国,汉语.解决问题
- GarsiaWachs算法
解决石子问题: 题目描述如下: 有n堆石子排成一列,每堆石子有一个重量w[i], 每次合并可以合并相邻的两堆石子,一次合并的代价为两堆石子的重量和w[i]+w[i+1].问安排怎样的合并顺序,能够使得 ...
- jq-demo-放大镜
<!DOCTYPE html> <html> <head> <meta charset="UTF-8"> <title> ...
- Kotlin Hello World
{ https://github.com/zhmmmm/Kotlin }
- spark在collect收集数据的时候出现outOfMemoryError:java heap space
spark的collect是action算子,所有最后会以数组的形式返回给driver端,当数据太大的时候就会出现堆内存溢出.OutofMemoryError:java heap space. 在sp ...
- Core Text 入门
本文所涉及的代码你可以在这里下载到 https://github.com/kejinlu/CTTest,包含两个项目,一个Mac的NSTextView的测试项目,一个iOS的Core Text的测试项 ...
- Dll注入技术之ComRes注入
DLL注入技术之ComRes注入 ComRes注入的原理是利用Windows 系统中C:\WINDOWS\system32目录下的ComRes.dll这个文件,当待注入EXE如果使用CoCreateI ...
- mysql查看数据库大小或者表大小
要想知道每个数据库的大小的话,步骤如下: 1.进入information_schema 数据库(存放了数据库的信息) use information_schema; 2.查询所有数据库的大小: sel ...
- beautifulsoup的使用
解析库 解析器 使用方法 优势 劣势 Python标准库 BeautifulSoup(markup, "html.parser") Python的内置标准库.执行速度适中 .文档容 ...
- 7.springboot+mybatis+redis整合
选择生成的依赖 选择保存的工程路径 查询已经生成的依赖,并修改mysql的版本 <dependencies> <dependency> <groupId>org.s ...
