序列比对之Biostrings包
基本概念
Biostrings包很重要的3个功能是进行Pairwise sequence alignment 和Multiple sequence alignment及 Pattern finding in a sequence
序列比对一般有2个过程:
1)构建计分矩阵公式(the scoring matrix formulation)
2)比对(alignment itself)
global alignment methods (全局比对):align every residue in the sequences ,例如Needleman-Wunsch algorithm.
local alignment technique(局部比对): align regions of high similarity in the sequences,例如Smith-Waterman algorithm
安装
if("Biostrings" %in% rownames(installed.packages()) == FALSE) {source("http://bioconductor.org/biocLite.R");biocLite("Biostrings")}
suppressMessages(library(Biostrings))
ls('package:Biostrings')
----------------Pairwise sequence alignment---------------
步骤:首先构建罚分规则,然后按照规则进行比对。用pairwiseAlignment()函数
举例1:核酸序列
(myScoringMat <- nucleotideSubstitutionMatrix(match = 1, mismatch = -1, baseOnly = TRUE))#构建罚分规则
gapOpen <- 2 #gap分为2
gapExtend <- 1 #延伸gap分为1 sequence1 <- "GAATTCGGCTA" #序列1
sequence2 <- "GATTACCTA" #序列2
myAlignment <- pairwiseAlignment(sequence1, sequence2,
substitutionMatrix = myScoringMat, gapOpening = gapOpen,
gapExtension = gapExtend, type="global", scoreOnly = FALSE) #进行比对
myAlignment

举例2:对蛋白序列进行比对
蛋白比对会更复杂,因此模型更多,
data(package="Biostrings") #查看所有数据集
data(BLOSUM62) #这里选择BLOSUM62数据
subMat <- "BLOSUM62" #赋值
gapOpen <- 2
gapExtend <- 1
sequence1 <- "PAWHEAE"
sequence2 <- "HEAGAWGHE"
myAlignProt <- pairwiseAlignment(sequence1, sequence2,
substitutionMatrix = subMat, gapOpening = gapOpen, gapExtension =
gapExtend, type="global", scoreOnly = FALSE) #全局比对
myAlignProt2 <- pairwiseAlignment(sequence1, sequence2,
substitutionMatrix = subMat, gapOpening = gapOpen, gapExtension =
gapExtend, type="local",scoreOnly = FALSE) ##局部比对
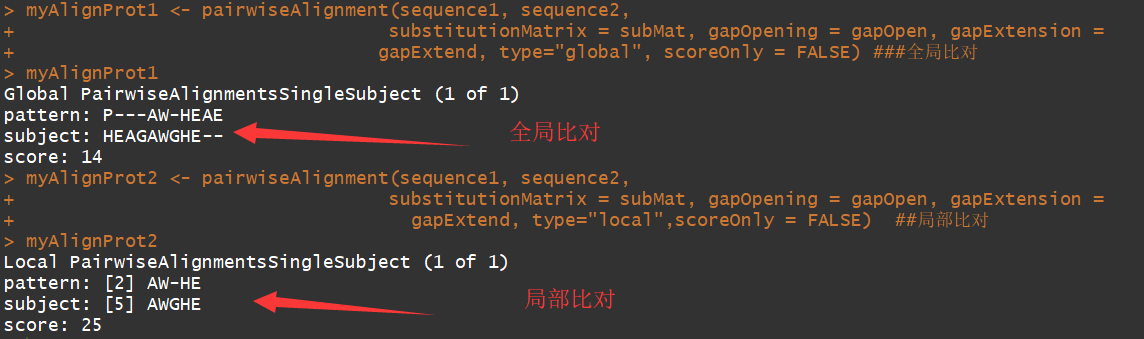
可以看到局部比对返回的是,高度相似的序列部分.
3)可视化,对于序列可以用最经典的对角线来可视化(以人和黑猩猩的hemoglobin beta为例)
library(seqinr) # 为了读取fasta序列
myseq <- read.fasta(file = "F:/R/Bioconductor/biostrings/prtein_example_seq.fas")
dotPlot(myseq[[1]], myseq[[2]], col=c("white", "red"), xlab="Human", ylab="Chimpanzee")

##########Multiple sequence alignment############
一般多序列比对可以用于进化分析
install.packages("muscle") #需要安装该包,因为该包在我的版本上没法安装,所以这里就不讲了
library(muscle)
######Phylogenetic analysis and tree plotting########
这里先不做分析
#########blast格式的解析######
install.packages("RFLPtools",dependencies=TRUE)
library(RFLPtools)
data(BLASTdata) #先查看数据集了解一下相关数据格式情况
head(BLASTdata)
colnames(BLASTdata)
DIR <- system.file("extdata", package = "RFLPtools") #用自带数据集
MyFile <- file.path(DIR, "BLASTexample.txt")
MyBLAST <- read.blast(file = MyFile)
mySimMat <- simMatrix(MyBLAST) #可以根据blast结果用来生成相似性矩阵,太厉害了
#########Pattern finding in a sequence######
library(Biostrings)
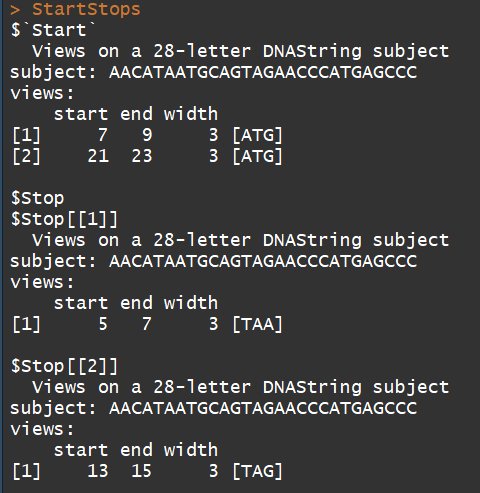
mynucleotide <- DNAString("aacataatgcagtagaacccatgagccc")
matchPattern(DNAString("ATG"), mynucleotide) #示例1
matchPattern("TAA", mynucleotide) #示例2
##以下函数可以用来寻找orf(需要修改)
myCodonFinder <- function(sequence){
startCodon = DNAString("ATG") # 指定起始密码子
stopCodons = list("TAA", "TAG", "TGA") # 指定终止密码子
codonPosition = list() #initialize the output to be returned as a list
codonPosition$Start = matchPattern(startCodon, sequence) # search start codons
x=list()
for(i in 1:3){ # iterate over all stop codons
x[[i]]= matchPattern(DNAString(stopCodons[[i]]), sequence)
codonPosition$Stop=x
}
return(codonPosition) # returns results
}
StartStops <- myCodonFinder(mynucleotide)
序列比对之Biostrings包的更多相关文章
- Python_序列与映射的解包操作
解包就是把序列或映射中每个元素单独提取出来,序列解包的一种简单用法就是把首个或前几个元素与后面几个元素分别提取出来,例如: first, seconde, *rest = sequence 如果seq ...
- Python 序列与映射的解包操作
解包就是把序列或映射中每个元素单独提取出来,序列解包的一种简单用法就是把首个或前几个元素与后面几个元素分别提取出来,例如: first, seconde, *rest = sequence 如果seq ...
- Python 序列与映射的解包操作-乾颐堂
解包就是把序列或映射中每个元素单独提取出来,序列解包的一种简单用法就是把首个或前几个元素与后面几个元素分别提取出来,例如: first, seconde, *rest = sequence 如果seq ...
- python基础之打/解包及运算符与控制流程
python基础之打/解包及运算符与控制流程 python中的解压缩(即序列类型的打包和解包) python提供了两个设计元祖和其他序列类型的处理的便利,也就是自动打包与自动解包功能,比如: data ...
- Oracle学习笔记一 初识Oracle
数据库简介 数据库(Database)是按照数据结构来组织.存储和管理数据的仓库.SQL 是 Structured Query Language(结构化查询语言)的首字母缩写词. 定义 数据库,简单来 ...
- Makefile <网络转载>
陈皓 (CSDN)概述——什 么是makefile?或许很多Winodws的程序员都不知道这个东西,因为那些Windows的IDE都为你做了这个工作,但我觉得要作一个好的和 professional的 ...
- oracle 体系结构
oracle 体系结构 数据库的体系结构是指数据库的组成.工作过程与原理,以及数据在数据库中的组织与管理机制. 1. oracle工作原理: 1).在数据库服务器上启动Oracle实例:2).应用程序 ...
- Navicat for Oracle实现连接Oracle
不知道为什么,从一开始,我就不喜欢Oracle,名字好听,功能强大,但总感觉"高不可攀";或许是因为我觉得其他的数据库就可以解决数据问题,不太了解Oracle的优势:而且它长得也不 ...
- 三、oracle 体系结构
1.oracle内存由SGA+PGA所构成 2.oracle数据库体系结构数据库的体系结构是指数据库的组成.工作过程与原理,以及数据在数据库中的组织与管理机制. oracle工作原理: 1).在数据库 ...
随机推荐
- SQL 计算某月有多少天
今天用SQL Server 2005写查询语句,要求计算一个月平均每天发生的金额.以前往往喜欢查询相关的所有列,在代码中进行计算,还没有在SQL中写过. 第一印象就是:要考虑到润年还是平年,再判断是大 ...
- POSIX 消息队列 之 概述 链接方式
NAMEmq_overview —— POSIX消息队列概述 DESCRIPTIONPOSIX消息队列允许进程以消息的形式交换数据.此API与System V消息队列(msgget(2),msgsnd ...
- iview-admin打包笔记
在程序根目录右键打开cmd,输入 npm run build 这时就会进入打包的步骤,打包好之后就会在程序根目录出现一个dist的文件夹,里面有一个index.html文件和另一个dist的文件夹. ...
- R语言学习——欧拉计划(3)Largest prime factor 求最大质因数
The prime factors of 13195 are 5, 7, 13 and 29. What is the largest prime factor of the number 60085 ...
- Floyd最短路径算法(来自微信公众号“算法爱好者”改编)
暑假,小哼准备去一些城市旅游.有些城市之间有公路,有些城市之间则没有,如下图.为了节省经费以及方便计划旅程,小哼希望在出发之前知道任意两个城市之前的最短路程. 上图中有4个城市8条公路,公路上的数字表 ...
- ESXI5.5设置主机的时间自动同步服务 NTP
背景:现在公司的很多线上服务也都通过虚拟化来实现,最近遇到一个小问题,虚拟机上的时间不准确.原来是虚拟机会主动同步宿主机时间,一般虚拟机中都安装vmware tool工具,这个工具会自动和宿主机进行时 ...
- 哈希与位图(Hash and BitMap)
Hash:哈希机制 BitMap:位图机制 目的:都是为了保证检索方便而设置的数据结构 对于大数据进行排序,由于内存限制,不可能在内存中进行,所以采取BitMap机制 为了在大数据中快速检索以及操作数 ...
- dos批处理文件中的变量小结
很多情况下我们需要用到一些批处理文件中的变量,方便我们执行一些操作,这里简单整理下,方便需要的朋友 批处理中的变量,我把他分为两类,分别为"系统变量"和"自定义变量& ...
- jieba库及wordcloud库的使用
知识内容: 1.jieba库的使用 2.wordcloud库的使用 参考资料: https://github.com/fxsjy/jieba https://blog.csdn.net/fontthr ...
- 暴搜 - Codeforces Round #327 (Div. 2) E. Three States
E. Three States Problem's Link Mean: 在一个N*M的方格内,有五种字符:'1','2','3','.','#'. 现在要你在'.'的地方修路,使得至少存在一个块'1 ...
