xgboost-Python&R
python机器学习-乳腺癌细胞挖掘(博主亲自录制视频)
https://study.163.com/course/introduction.htm?courseId=1005269003&utm_campaign=commission&utm_source=cp-400000000398149&utm_medium=share
xgboost官网教程
https://xgboost.readthedocs.io/en/latest/python/python_intro.html
下载地址
https://www.lfd.uci.edu/~gohlke/pythonlibs/#xgboost

然后打开cmd,用cd切换路径到下载地址
最后 pip install.....
乳腺癌数据测试代码
# -*- coding: utf-8 -*-
"""
Created on Thu Apr 19 21:04:14 2018 @author: Administrator
""" from sklearn.cross_validation import train_test_split
from sklearn.datasets import load_breast_cancer
cancer=load_breast_cancer()
train_x, test_x, train_y, test_y=train_test_split(cancer.data,cancer.target,random_state=0) import xgboost as xgb
dtrain=xgb.DMatrix(train_x,label=train_y)
dtest=xgb.DMatrix(test_x) params={'booster':'gbtree',
'objective': 'binary:logistic',
'eval_metric': 'auc',
'max_depth':4,
'lambda':10,
'subsample':0.75,
'colsample_bytree':0.75,
'min_child_weight':2,
'eta': 0.025,
'seed':0,
'nthread':8,
'silent':1} watchlist = [(dtrain,'train')] bst=xgb.train(params,dtrain,num_boost_round=100,evals=watchlist) ypred=bst.predict(dtest) # 设置阈值, 输出一些评价指标
y_pred = (ypred >= 0.5)*1 #模型校验
from sklearn import metrics
print ('AUC: %.4f' % metrics.roc_auc_score(test_y,ypred))
print ('ACC: %.4f' % metrics.accuracy_score(test_y,y_pred))
print ('Recall: %.4f' % metrics.recall_score(test_y,y_pred))
print ('F1-score: %.4f' %metrics.f1_score(test_y,y_pred))
print ('Precesion: %.4f' %metrics.precision_score(test_y,y_pred))
metrics.confusion_matrix(test_y,y_pred) '''
模型区分能力相当好
AUC: 0.9981
ACC: 0.9860
Recall: 0.9889
F1-score: 0.9889
Precesion: 0.9889 ''' print("xgboost:")
#print("accuracy on the training subset:{:.3f}".format(bst.get_score(train_x,train_y)))
#print("accuracy on the test subset:{:.3f}".format(bst.get_score(test_x,test_y)))
print('Feature importances:{}'.format(bst.get_fscore()))
'''
Feature importances:{'f20': 33, 'f27': 50, 'f21': 54, 'f1': 29, 'f7': 33, 'f22': 38,
'f26': 17, 'f13': 46, 'f23': 41, 'f24': 13, 'f15': 2, 'f0': 6, 'f14': 5, 'f25': 7,
'f3': 6, 'f12': 3, 'f9': 3, 'f28': 11, 'f8': 2, 'f10': 9, 'f6': 9, 'f16': 2, 'f29': 1,
'f4': 4, 'f18': 3, 'f19': 2, 'f17': 2, 'f11': 1}
'''
'''
import matplotlib.pylab as plt
import pandas as pd
feat_imp = pd.Series(alg.booster().get_fscore()).sort_values(ascending=False)
feat_imp.plot(kind='bar', title='Feature Importances')
plt.ylabel('Feature Importance Score')
'''
#preds = bst.predict(test_x)
乳腺癌数据测试代码2
# -*- coding: utf-8 -*-
"""
Created on Sun Jun 17 10:10:45 2018 @author: Administrator
"""
import xgboost as xgb
from sklearn.cross_validation import train_test_split
from sklearn.datasets import load_breast_cancer
#gridSearchCv调参
from sklearn.grid_search import GridSearchCV
import pickle
import numpy as np
import graphviz cancer=load_breast_cancer()
train_x, test_x, train_y, test_y=train_test_split(cancer.data,cancer.target,random_state=0)
x=cancer.data
y=cancer.target #加载数据
#XGBoost可以加载libsvm格式的文本数据,加载的数据格式可以为Numpy的二维数组和XGBoost的二进制的缓存文件。加载的数据存储在对象DMatrix中
dtrain=xgb.DMatrix(train_x,label=train_y)
dtest=xgb.DMatrix(test_x) params={'booster':'gbtree',
'objective': 'binary:logistic',
#'eval_metric': 'auc',
'max_depth':4,
'lambda':10,
'subsample':0.75,
'colsample_bytree':0.75,
'min_child_weight':2,
'eta': 0.025,
'seed':0,
'nthread':8,
'silent':1} params['eval_metric'] = ['auc', 'ams@0']
#params['eta']=list(np.arange(0,1,0.1))
#Specify validations set to watch performance
evallist=[(dtrain, 'train')]
'''
eta_range=list(np.arange(0,1,0.1))
#参数格子
param_grid=dict(eta=eta_range)
''' #num_boost_round这是指提升迭代的个数
#evals 这是一个列表,用于对训练过程中进行评估列表中的元素。形式是evals = [(dtrain,’train’),(dval,’val’)]或者是evals = [(dtrain,’train’)],对于第一种情况,它使得我们可以在训练过程中观察验证集的效果。
bst=xgb.train(params,dtrain,num_boost_round=100,evals=evallist) ypred=bst.predict(dtest)
# 设置阈值, 输出一些评价指标
y_pred = (ypred >= 0.5)*1 from sklearn import metrics
print ('AUC: %.4f' % metrics.roc_auc_score(test_y,ypred))
print ('ACC: %.4f' % metrics.accuracy_score(test_y,y_pred))
print ('Recall: %.4f' % metrics.recall_score(test_y,y_pred))
print ('F1-score: %.4f' %metrics.f1_score(test_y,y_pred))
print ('Precesion: %.4f' %metrics.precision_score(test_y,y_pred))
metrics.confusion_matrix(test_y,y_pred) #绘制重要性特征
xgb.plot_importance(bst)
#绘制树
#xgb.plot_tree(bst, num_trees=2) #保存模型
bst.save_model('xgboost.model')
#The model and its feature map can also be dumped to a text file.
# dump model
bst.dump_model('dump.raw.txt')
# dump model with feature map
bst.dump_model('dump.raw.txt', 'featmap.txt') #保存分类器
save_classifier = open("xgboost.pickle","wb")
pickle.dump(bst, save_classifier)
save_classifier.close() '''
#打开分类器文件测试
classifier_f = open("xgboost.pickle", "rb")
classifier = pickle.load(classifier_f)
classifier_f .close()
'''
xgboost-feature importance
# -*- coding: utf-8 -*-
"""
Created on Sun Jun 17 10:34:20 2018 @author: Administrator
""" import pandas as pd
import xgboost as xgb
import operator
from matplotlib import pylab as plt def ceate_feature_map(features):
outfile = open('xgb.fmap', 'w')
i = 0
for feat in features:
outfile.write('{0}\t{1}\tq\n'.format(i, feat))
i = i + 1 outfile.close() def get_data():
train = pd.read_csv("../input/train.csv") features = list(train.columns[2:]) y_train = train.Hazard for feat in train.select_dtypes(include=['object']).columns:
m = train.groupby([feat])['Hazard'].mean()
train[feat].replace(m,inplace=True) x_train = train[features] return features, x_train, y_train def get_data2():
from sklearn.datasets import load_iris
#获取数据
iris = load_iris()
x_train=pd.DataFrame(iris.data)
features=["sepal_length","sepal_width","petal_length","petal_width"]
x_train.columns=features
y_train=pd.DataFrame(iris.target)
return features, x_train, y_train #features, x_train, y_train = get_data()
features, x_train, y_train = get_data2()
ceate_feature_map(features) xgb_params = {"objective": "reg:linear", "eta": 0.01, "max_depth": 8, "seed": 42, "silent": 1}
num_rounds = 1000 dtrain = xgb.DMatrix(x_train, label=y_train)
xgb_model = xgb.train(xgb_params, dtrain, num_rounds) importance = xgb_model.get_fscore(fmap='xgb.fmap')
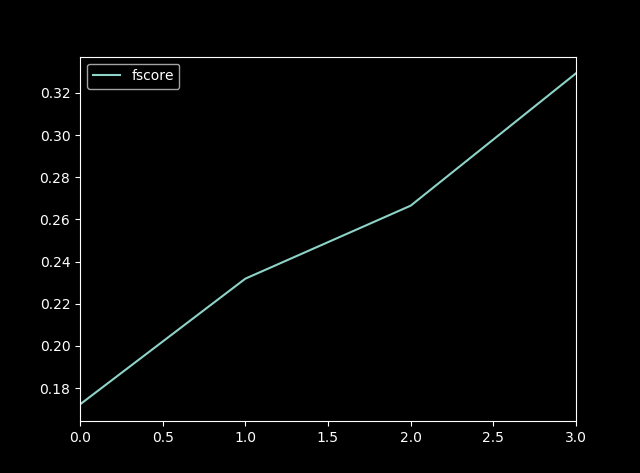
importance = sorted(importance.items(), key=operator.itemgetter(1)) df = pd.DataFrame(importance, columns=['feature', 'fscore'])
df['fscore'] = df['fscore'] / df['fscore'].sum() plt.figure()
df.plot()
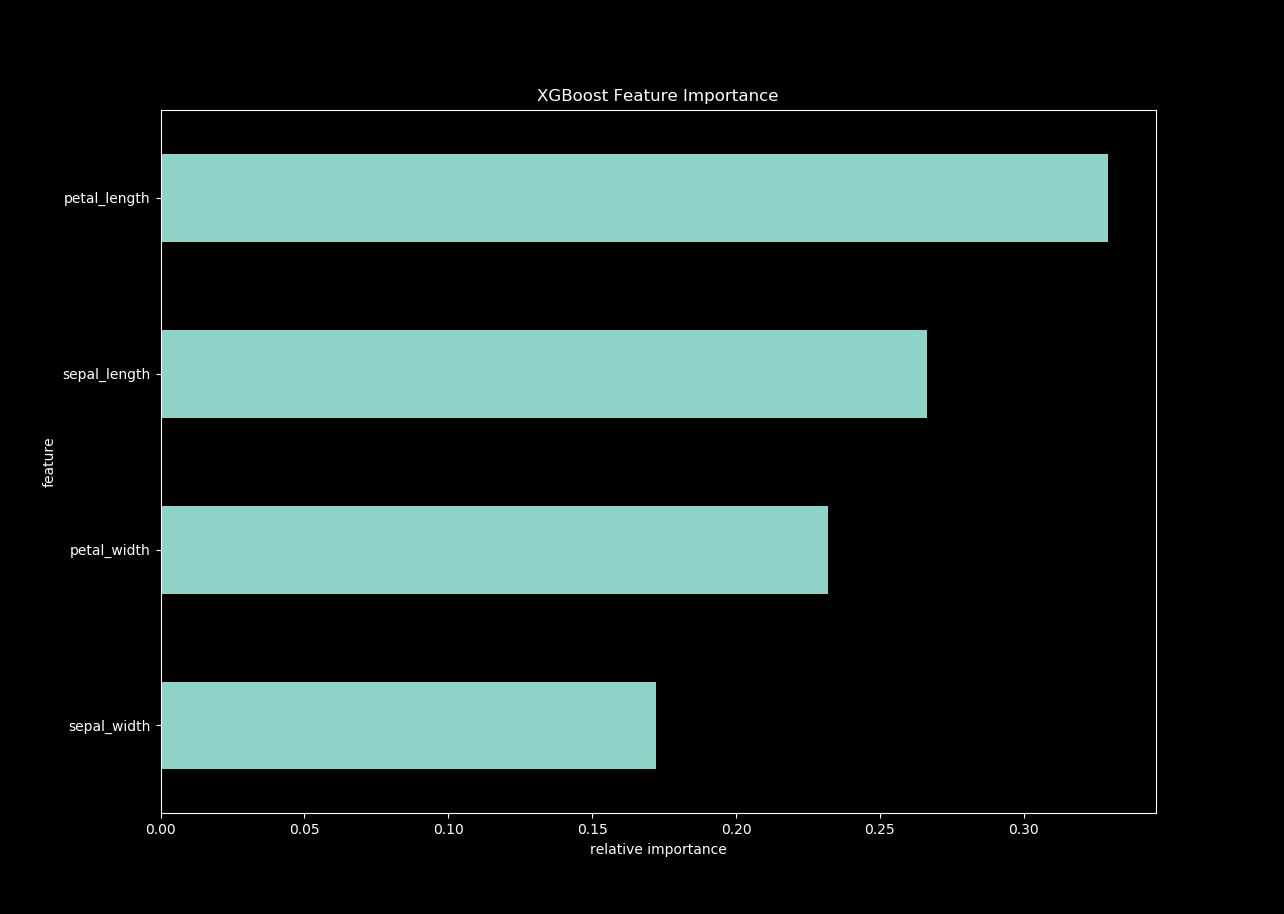
df.plot(kind='barh', x='feature', y='fscore', legend=False, figsize=(16, 10))
plt.title('XGBoost Feature Importance')
plt.xlabel('relative importance')
plt.gcf().savefig('feature_importance_xgb.png')
xgboost R语言函数索引:
agaricus.test Test part from Mushroom Data Set
agaricus.train Training part from Mushroom Data Set
callbacks Callback closures for booster training.
cb.cv.predict Callback closure for returning cross-validation
based predictions.
cb.early.stop Callback closure to activate the early
stopping.
cb.evaluation.log Callback closure for logging the evaluation
history
cb.gblinear.history Callback closure for collecting the model
coefficients history of a gblinear booster
during its training.
cb.print.evaluation Callback closure for printing the result of
evaluation
cb.reset.parameters Callback closure for restetting the booster's
parameters at each iteration.
cb.save.model Callback closure for saving a model file.
dim.xgb.DMatrix Dimensions of xgb.DMatrix
dimnames.xgb.DMatrix Handling of column names of 'xgb.DMatrix'
getinfo Get information of an xgb.DMatrix object
predict.xgb.Booster Predict method for eXtreme Gradient Boosting
model
print.xgb.Booster Print xgb.Booster
print.xgb.DMatrix Print xgb.DMatrix
print.xgb.cv.synchronous
Print xgb.cv result
setinfo Set information of an xgb.DMatrix object
slice Get a new DMatrix containing the specified rows
of orginal xgb.DMatrix object
xgb.Booster.complete Restore missing parts of an incomplete
xgb.Booster object.
xgb.DMatrix Construct xgb.DMatrix object
xgb.DMatrix.save Save xgb.DMatrix object to binary file
xgb.attr Accessors for serializable attributes of a
model.
xgb.create.features Create new features from a previously learned
model
xgb.cv Cross Validation
xgb.dump Dump an xgboost model in text format.
xgb.gblinear.history Extract gblinear coefficients history.
xgb.ggplot.deepness Plot model trees deepness
xgb.ggplot.importance Plot feature importance as a bar graph
xgb.importance Importance of features in a model.
xgb.load Load xgboost model from binary file
xgb.model.dt.tree Parse a boosted tree model text dump
xgb.parameters<- Accessors for model parameters.
xgb.plot.multi.trees Project all trees on one tree and plot it
xgb.plot.shap SHAP contribution dependency plots
xgb.plot.tree Plot a boosted tree model
xgb.save Save xgboost model to binary file
xgb.save.raw Save xgboost model to R's raw vector, user can
call xgb.load to load the model back from raw
vector
xgb.train eXtreme Gradient Boosting Training
xgboost-deprecated Deprecation notices.
可以在目录‘C:/Users/zhi.li04/Documents/R/win-library/3.3/xgboost/doc’中的小文品内找到更多的信息
discoverYourData: Discover your data (source, pdf)
xgboostPresentation: Xgboost presentation (source, pdf)
xgboost: xgboost: eXtreme Gradient Boosting (source, pdf)
xgboost调参
https://blog.csdn.net/wdxin1322/article/details/71698659?utm_source=itdadao&utm_medium=referral
具体参数树状图:
eta:默认值设置为0.3。您需要指定用于更新步长收缩来防止过度拟合。每个提升步骤后,我们可以直接获得新特性的权重。实际上 eta 收缩特征权重的提高过程更为保守。范围是0到1。低η值意味着模型过度拟合更健壮。
gamma:默认值设置为0。您需要指定最小损失减少应进一步划分树的叶节点。更大,更保守的算法。范围是0到∞。γ越大算法越保守。
max_depth:默认值设置为6。您需要指定一个树的最大深度。参数范围是1到∞。
min_child_weight:默认值设置为1。您需要在子树中指定最小的(海塞)实例权重的和,然后这个构建过程将放弃进一步的分割。在线性回归模式中,在每个节点最少所需实例数量将简单的同时部署。更大,更保守的算法。参数范围是0到∞。
max_delta_step:默认值设置为0。max_delta_step 允许我们估计每棵树的权重。如果该值设置为0,这意味着没有约束。如果它被设置为一个正值,它可以帮助更新步骤更为保守。通常不需要此参数,但是在逻辑回归中当分类是极为不均衡时需要用到。将其设置为1 - 10的价值可能有助于控制更新。参数范围是0到∞。
subsample: 默认值设置为1。您需要指定训练实例的子样品比。设置为0.5意味着XGBoost随机收集一半的数据实例来生成树来防止过度拟合。参数范围是0到1。
colsample_bytree : 默认值设置为1。在构建每棵树时,您需要指定列的子样品比。范围是0到1。
colsample_bylevel:默认为1
max_leaf_nodes:叶结点最大数量,默认为2^6
线性上升具体参数
lambda and alpha : L2正则化项,默认为1、L1正则化项,默认为1。这些都是正则化项权重。λ默认值假设是1和α= 0。
lambda_bias : L2正则化项在偏差上的默认值为0。
scale_pos_weight:加快收敛速度,默认为1
任务参数
base_score : 默认值设置为0.5。您需要指定初始预测分数作为全局偏差。
objective : 默认值设置为reg:linear。您需要指定你想要的类型的学习者,包括线性回归、逻辑回归、泊松回归等。
eval_metric : 您需要指定验证数据的评估指标,一个默认的指标分配根据客观(rmse回归,错误分类,意味着平均精度等级
seed : 随机数种子,确保重现数据相同的输出。
调参方式
- 首先调整max_depth ,通常max_depth 这个参数与其他参数关系不大,初始值设置为10,找到一个最好的误差值,然后就可以调整参数与这个误差值进行对比。比如调整到8,如果此时最好的误差变高了,那么下次就调整到12;如果调整到12,误差值比10 的低,那么下次可以尝试调整到15.
- 在找到了最优的max_depth之后,可以开始调整subsample,初始值设置为1,然后调整到0.8 如果误差值变高,下次就调整到0.9,如果还是变高,就保持为1.0
- 接着开始调整min_child_weight , 方法与上面同理
- 再接着调整colsample_bytree
- 经过上面的调整,已经得到了一组参数,这时调整eta 到0.05,然后让程序运行来得到一个最佳的num_round,(在 误差值开始上升趋势的时候为最佳 )
# General Parameters, see comment for each definition
2 # choose the booster, can be gbtree or gblinear
3 booster = gbtree
4 # choose logistic regression loss function for binary classification
5 objective = binary:logistic
6
7 # Tree Booster Parameters
8 # step size shrinkage
9 eta = 1.0
10 # minimum loss reduction required to make a further partition
11 gamma = 1.0
12 # minimum sum of instance weight(hessian) needed in a child
13 min_child_weight = 100
14 # maximum depth of a tree
15 max_depth = 6
16
17 # Task Parameters
18 # the number of round to do boosting
19 num_round = 50
20 # 0 means do not save any model except the final round model
21 save_period = 0
22 # The path of training data
23 data = "a.train"
24 # The path of validation data, used to monitor training process, here [test] sets name of the validation set
25 eval[test] = "a.test"
26 # evaluate on training data as well each round
27 #eval_train = 1
28 # The path of test data
29 eval_metric = "auc"
30 eval_metric = "error"
31 test:data = "a.test"



微信扫二维码,免费学习更多python资源

xgboost-Python&R的更多相关文章
- VS code 配置为 Python R LaTeX IDE
VS code配置为Python R LaTeX IDE VS code的中文断行.编辑功能强大,配置简单. VSC的扩展在应用商店搜索安装,快捷键ctrl+shift+x调出应用商店. 安装扩展后, ...
- 配置 Sublime Text 3 作为Python R LaTeX Markdown IDE
配置 Sublime Text 3 作为Python R LaTeX Markdown IDE 配置 Sublime Text 3 作为Python IDE IDE的基本功能:代码提醒.补全:编译文件 ...
- 机器学习算法 Python&R 速查表
sklearn实战-乳腺癌细胞数据挖掘( 博主亲自录制) https://study.163.com/course/introduction.htm?courseId=1005269003&u ...
- ubuntu 执行Python脚本出现: /usr/bin/env: ‘python\r’: No such file or directory
原因: #!/usr/bin/env python 在ubuntu会变成 #!/usr/bin/env python\r 而\r 会被shell 当成参数 所以出现: /usr/bin/env: ‘ ...
- 建模分析之机器学习算法(附python&R代码)
0序 随着移动互联和大数据的拓展越发觉得算法以及模型在设计和开发中的重要性.不管是现在接触比较多的安全产品还是大互联网公司经常提到的人工智能产品(甚至人类2045的的智能拐点时代).都基于算法及建模来 ...
- python '%r'或者'{!r}'的意思
转载:https://blog.csdn.net/a19990412/article/details/80149112 这两个都是python的转译字符, 类似于%r, %d,%f >>& ...
- Python&R&量化 金融之路
[ 分类 ]- 金融之路 - 闲云孤鹤(人生在世五十年,大千世界一瞬间,浮生若梦,仿佛间,幻境一场,生者无常,终须尽.) - CSDN博客 https://blog.csdn.net/robertso ...
- kNN算法及其python&R实现
iris数据集,这一教科书级别的数据,分类前不需要做任何数据预处理什么的,相当的理想!但请注意你自己的数据99%的可能需要做预处理. 下面分别用R语言和Python来实现iris数据集的分类: R语言 ...
- xgboost python windows编译问题
1.作为kaggle上非常火的机器学习包xgboost,windows python包安装起来真的非常麻烦,安装了一整天才成功. 2.请先下载xgboost-master,csdn上有资源的,能够在这 ...
- 分析脚本搭建docker环境:python, R
1. 搭建Anaconda Python3.6 FROM nvidia/cuda:8.0-cudnn6-devel-ubuntu16.04 MAINTAINER Tyan <tyan.liu.g ...
随机推荐
- mac git从代码仓库克隆代码,修改并上传
1:添加本地秘钥到代码仓库中 open ~/ .ssh 以github为例: mac 命令行输入open ~/ .ssh,打开id_rsa.pub文件中的内容,复制到github->settin ...
- MFC俄罗斯方块
经典俄罗斯方块游戏 源码百度云链接 链接:https://pan.baidu.com/s/14frk2EuFoiRCzudol2Xgvg提取码:syzk GitHub https://github.c ...
- 前端 聊聊Ajax
转载:关于Ajax的全面总结.
- JavaScript调试总结
一.vs调试 1.设置断点 2.设置浏览模式:一定要设置IE浏览器 3.设置起始页 4.运行 二.谷歌浏览器调试 1.运行项目,进入后F12,找到Sources
- 数论 C - Aladdin and the Flying Carpet
It's said that Aladdin had to solve seven mysteries before getting the Magical Lamp which summons a ...
- 深入研究 Mini ASP.NET Core(迷你 ASP.NET Core),看看 ASP.NET Core 内部到底是如何运行的
前言 几年前,Artech 老师写过一个 Mini MVC,用简单的代码告诉读者 ASP.NET MVC 内部到底是如何运行的.当时我研究完以后,受益匪浅,内心充满了对 Artech 老师的感激,然后 ...
- [JSOI2008]Blue Mary的旅行
嘟嘟嘟 看\(n\)那么小,就知道是网络流.然后二分,按时间拆点. 刚开始我看成所有航班一天只能起飞一次,纠结了好一会儿.但实际上是每一个航班单独考虑,互不影响. 建图很显然,拆完点后每一个点的第\( ...
- gdb cheat sheet
0x01 控制流 r run,运行程序. r < a.txt run,重定向输入 si step instruction 进入函数 ni next instruction 下一 ...
- 【alpha阶段】第十次Scrum Meeting
每日任务内容 队员 昨日完成任务 明日要完成的任务 牛宇航 #26 评价总览接口编写https://github.com/rRetr0Git/rateMyCourse/issues/26 alpha阶 ...
- ModuleNotFoundError: No module named 'redis'
在安装过Redis后,通过Python程序导入redis时,遇到一个“ModuleNotFoundError: No module named redis”错误,网上查了下原因,解决办法如下: Pyt ...