GO 功能注释
文章转载于 Original 2017-06-12 liuhui 生信百科
相似的基因在不同物种中,其功能往往保守的。显然,需要一个统一的术语用于描述这些跨物种的同源基因及其基因产物的功能,否则,不同的实验室对相同的基因的功能的描述不同,将极大限制学术的交流。而 Gene Ontology (GO) 项目正是为了能够使对各种数据库中基因获基因产物功能描述相一致的努力结果。
所谓的 GO,是生物学功能注释的一个标准词汇表术语(GO term),将基因的功能分为三部分:
基因执行的分子功能(Molecular Function)
基因所处的细胞组分(Cellular Component)
基因以及参与的生物学过程(Biological Process)
不同的 GO term 通过有向无环图关联起来,如下图所示:
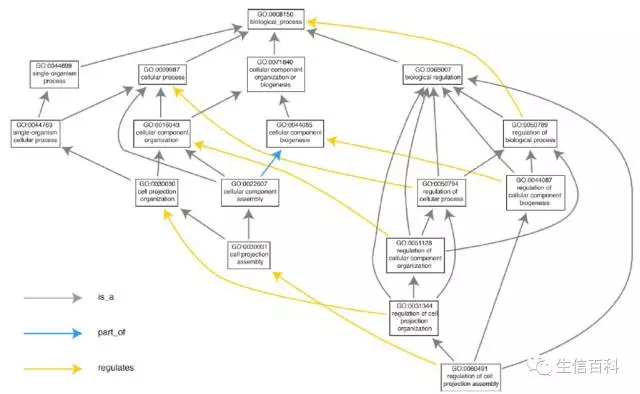
可以看出,不同的 GO term 间的关系由三类:is_a、part_of 和 regulates。
如 regulation of cell projection assembly 是一种生物学过程,是 regulation of cell projection organization 中的一类(is_a),还调节(regulates)cell projection assembly;又如 cellular component assembly 是 celluar component biogenesis 的一部分(part_of)。值得注意的是,这些关系都是有方向的,即反过来不成立,因而叫做有向无环图。
目前,GO 注释主要有两种方法:
(1)序列相似性比对(BLAST)
(2)结构域相似性比对(InterProScan)
这里以序列相似性比对为例,简单介绍 GO 注释的步骤:
将基因序列与 swiss-prot 蛋白质数据库进行 BLAST (blastp 或者 blastx,这篇文章介绍了如何做 BLAST 分析:BLAST 知多少?)比对,得到如下结果:
c49_g1_i1 RNF13_MOUSE 52.00 50 23 1 17 166 240 288 2e-11 65.5
c72_g1_i1 RS25_NEUCR 78.72 94 20 0 375 94 1 94 1e-32 116
c75_g1_i1 POLX_TOBAC 45.28 53 29 0 162 4 457 509 1e-08 55.1
c86_g2_i1 POLX_TOBAC 46.43 112 60 0 339 4 879 990 2e-30 120
c91_g1_i1 BUB1_ARATH 55.71 70 28 2 61 264 289 357 1e-14 73.6
c143_g1_i1 STL1_YEAST 31.98 172 85 4 6 518 407 547 6e-17 82.8
c150_g1_i1 CST26_YEAST 37.63 93 38 3 223 5 142 234 6e-10 58.2
c150_g2_i1 YHOE_SCHPO 42.67 75 41 1 227 3 54 126 5e-16 74.7
c156_g2_i1 EXOL2_ARATH 47.17 53 28 0 299 141 229 281 6e-06 47.0
c169_g1_i1 SPT5_ASPFU 60.98 82 31 1 20 262 725 806 2e-18 84.0
其中,第二列 swiss-prot 蛋白质数据库序列的 ID(UniProtKB ID)。
从 ftp://ftp.pir.georgetown.edu/databases/idmapping 下载 idmapping.tb.gz,该文件共有 22 列(tab 键分割):
Q6GZX4 001R_FRG3G 2947773 YP_031579.1 81941549; 49237298 PF04947 GO:0006355; GO:0046782; GO:0006351 UniRef100_Q6GZX4 UniRef90_Q6GZX4 UniRef50_Q6GZX4 UPI00003B0FD4 654924 15165820 AY548484 AAT09660.1
每一列的含义分别为 (可以看出,许多数据库已经和GO关联了):
1. UniProtKB accession
2. UniProtKB ID
3. EntrezGene
4. RefSeq
5. NCBI GI number
6. PDB
7. Pfam
8. GO
9. PIRSF
10. IPI
11. UniRef100
12. UniRef90
13. UniRef50
14. UniParc
15. PIR-PSD accession
16. NCBI taxonomy
17. MIM
18. UniGene
19. Ensembl
20. PubMed ID
21. EMBL/GenBank/DDBJ
22. EMBL protein_id
根据文件 idmapping.tb.gz,将 blast 的结果,通过 UniProtKB ID,将第八列的 GO 号注释到对应的基因上
python UniProt2GO_annotate.py idmapping.tb.gz blastout outputfile
结果如下:
c93619_g2_i1 GO:0005506,GO:0016705,GO:0016021,GO:0004497,GO:0020037
c93619_g2_i3 GO:0009733,GO:0020037,GO:0044550,GO:0016021,GO:0016020,GO:0016711,GO:0009813,GO:0005789,GO:0005506
c70056_g1_i1 GO:0005737,GO:0019722,GO:0071889,GO:0005829,GO:0001077,GO:0006357,GO:0097720,GO:0000978,GO:0046872,GO:0005634,GO:0006874
c93748_g1_i1 GO:0006729,GO:0008124
c107639_g1_i1 GO:0009737,GO:0009738,GO:0005623,GO:0006970,GO:0009651,GO:0045454,GO:0009789
c106424_g1_i1 GO:0043565,GO:0009555,GO:0003700,GO:0005634,GO:0009793,GO:0006351
c66585_g1_i1 GO:0005737,GO:0003746,GO:0003924,GO:0005525
c110618_g1_i8 GO:0015297,GO:0016021,GO:0015238
c105249_g1_i5 GO:0046872,GO:0043161,GO:0005829,GO:0006915,GO:0032648,GO:0050691,GO:0005654,GO:0070936,GO:0061630,GO:0005634
c134727_g1_i1 GO:0072546,GO:0030246,GO:0005783
拓展阅读:
Ontology Relations:http://geneontology.org/page/ontology-relations#basics
Frequently Asked Questions (FAQ):http://geneontology.org/faq-page
脚本 UniProt2GO_annotate.py 下载:
链接:http://pan.baidu.com/s/1kVjzJYv 密码:vigu
GO 功能注释的更多相关文章
- SNP功能注释网站合集
这篇文章是对SNP位点功能注释在线网站的一个总结帖. 软件排名不分先后,优先顺序可以看推荐指数. 彩蛋在最后,请坚持看完 1.GWAS4D, 推荐指数:**** 网址:http://mulinlab. ...
- Journal of Proteome Research | SAAVpedia: identification, functional annotation, and retrieval of single amino acid variants for proteogenomic interpretation | SAAV的识别、功能注释和检索 | (解读人:徐洪凯)
文献名:SAAVpedia: identification, functional annotation, and retrieval of single amino acid variants fo ...
- GO 和 KEGG 的区别 | GO KEGG数据库用法 | 基因集功能注释 | 代谢通路富集
一直都搞不清楚这两者的具体区别. 其实初学者搞不清楚很正常,因为它们的本质是相通的,都是对基因进行归类注释的数据库. 建议初学者自己使用一下这两个数据库,应该很快就能明白其中的区别. (抱歉之前没讲清 ...
- [Servlet3.0新功能]注释替代配置文件
作者信息 作者名称:金云龙 个人网站:http://www.longestory.com 个人公众帐号:搜索"longestory"或"龙哥有话说" 须要注意的 ...
- COG注释--转载
http://blog.sina.com.cn/s/blog_670445240102uxwy.html 一 COG简介 COG,即Clusters of Orthologous Groups of ...
- 让Jayrock插上翅膀(加入输入输出参数注释,测试页面有注释,下拉框可以搜索)
继上一篇文章介绍了Jayrock组件开发接口的具体步骤和优缺点之后,今天给大家带来的就是,如何修复这些缺点. 首先来回顾一下修复的缺点有哪些: 1.每个接口的只能写大概的注释,不能分开来写,如接口的主 ...
- WebService开发常用功能详解
一.WebService中常用的属性(Attributes)1. Web Service(Web服务)提供以下三个属性. Namespace:此属性的值包含 XML Web Service的默认 ...
- JAVA注释方式--目前用的
代码整洁,规范,可读,注释是关键之一. 1.整个类文件注释 注释结构:/* * @(#){类名称}.java {创建时间} * * {某人或某公司具有完全的版权} * {使用者必须经过许可 ...
- GO | KEGG的注释是怎么来的?
但凡是做过基因表达数据分析的(芯片.RNA-seq,scRNA-seq),肯定是跑过基因集功能注释和通路富集的,因为它是研究未知基因集的利器. 但跑过之后老板肯定会给反馈,通常得到的注释都是没有太多意 ...
随机推荐
- JSON Schema(模式)
JSON Schema指的是数据交换中的一种虚拟的“合同”. JSON验证器负责验证语法错误,JSON Schema负责提供一致性检验. JSON Schema是数据接收方额第一道防线,也是数据发送方 ...
- 【Spring实战】Spring容器初始化完成后执行初始化数据方法
一.背景知识及需求 在做WEB项目时,经常在项目第一次启动时利用WEB容器的监听.Servlet加载初始化等切入点为数据库准备数据,这些初始化数据是系统开始运行前必须的数据,例如权限组.系统选项.默认 ...
- IOS开发 GCD介绍: 基本概念和Dispatch Queue
iOS的三种多线程技术 1.NSThread 每个NSThread对象对应一个线程,量级较轻(真正的多线程) 2.以下两点是苹果专门开发的“并发”技术,使得程序员可以不再去关心线程的具体使用问题 ØN ...
- [QT]数据库SQLITE使用错误记录
1.仿照创建数据库的例程编写代码,出现以下问题: ① 创建QSqlQuery query; 注意:这里没有将 query 与 QSqlSatabase db, 关联,正确的应该是 : QSqlQu ...
- 正确的使用margin:0 auto与body{text-align:center;}实现元素居中(转)
body{text-align:center}与margin:0 auto的异同? text-align是用于设置或对象中文本的对齐方式.一般情况下我们设置文本对齐方式的时候需要用此属性进行设置 我们 ...
- 快速排序的partition版本实现
int partition(int arr[], int low, int high) { int pivot = arr[high]; int i = low-1; for (int j = low ...
- UVALive - 4108 SKYLINE (吉司机线段树)
题目链接 题意:在一条直线上依次建造n座建筑物,每座建筑物建造完成后询问它在多长的部分是最高的. 比较好想的方法是用线段树分别维护每个区间的最小值mi和最大值mx,当建造一座高度为x的建筑物时,若mi ...
- BZOJ4975: [Lydsy1708月赛]区间翻转( 博弈&逆序对)
4975: [Lydsy1708月赛]区间翻转 Time Limit: 1 Sec Memory Limit: 256 MBSubmit: 265 Solved: 140[Submit][Stat ...
- java并发--Callable、Future和FutureTask
在前面的文章中我们讲述了创建线程的2种方式,一种是直接继承Thread,另外一种就是实现Runnable接口. 这2种方式都有一个缺陷就是:在执行完任务之后无法获取执行结果. 如果需要获取执行结果,就 ...
- BZOJ2555 SubString【后缀自动机+LCT】
Description 懒得写背景了,给你一个字符串init,要求你支持两个操作 (1):在当前字符串的后面插入一个字符串 (2):询问字符串s在当前字符串中出现了几次?(作为连续子串) 你必须在线支 ...
