利用ONT测序检测真核生物全基因组甲基化状态
摘要
甲基化在真核生物基因组序列中广泛存在,其中5mC最为普遍,在真核生物基因组中也有发现6mA。捕获基因组中的甲基化状态的常用技术是全基因组甲基化测序(WGBS)和简化甲基化测序(RRBS),而随着第三代测序技术的完善,ONT单分子纳米孔测序可以从单分子的角度来检出甲基化的胞嘧啶和腺嘌呤电流的变化,从而实现由基因组中的一段序列中检出5mC和6mA,然而精确地从单碱基级别检出5mC和6mA扔具有挑战。本文利用第三代ONT测序技术获得的序列及其电信号来检出真核生物全基因组范围的5mC和6mA甲基化状态。
背景
DNA甲基化主要发生在脱氧核糖核苷酸的第五位的胞嘧啶和第六位的腺嘌呤,前者普遍存在于真核生物,后者在原核生物中广泛存在,也有研究报道6mA存在于真核生物。这样的甲基化状态在ONT测序仪捕捉到的电流信号中,不仅单碱基的电流会发生改变,而且其上下文的一段基因组序列也会发生改变。基于此,一些生物信息学软件先后被开发出来针对于这两种甲基化的检出有各自的优缺点。有研究指出在真核基因组中检出5mC和6mA准确度较高的软件分别为nanopolish[1]和tombo[2]。
利用nanopolish检出真核生物基因组中5mC的甲基化位置
材料和方法
利用minION平台对目标生物血液提取的DNA不打断建库并进行全基因组测序,获得12G序列及其电信号文件。安装nanopolish(v0.13.2)。
步骤
- 建立索引
nanopolish index -d fast5_files/ output.fastq
- 比对
minimap2 -a -x map-ont reference.fasta output.fastq | samtools sort -T tmp -o output.sorted.bam
samtools index output.sorted.bam
- Calling methylation
nanopolish call-methylation --progress -q cpg -t NCPU --verbose -r reads.fastq -b output.sorted.bam -g reference_genome.fasta > nanopolish_call_methylation.tsv
- 筛选高置信度的甲基化和未甲基化位点
calculate_methylation_frequency.py[3]
nanopore-methylation-utilities/mtsv2bedGraph.py[4]
scripts/calculate_methylation_frequency.py -c 2 methylation_calls.tsv > methylation_frequency.tsv
# or
python nanopore-methylation-utilities/parseMethylbed.py frequency -i methylation_calls.tsv -o methylation_calls_freq.tsv --verbose -m cpg -u 2 -l -2
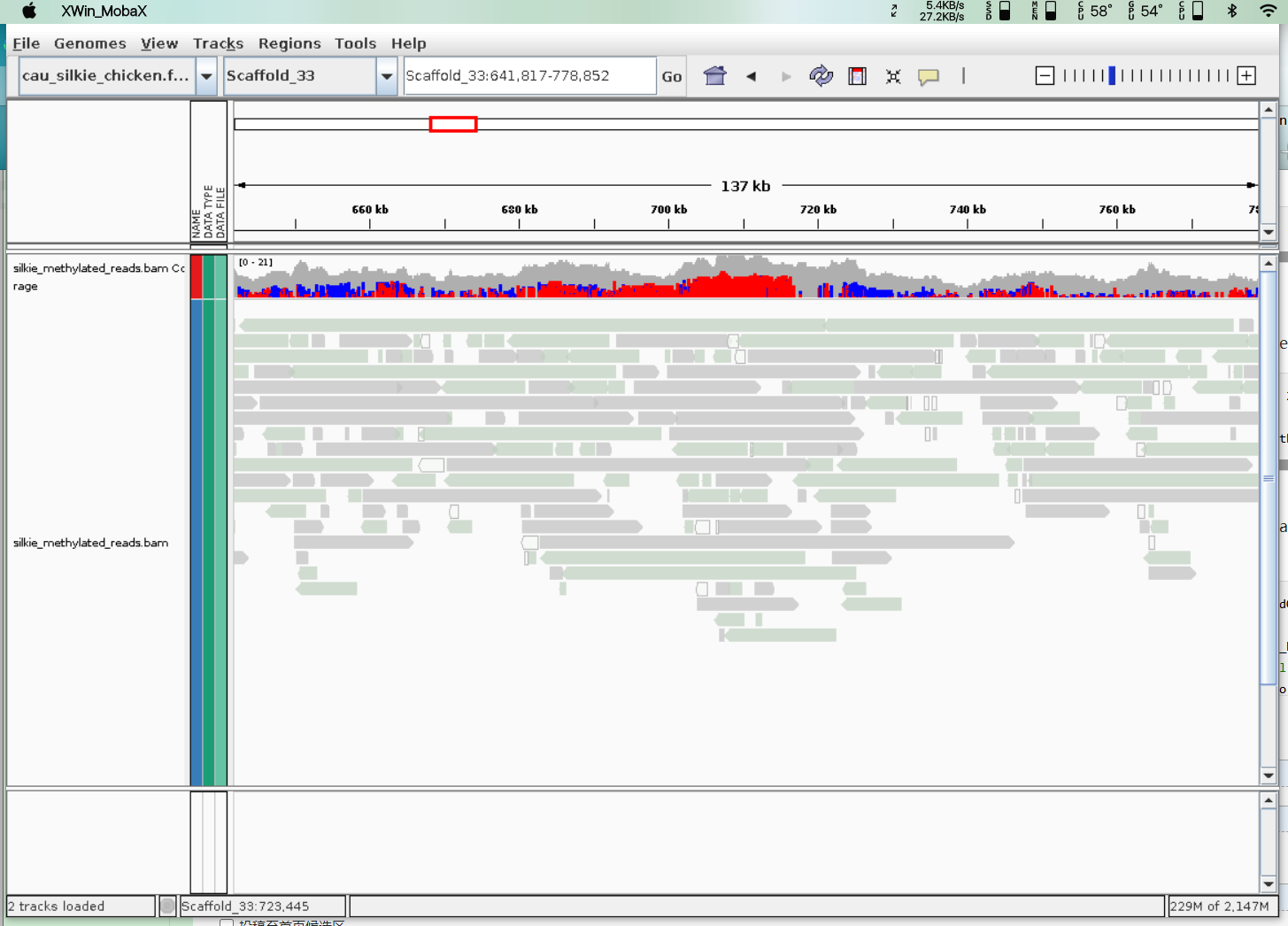
- IGV可视化或UCSC可视化
IGV可视化[5]
python nanopore-methylation-utilities/mtsv2bedGraph.py --verbose -c 2 -i methylation_calls.tsv -q cpg -g reference_genome.fasta | sort -k1,1 -k2,2n | bgzip > methylation_calls.bed.gz
tabix -p methylation_calls.bed.gz
python nanopore-methylation-utilities/convert_bam_for_methylation.py -t 100 --verbose --remove_poor -c methylation_calls.bed.gz -f reference_genome.fasta -b output.sorted.bam | samtools sort -o methylation_calls.bed.remove_no_or_poor_methylation_reads.bam
# if all reads' coverage was needed, remap all reads to reference to find out
samtools index methylation_calls.bed.remove_no_or_poor_methylation_reads.bam
# now bam file can be loaded to igv via their bisulfite mode to see methylation sites and unmethylation sites.
利用ONT测序检测真核生物全基因组甲基化状态的更多相关文章
- 全基因组测序 Whole Genome Sequencing
全基因组测序 Whole Genome Sequencing 全基因组测序(Whole Genome Sequencing,WGS)是利用高通量测序平台对一种生物的基因组中的全部基因进行测序,测定其 ...
- cfDNA(circulating cell free DNA)全基因组测序
参考资料: [cfDNA专题]cell-free DNA在非肿瘤疾病中的临床价值(好) ctDNA, cfDNA和CTCs有什么区别吗? cfDNA你懂多少? 新发现 | 基因是否表达,做个cfDNA ...
- 全基因组测序 从头测序(de novo sequencing) 重测序(re-sequencing)
全基因组测序 全基因组测序分为从头测序(de novo sequencing)和重测序(re-sequencing). 从头测序(de novo)不需要任何参考基因组信息即可对某个物种的基因组进行测序 ...
- PacBio全基因组测序和组装
PacBio公司的业务范围也就5个(官网): Whole Genome Sequencing Targeted Sequencing Complex Populations RNA Sequencin ...
- WGS 全基因组测序数据分析
1. DNA测序技术 https://www.jianshu.com/p/6122cecec54a 2.FASTA和FASTQ文件格式 https://www.jianshu.com/p/50ff30 ...
- GWAS | 全基因组关联分析 | Linkage disequilibrium (LD)连锁不平衡 | 曼哈顿图 Manhattan_plot | QQ_plot | haplotype phasing
现在GWAS已经属于比较古老的技术了,主要是碰到严重的瓶颈了,单纯的snp与表现的关联已经不够,需要具体的生物学解释,这些snp是如何具体导致疾病的发生的. 而且,大多数病找到的都不是个别显著的snp ...
- 如何鉴定全基因组加倍事件(WGD)
目前鉴定全基因组加倍(whole-genome duplication events)有3种 通过染色体共线性(synteny) 方法是比较两个基因组的序列,并将同源序列的位置绘制成点状图,如果能在点 ...
- 【GWAS文献解读】疟原虫青蒿素抗药性的全基因组关联分析
英文名:Genetic architecture of artemisinin-resistant Plasmodium falciparum 中文名:疟原虫青蒿素抗药性的全基因组关联分析 期刊:Na ...
- Genome-wide Complex Trait Analysis(GCTA)-全基因组复杂性状分析
GCTA(全基因组复杂性状分析)工具开发目的是针对复杂性状的全基因组关联分析,评估SNP解释的表型方差所占的比例(该网站地址:http://cnsgenomics.com/software/gcta/ ...
随机推荐
- 搭建LAMP环境部署Nextcloud私人网盘
搭建 LAMP 环境部署 Nextcloud 私人网盘 前言 Nextcloudd 是一个开源的.基于本地的文件共享和协作平台,它允许您保存文件并通过多个设备(如PC.智能手机和平板电脑)访问它们. ...
- pre -regulator 前端稳压器
regulator
- STM32关于多线程运行的疑问
我有一个疑问,如果 STM32在操作系统环境下 能够一个线程串口中断接收数据 另一个线程 继续进行其他的操作
- ThinkPHP 全局异常处理
wqy的笔记:http://www.upwqy.com/details/273.html 在thinkphp6 和 thinkphp5 全局异常处理 稍有不同 ThinkPHP6 在 tp6 中 框架 ...
- CPU消耗,跟踪定位理论与实践
CPU消耗,跟踪定位理论与实践 一.性能指标之资源指标定位方案 1.打tprof报告方法 抓取perfpmr文件 60秒. perfpmr.sh 60 从结果文件中取出tprof.sum 或直接抓取t ...
- 深度学*点云语义分割:CVPR2019论文阅读
深度学*点云语义分割:CVPR2019论文阅读 Point Cloud Oversegmentation with Graph-Structured Deep Metric Learning 摘要 本 ...
- YOLO v4分析
YOLO v4分析 YOLO v4 的作者共有三位:Alexey Bochkovskiy.Chien-Yao Wang 和 Hong-Yuan Mark Liao.其中一作 Alexey Bochko ...
- MindSpore:自动微分
MindSpore:自动微分 作为一款「全场景 AI 框架」,MindSpore 是人工智能解决方案的重要组成部分,与 TensorFlow.PyTorch.PaddlePaddle 等流行深度学习框 ...
- ONNX 实时graph优化方法
ONNX 实时graph优化方法 ONNX实时提供了各种图形优化来提高模型性能.图优化本质上是图级别的转换,从小型图简化和节点消除,到更复杂的节点融合和布局优化. 图形优化根据其复杂性和功能分为几个类 ...
- java8 函数式编程接口
java8 函数式接口java.util.function.* @param T 入参类型 @param R 出参类型 1. Function <T,R> 例: Function<I ...
