PPI | protein-protein interaction | 蛋白互作分析
前言
做RNA-seq基因表达数据分析挖掘,我们感兴趣的其实是“基因互作”,哪些基因影响了我们这个基因G,我们的基因G又会去影响哪些基因,从而得到基因调控的机制。
直觉确实是很明确的,但是细节处却有很多问题。
我们讨论的到底是基因表达的互作,还是基因产物的互作?
------------
对于蛋白编码基因,它翻译产生蛋白,如果此蛋白不参与转录过程,理论上不可能会影响另一个基因的表达,那也就不存在基因表达的互作的,它们的基因表达被很好的隔离起来了,相互独立,互不影响。
但现在鉴定出了很多调控基因或其他在基因组上的调控序列,比如miRNA、lncRNA等,它们也都需要从基因组上转录出来,然后转录产物会去影响其他基因的表达(影响转录)。这才是基因表达互作,虽然MiRNA、lncRNA不能被称作基因。
------------
基因产物的互作就普遍了,那就是蛋白互作,也就是STRING等数据库里收集的信息。
蛋白互作也容易直观理解些,复杂的多细胞生命体,几乎所有的功能都是靠蛋白来实现的,所以有很多蛋白要互相结合(空间上)在一起来行使自己的功能。
------------
还有一个就是遗传学领域的基因互作,这与生物学的基因互作完全不同,遗传学考虑的是宏观的基因互作,站在表型的基础上。 Novel phenotypes often result from the interactions of two genes。
遗传学的基因互作是生物学基因产物互作的结果。
STRING database的挖掘
这个数据库绝对是做实验人的宝藏,里面包含了各种蛋白互作关系,不用做实验就有一大堆证据。
IPA了解一下,收费的高端分析软件,大部分就是整合的这个数据库,很多大佬喜欢用IPA来找明星基因,再来讲故事,实例请看之前解读的CSC paper。
首先了解一下STRING里面有哪些文件可以下载:
https://string-db.org/cgi/download.pl?sessionId=yMNmD7s36wS8
选你的物种,减少文件大小,常用的就是互作数据:
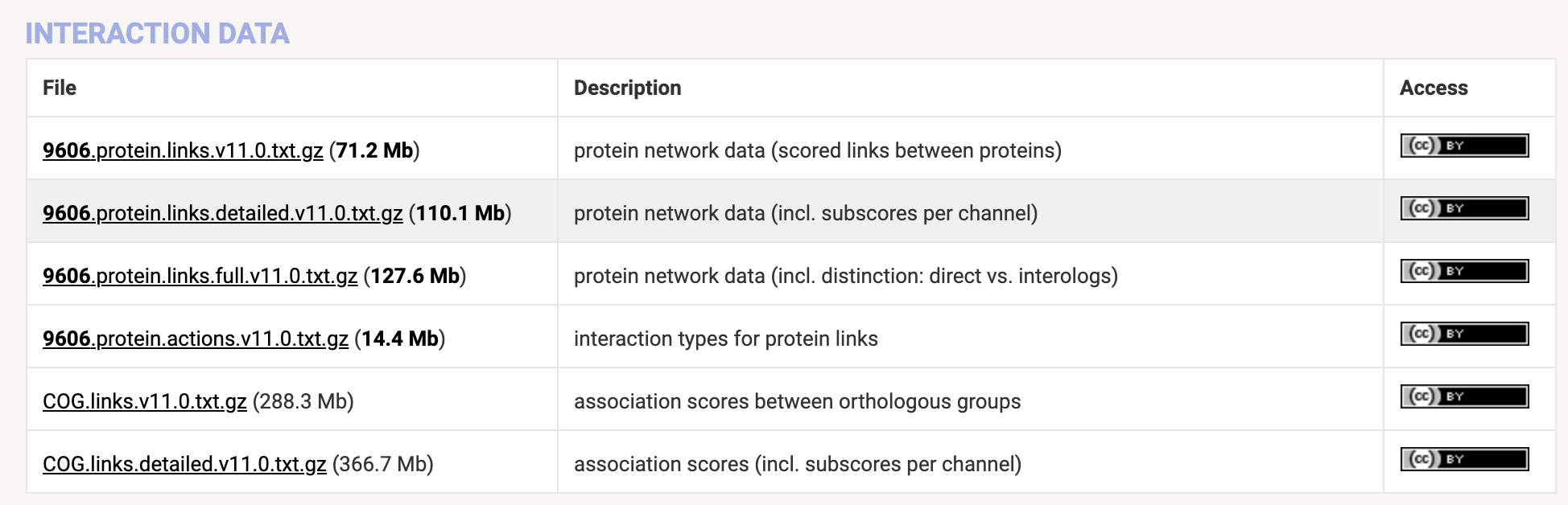
一般我们想知道某个蛋白会与哪些其他蛋白互作,以及互作的类型,然后做下游分析,信息都在这几个文件里了。
注:有哪些互作关系需要好好搞清楚,移步help,https://string-db.org/cgi/help.pl?sessionId=yMNmD7s36wS8
Docs » User documentation » Getting started » Evidence
Conserved Neighborhood
Co-occurrence
Fusion
Co-expression
Experiments
Databases
Text mining
每一个PPI关系的证据来源是不同的,选择你需要的证据。我觉得里面最可靠的就是Experiments, Databases和Text mining了。
当然,我们是高手,能用更简单的方法绝不用复杂的,那么STRING的API了解一下。
用任意脚本语言读以下格式化地址:
https://string-db.org/api/[output-format]/interaction_partners?identifiers=[your_identifiers]&[optional_parameters]
就能得到一个dataframe结果,不用下载,不用筛选,速度更快,随调随用。
实例,我想知道HDAC4的互作蛋白,可以这么抓:
老鼠:Mus%20musculus
url <- "https://string-db.org/api/tsv/interaction_partners?identifiers=HDAC4&species=Homo%20sapiens"
webDf <- read.table(url, header=T)
head(webDf) stringId_A stringId_B preferredName_A preferredName_B ncbiTaxonId score
1 ENSP00000264606 ENSP00000080059 HDAC4 HDAC7 9606 0.934
2 ENSP00000264606 ENSP00000202967 HDAC4 SIRT4 9606 0.809
3 ENSP00000264606 ENSP00000209873 HDAC4 AAAS 9606 0.901
4 ENSP00000264606 ENSP00000209875 HDAC4 CBX5 9606 0.779
5 ENSP00000264606 ENSP00000212015 HDAC4 SIRT1 9606 0.988
6 ENSP00000264606 ENSP00000215832 HDAC4 MAPK1 9606 0.572
nscore fscore pscore ascore escore dscore tscore
1 0 0 0 0.061 0.320 0.90 0.061985
2 0 0 0 0.052 0.166 0.00 0.778000
3 0 0 0 0.058 0.000 0.90 0.000000
4 0 0 0 0.062 0.463 0.54 0.159000
5 0 0 0 0.052 0.415 0.90 0.812000
6 0 0 0 0.000 0.433 0.00 0.276000
结果解读:
Output fields (TSV and JSON formats):
| Field | Description |
|---|---|
| stringId_A | STRING identifier (protein A) |
| stringId_B | STRING identifier (protein B) |
| preferredName_A | common protein name (protein A) |
| preferredName_B | common protein name (protein B) |
| ncbiTaxonId | NCBI taxon identifier |
| score | combined score |
| nscore | gene neighborhood score |
| fscore | gene fusion score |
| pscore | phylogenetic profile score |
| ascore | coexpression score |
| escore | experimental score |
| dscore | database score |
| tscore | textmining score |
抓其他信息改下API就行了
还有很多工具是基于STRING做富集分析的,也可以了解一下,主要看自己需求。
待续~
PPI | protein-protein interaction | 蛋白互作分析的更多相关文章
- BioGRID 互作数据库
01 — BioGRID BioGRID 是 Biological General Repository for Interactionh Datasets 的缩写(网址为 https://thebi ...
- 下载STRING数据库检索互作关系结果为空,但是在STRING网站却能检索出互作关系,为什么呢???关键词用的是蛋白ID(ENSP开头)
首先介绍下两种方法: 一.本地分析 1.在STRING数据库下载人的互作文件,如下图,第一个文件 https://string-db.org/cgi/download.pl?sessionId=HGr ...
- 汇编语言(学习笔记----寄存器CPU互作原理)
一.段寄存器 1.段寄存器就是提供段地址的,8086CPU有4个段寄存器:CS(代码段寄存器),DS(数据段寄存器),SS(堆栈段寄存器),ES(附加段寄存器) 2.当8086CPU要访问内存时,由这 ...
- 解读人:谭亦凡,Macrophage phosphoproteome analysis reveals MINCLE-dependent and -independent mycobacterial cord factor signaling(巨噬细胞磷酸化蛋白组学分析揭示MINCLE依赖和非依赖的分支杆菌索状因子信号通路)(MCP换)
发表时间:2019年4月 IF:5.232 一. 概述: 分支杆菌索状因子TDM(trehalose-6,6’-dimycolate)能够与巨噬细胞C-型凝集素受体(CLR)MINCLE结合引起下游通 ...
- 蛋白组DIA分析:Spectronaut软件使用指南
官方文档: https://biognosys.com/media.ashx/spectronautmanual.pdf 0. 准备 Spectronaut软件是蛋白组DIA分析最常用的谱图解析软件之 ...
- 用R的igraph包来画蛋白质互作网络图 | PPI | protein protein interaction network | Cytoscape
igraph语法简单,画图快速. Cytoscape专业,个性定制. 最终效果图: 当然也可以用Cytoscape来画. 参考:Network visualization with R Cytosca ...
- Quantitative proteomics of Uukuniemi virus-host cell interactions reveals GBF1 as proviral host factor for phleboviruses(乌库涅米病毒-宿主细胞互作的定量蛋白质组学揭示了GBF1是个白蛉病毒的前病毒宿主因子)-解读人:谭亦凡
期刊名:Molecular & Cellular Proteomics 发表时间:(2019年12月) IF:4.828 单位:1德国海德堡大学附属医院2德国汉诺威医科大学3德国亥姆霍茲感染研 ...
- Mol Cell Proteomics. |王欣然| 基于微粒的蛋白聚合物捕获技术让能满足多种不同需求的蛋白质组学样品制备方法成为可能
大家好,本周分享的是发表在Molecular & Cellular Proteomics. 上的一篇关于蛋白质组学样本质谱分析前处理方法改进的文章,题目是Protein aggregation ...
- Journal of Proteome Research | SAAVpedia: identification, functional annotation, and retrieval of single amino acid variants for proteogenomic interpretation | SAAV的识别、功能注释和检索 | (解读人:徐洪凯)
文献名:SAAVpedia: identification, functional annotation, and retrieval of single amino acid variants fo ...
随机推荐
- CSRF原理
全称跨站伪造
- GIT学习总结--从git上拉取代码到本地
步骤: 1.输入公司的git地址https://git5b.XXXXX.com,回车: 2.在登录框中输入用户名和密码: 3.选取需要的文件下的代码 4.复制该项目的地址 5.在本地的windows命 ...
- 【论文速读】Pan He_ICCV2017_Single Shot Text Detector With Regional Attention
Pan He_ICCV2017_Single Shot Text Detector With Regional Attention 作者和代码 caffe代码 关键词 文字检测.多方向.SSD.$$x ...
- getparameter的使用
在做项目的过程中,会遇到跳转的页面,直接打开到里面的子项,这个时候,看了UI给我设计了四个页面,如果做四个页面,肯定是可以实现的.但是这个不符合前端的设计.就在想通过点击传值进去,肯定是能够获取到的. ...
- VDSR
提出SRCNN问题 context未充分利用 Convergence 慢 Scale Factor 训练指定fator的模型再重新训练其他fator的模型低效 context 对于更大的scale-f ...
- Nginx 出现 _STORAGE_WRITE_ERROR_:./Runtime/Cache/Home/
Nginx 出现 _STORAGE_WRITE_ERROR_:./Runtime/Cache/Home/ 这种情况是因为 application 没有足的权限 .需要给予777的权限就能解决了
- 在 Azure 上部署 Kubernetes 集群
在实验.演示的时候,或者是生产过程中,我经常会需要运行一些 Docker 负载.虽然这在本地计算机上十分容易,但是当你要在云端运行的时候就有点困难了.相比于本地运行,在云端运行真的太复杂了.我尝试了几 ...
- charls 抓包
一.HTTPS原理: HTTPS(Hyper Text Transfer Protocol Secure),是一种基于SSL/TLS的HTTP,所有的HTTP数据都是在SSL/TLS协议封装之上进行传 ...
- oracle使用 extract获取当前时间,并比较两个时间
--根据当前时间戳获取年月日时分秒,其中sysdate不能用于获取时分秒select systimestamp, extract(year from systimestamp) year ,extra ...
- 妙用valueForKeyPath
valueForKey与valueForKeyPath在KVC中同时出现,都可以使用,难免让开发者迷惑:心里知道肯定是不一样,但具体的用法你会吗?其实valueForKeyPath的功能更强大,支持深 ...
