吴裕雄--天生自然 R语言开发学习:基础知识
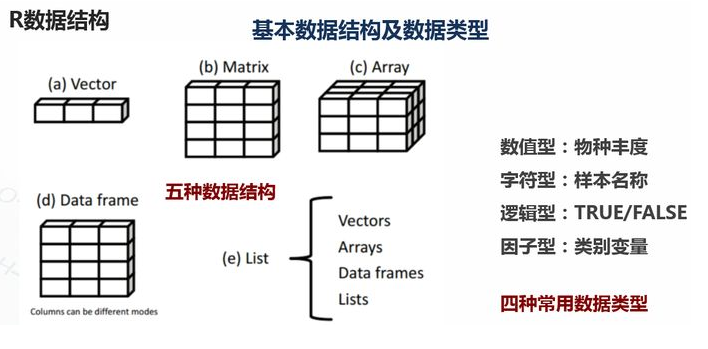

1.基础数据结构
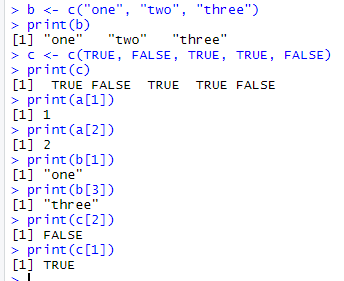
1.1 向量 # 创建向量a
a <- c(1,2,3)
print(a)


1.2 矩阵
#创建矩阵
mymat <- matrix(c(1:10), nrow=2, ncol=5, byrow=TRUE)
#取第二行
mymat[2,]
#取第二列
mymat[,2]
#第一行第五列的元素
mymat[1,5]


1.3 数组
#创建数组
myarr <- array(c(1:12),dim=c(2,3,2))
print(myarr)
#取矩阵或数组的维度
dim(myarr)
#取第一个矩阵的第一行第二列
myarr[1,2,1]


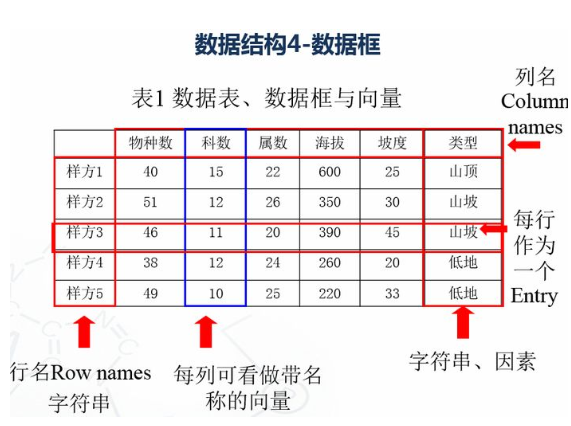
1.4 数据框
# 创建数据框
kids <- c("Wang", "Li")
age <- c("", "")
df <- data.frame(kids, age)
print(df)
#第一行
df[1,]
#第二列
df[,2]
#前两行,前两列
df[1:2,1:2]
#根据列名称
df$kids
#行名称
rownames(df)
#列名称
colnames(df) str(df)

1.4.1 因子变量
变量:类别变量,数值变量
类别数据对于分组数据研究非常有用。(男女,高中低)
R中的因子变量类似于类别数据。

1.5 列表
列表以一种简单的方式组织和调用不相干的信息,R函数的许多运行结果都是以列表的形式返回 #创建列表
lis <- list(name='fred',wife='mary',no.children=3,child.ages=c(4,7,9))
print(lis)
#列表组件名
lis$name
#列表位置访问
lis[[1]]


p <- 0.1
if(p<=0.05){
print("p<=0.05!")
}else{
print("p>0.05!")
}


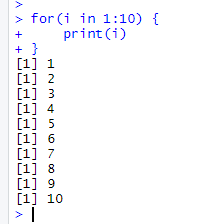
for(i in 1:10) {
print(i)
}
i <- 1
while(i<10) {
print(i)
i <- i + 1
}

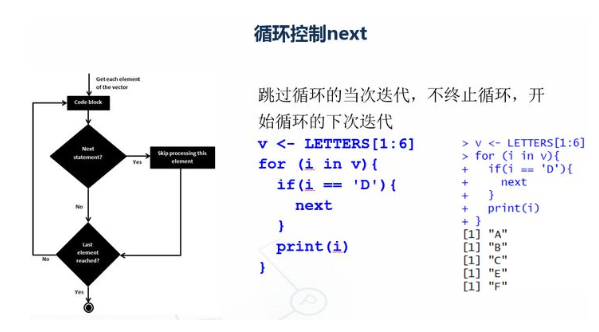
v <- LETTERS[1:6]
print(v)
for (i in v){
if(i == 'D'){
next
}
print(i)
}

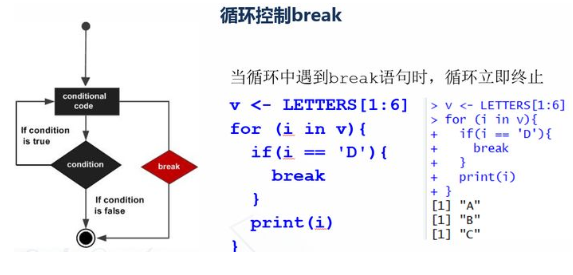
v <- LETTERS[1:6]
for (i in v){
if(i == 'D'){
break
}
print(i)
}

2.5 R函数
函数是组织好的,可重复使用的,用来实现单一,或相关联功能的代码段
rcal <- function(x,y){
z <- x^2 + y^2;
result <- sqrt(z) ;
result;
} # 调用函数
rcal(3,4)

3. 读写数据
#数据读入
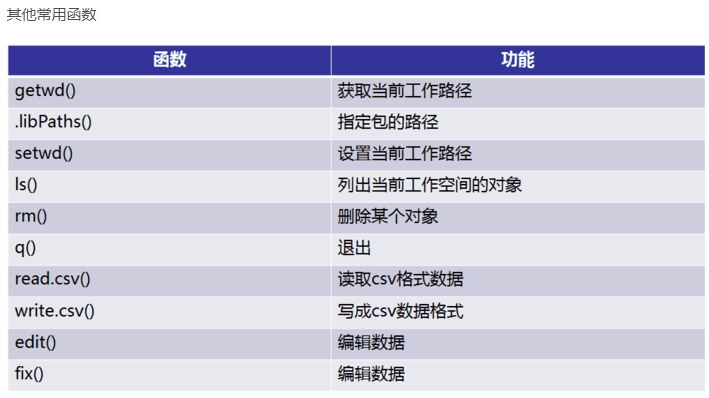
getwd()
setwd('C:/Users/Administrator/Desktop/file')
dir()
top <- read.table("otu_table.p10.relative.tran.xls",header=T,row.names=1,sep='\t',stringsAsFactors = F)
top10 <- t(top)
#数据写出logtop10<-log(top10+0.000001)
head(top10, n=2)
write.csv(logtop10,file="logtop10.csv", quote=FALSE, row.names = TRUE)
write.table(logtop10,file="logtop10.xls",sep="\t", quote=FALSE, row.names = TRUE, col.names = TRUE)

4.1 tidyr包
tidyr包的四个函数
宽数据转为长数据:gather()
长数据转为宽数据:spread()
多列合并为一列: unite()
将一列分离为多列:separate()
library(tidyr)
gene_exp <- read.table('geneExp.csv',header = T,sep=',',stringsAsFactors = F)
head(gene_exp)
#gather 宽数据转为长数据
gene_exp_tidy <- gather(data = gene_exp, key = "sample_name", value = "expression", -GeneID)
head(gene_exp_tidy)
#spread 长数据转为宽数据
gene_exp_tidy2<-spread(data = gene_exp_tidy, key = "sample_name", value = "expression")
head(gene_exp_tidy2)

4.2 dplyr包
dplyr包五个函数用法:
筛选: filter
排列: arrange()
选择: select()
变形: mutate()
汇总: summarise()
分组: group_by()
library(tidyr)
library(dplyr)
gene_exp <- read.table("geneExp.csv",header=T,sep=",",stringsAsFactors = F)
gene_exp_tidy <- gather(data = gene_exp, key = "sample_name", value = "expression", -GeneID)
#arrange 数据排列
gene_exp_GeneID <- arrange(gene_exp_tidy, GeneID)
#降序加
deschead(gene_exp_GeneID )
#filter 数据按条件筛选
gene_exp_fiter <- filter(gene_exp_GeneID ,expression>10)
head(gene_exp_fiter)
#select 选择对应的列
gene_exp_select <- select(gene_exp_fiter ,sample_name,expression)
head(gene_exp_select)


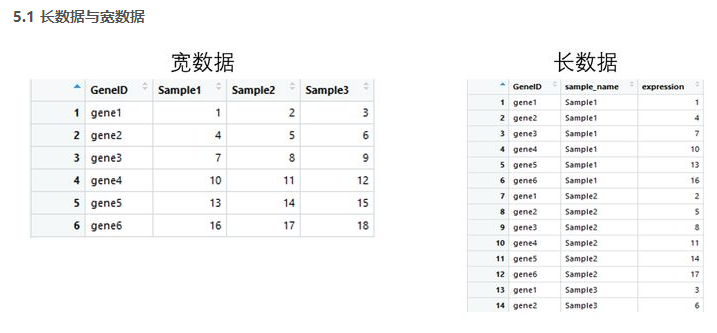
library(tidyr)
library(ggplot2)
#基础绘图
#宽数据file
file <- read.table("geneExp.csv",header=T,sep=",",stringsAsFactors = F,row.names = 1)
barplot(as.matrix(file),names.arg = colnames(file), beside =T ,col=terrain.colors(6))
legend("topleft",legend = rownames(file),fill = terrain.colors(6))
#ggplot2绘图
gene_exp <- read.table("geneExp.csv",header=T,sep=",",stringsAsFactors = F)
gene_exp_tidy <- gather(data = gene_exp, key = "sample_name", value = "expression", -GeneID)
#长数据head(gene_exp_tidy)
ggplot(gene_exp_tidy,aes(x=sample_name,y=expression,fill=GeneID)) + geom_bar(stat='identity',position='dodge')
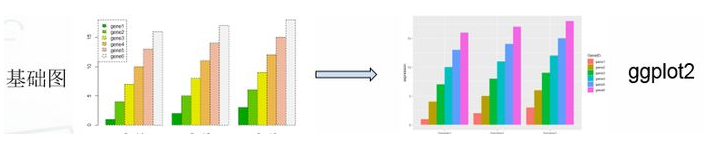

#Rnorm正态分布 个数 平均值 标准差
x <- rnorm(20, 2, 1)
y <- rnorm(20, 4, 2)
# plot是泛型函数,根据输入类型的不同而变化
#Type p 代表点 l 代表线 b 代表两者叠加
plot(x, y, cex=c(1:3),
type="p", pch=19, col = "blue",
cex.axis=1.5, col.axis="darkgreen", font.axis=2,
main="这是主标题:plot初试",
font.main=2, cex.main=2, col.main="green",
sub="这是副标题:图1",
font.sub=3, cex.sub=1.5, col.sub="red",
xlab="这是x轴标签",
ylab="这是y轴标签",
cex.lab=1.5, font.lab=2, col.lab="grey20",
xlim=c(0,3),
ylim=c(0,7)) abline(h=2, v=3, lty=1:2, lwd=2,col="red")
legend("topright", legend="我是图例\n我在这儿",text.col="red", text.width=0.5)

图形参数:
符号和线条:pch、cex、lty、lwd
颜色:col、col.axis、col.lab、col.main、col.sub、fg、bg
文本属性:cex、cex.axis、cex.lab、cex.main、cex.sub、font、font.axis、font.lab、font.main、font.sub 文本添加、坐标轴的自定义和图例
title()、main、sub、xlab、ylab、text()
axis()、abline()
legend() 多图绘制时候,可使用par()设置默认的图形参数
par(lwd=2, cex=1.5) 图形参数设置:
par(optionname=value,…)
par(pin=c(width,height)) 图形尺寸
par(mfrow=c(nr,nc)) 图形组合,一页多图
layout(mat) 图形组合,一页多图
par(mar=c(bottom,left,top,right)) 边界尺寸
par(fig=c(x1,x2,y1,y2),new=TURE) 多图叠加或排布成一幅图
#图形组合:
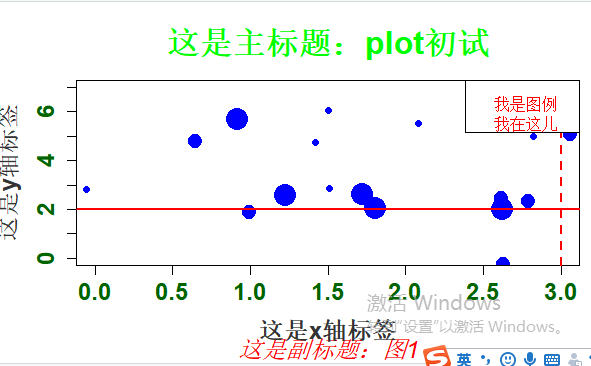
attach(mtcars)
#复制当前图形参数设置
opar <- par(no.readonly=TRUE)
#设置图形参数
par(mfrow=c(2,2))
layout(matrix(c(1,2,2,3),2,2,byrow=TRUE))
plot(wt,mpg,main="Scatterplot of wt vs mpg")
hist(wt,main="Histogram of wt")
boxplot(wt,main="Boxplot of wt")
#返回原始图形参数detach(mtcars)
par(opar)

5.3 柱形图
file <- read.table("barData.csv",header=T,row.names=1,sep=",",stringsAsFactors = F)
#转化为矩阵
dataxx <- as.matrix(file)
#抽取颜色
cols <- terrain.colors(3)
#误差线函数
plot.error <- function(x, y, sd, len = 1, col = "black") {
len <- len * 0.05
arrows(x0 = x, y0 = y, x1 = x, y1 = y - sd, col = col, angle = 90, length = len)
arrows(x0 = x, y0 = y, x1 = x, y1 = y + sd, col = col, angle = 90, length = len)
}
x <- barplot(dataxx, offset = 0, ylim=c(0, max(dataxx) * 1.1),axis.lty = 1, names.arg = colnames(dataxx), col = cols, beside = TRUE)
box()
legend("topright", legend = rownames(dataxx), fill = cols, box.col = "transparent")
title(main = "An example of barplot", xlab = "Sample", ylab = "Value")
sd <- dataxx * 0.1
for (i in 1:3) {
plot.error(x[i, ], dataxx[i, ], sd = sd[i, ])
}
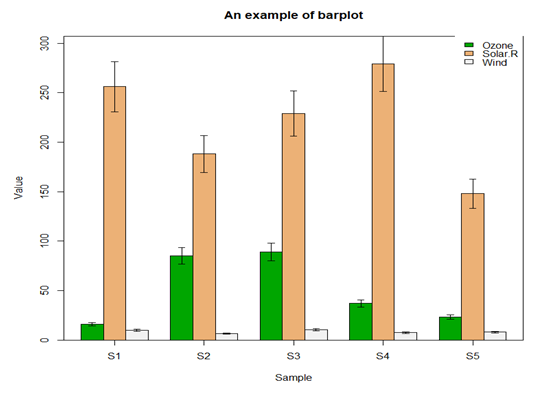
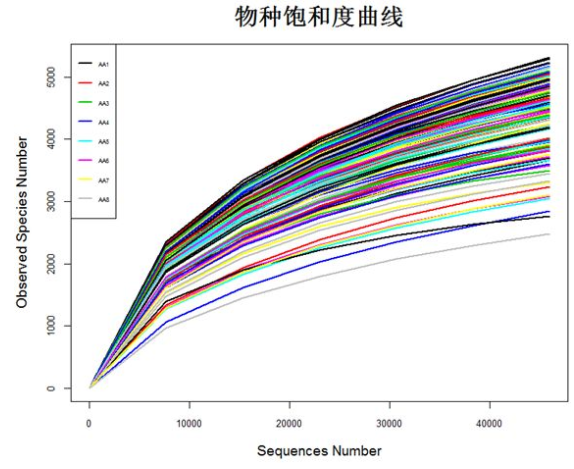
5.4 二元图
matdata <- read.table("plot_observed_species.xls", header=T)
#查看数据属性和结构
tbl_df(matdata)
y<-matdata[,2:145]
attach(matdata)
matplot(series,y,
ylab="Observed Species Number",xlab="Sequences Number",
lty=1,lwd=2,type="l",col=1:145,cex.lab=1.2,cex.axis=0.8)
legend("topleft",lty=1, lwd=2, legend=names(y)[1:8], cex=0.5,col=1:145)
detach(matdata)
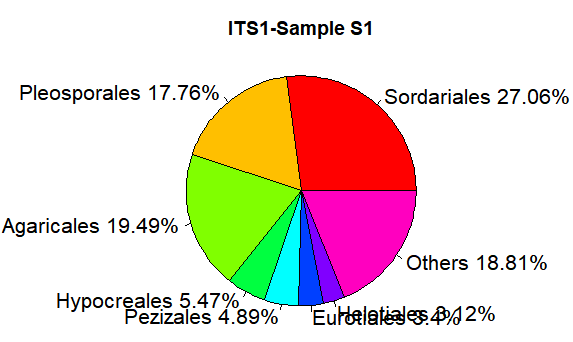
5.5 饼状图
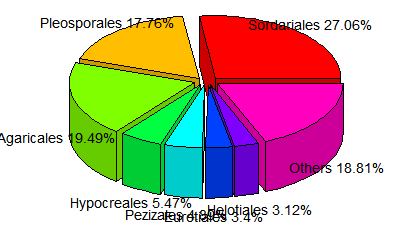
relative <- c(0.270617,0.177584,0.194911,0.054685,0.048903,0.033961, 0.031195,0.188143)
taxon <- c("Sordariales","Pleosporales","Agaricales","Hypocreales","Pezizales","Eurotiales","Helotiales","Others")
ratio <- round(relative*100,2)
ratio <- paste(ratio,"%",sep="")
label <- paste(taxon,ratio,sep=" ")
pie(relative,labels=label, main="ITS1-Sample S1",radius=1,col=rainbow(length(label)),cex=1.3)
library(plotrix)
fan.plot(relative,labels=label,main="Fan plot")
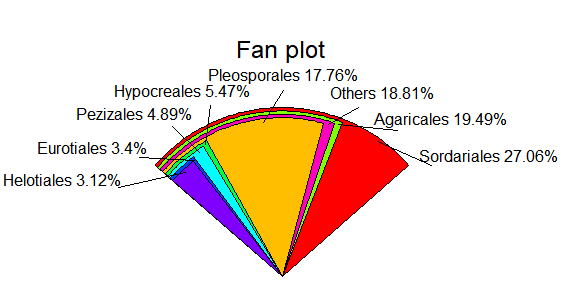
pie3D(relative,labels=label, height=0.2, theta=pi/4, explode=0.1, col=rainbow(length(label)),border="black",font=2,radius=1,labelcex=0.9)
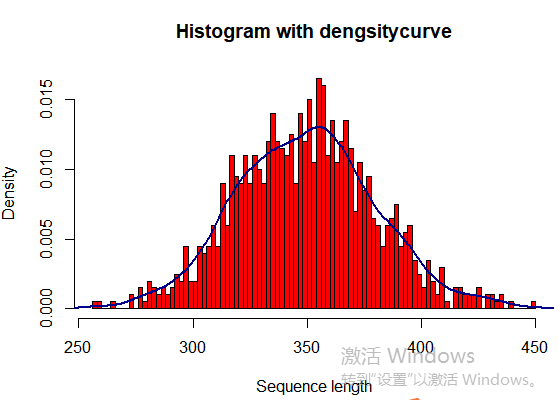
5.6 直方图
seqlength <- rnorm(1000, 350, 30) hist(seqlength,breaks=100,col="red",
freq=FALSE,
main="Histogram with dengsitycurve",
ylab="Density",
xlab="Sequence length")
lines(density(seqlength),col="blue4",lwd=2)
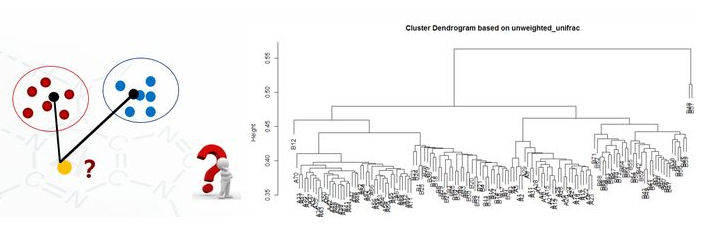
5.7 聚类图
clu <- read.table("unweighted_unifrac_dm.txt", header=T, row.names=1, sep="\t")
head(clu)
dis <- as.dist(clu)
h <- hclust(dis, method="average")
plot(h, hang = 0.1, axes = T, frame.plot = F, main="Cluster Dendrogram based on unweighted_unifrac", sub="UPGMA")

#保存图片代码
pdf(file="file.pdf", width=7, height=10)
png(file="file.png",width=480,height=480)
jpeg(file="file.png",width=480,height=480)
tiff(file="file.png",width=480,height=480) dev.off()
吴裕雄--天生自然 R语言开发学习:基础知识的更多相关文章
- 吴裕雄--天生自然 R语言开发学习:R语言的安装与配置
下载R语言和开发工具RStudio安装包 先安装R
- 吴裕雄--天生自然 R语言开发学习:数据集和数据结构
数据集的概念 数据集通常是由数据构成的一个矩形数组,行表示观测,列表示变量.表2-1提供了一个假想的病例数据集. 不同的行业对于数据集的行和列叫法不同.统计学家称它们为观测(observation)和 ...
- 吴裕雄--天生自然 R语言开发学习:导入数据
2.3.6 导入 SPSS 数据 IBM SPSS数据集可以通过foreign包中的函数read.spss()导入到R中,也可以使用Hmisc 包中的spss.get()函数.函数spss.get() ...
- 吴裕雄--天生自然 R语言开发学习:使用键盘、带分隔符的文本文件输入数据
R可从键盘.文本文件.Microsoft Excel和Access.流行的统计软件.特殊格 式的文件.多种关系型数据库管理系统.专业数据库.网站和在线服务中导入数据. 使用键盘了.有两种常见的方式:用 ...
- 吴裕雄--天生自然 R语言开发学习:R语言的简单介绍和使用
假设我们正在研究生理发育问 题,并收集了10名婴儿在出生后一年内的月龄和体重数据(见表1-).我们感兴趣的是体重的分 布及体重和月龄的关系. 可以使用函数c()以向量的形式输入月龄和体重数据,此函 数 ...
- 吴裕雄--天生自然 R语言开发学习:图形初阶(续二)
# ----------------------------------------------------# # R in Action (2nd ed): Chapter 3 # # Gettin ...
- 吴裕雄--天生自然 R语言开发学习:图形初阶(续一)
# ----------------------------------------------------# # R in Action (2nd ed): Chapter 3 # # Gettin ...
- 吴裕雄--天生自然 R语言开发学习:图形初阶
# ----------------------------------------------------# # R in Action (2nd ed): Chapter 3 # # Gettin ...
- 吴裕雄--天生自然 R语言开发学习:基本图形(续二)
#---------------------------------------------------------------# # R in Action (2nd ed): Chapter 6 ...
随机推荐
- javaweb05 文件的上传一
2.使用fileupload组件完成文件的上传应用 1).需求: I. 上传 >在upload.jsp页面上使用jQuery实现"新增一个附件","删除附件&quo ...
- Android自定义的弹窗
package com.microduino.qoobot.view; import android.app.Activity; import android.app.Dialog; import a ...
- MySQL--MySQL 日志
在 MySQL中,有 4 种不同的日志,分别是错误日志.二进制日志(BINLOG 日志).查询日志和慢查询日志. 1.错误日志 错误日志是 MySQL 中最重要的日志之一,它记录了当 mysqld 启 ...
- python-day5爬虫基础之正则表达式2
dot: '.'匹配任意的字符 '*'匹配任意多个(0到多个) 如图所示, 程序运行结果是abc,之所以没有匹配\n,是因为\n是换行符,它就代表这个字符串是两行的,而正则表达式是一行一行去匹配的.在 ...
- UI Automation编程辅助工具Inspect的下载和使用
UIAutomation微软提供的UI自动化库,主要用AutomationElement类来表示UI 自动化目录树中的一个UI自动化元素,.NET Windows的窗体应用程序和WPF应用程序. In ...
- vim下看C++代码
看C++代码, 缺少合适的编辑器,捣鼓vim. 安装Vundle, 用于插件管理 git clone https://github.com/VundleVim/Vundle.vim.git ~/.vi ...
- 9)用request方式
一个带有html的代码: <!DOCTYPE html PUBLIC "-//W3C//DTD XHTML 1.0 Transitional//EN" "http: ...
- ios AVPlayer参考
http://www.cnblogs.com/kenshincui/p/4186022.html#avPlayer
- android上线之前代码混淆加密
https://blog.csdn.net/zuiwuyuan/article/details/48552701# https://blog.csdn.net/chaoyu168/article/de ...
- Opencv笔记(十五)——图像金字塔
参考文献 目标 学习图像金字塔 学习函数cv2.pyrUp()和cv2.pyrDown() 原理 当我们需要将图像转换到另一个尺寸的时候, 有两种可能,一种是放大图像,另一种是缩小图像.尽管在Open ...
