【转录组入门】6:reads计数
作业要求:
实现这个功能的软件也很多,还是烦请大家先自己搜索几个教程,入门请统一用htseq-count,对每个样本都会输出一个表达量文件。
需要用脚本合并所有的样本为表达矩阵。参考:生信编程直播第四题:多个同样的行列式文件合并起来
对这个表达矩阵可以自己简单在excel或者R里面摸索,求平均值,方差。
看看一些生物学意义特殊的基因表现如何,比如GAPDH,β-ACTIN等等。
【1】安装计数软件:htseq-count
# conda安装
$ conda install -c bioconda htseq # 测试
# 能够在Python中导入HTSeq这个包,说明安装成功
$ python
>>> import HTSeq
>>>
【2】用htseq对排序后的bam文件进行计数
# 用htseq对排序后的bam文件进行计数
# htseq-count [options] <alignment_file> <gtf_file>
for i in `seq `
do
htseq-count -s no -r pos -f bam RNA-Seq/aligned/SRR35899${i}_sorted.bam reference/gencode.v26lift37.annotation.sorted.gtf > RNA-Seq/matrix/SRR35899${i}.count > RNA-Seq/matrix/SRR35899${i}.log
done # (实际情况:没能对gtf文件进行排序,所以使用未排序的bam文件与原始gtf文件)
for i in `seq `
do
htseq-count -s no -r pos -f bam hisat2_result/SRR35899${i}.bam reference/genome/gencode/gencode.v26lift37.annotation.gtf > htseq-count_result/SRR35899${i}.count >log_SRR35899${i}_htseq
done
-f bam/sam: 指定输入文件格式,默认SAM
-r name/pos: 你需要利用samtool sort对数据根据read name或者位置进行排序,默认是name
-s yes/no/reverse: 数据是否来自于strand-specific assay。DNA是双链的,所以需要判断到底来自于哪条链。如果选择了no, 那么每一条read都会跟正义链和反义链进行比较。默认的yes对于双端测序表示第一个read都在同一个链上,第二个read则在另一条链上。
程序运行结束后得到矩阵文件:XXXXX.count
【3】合并表达矩阵
# R脚本
# . 导入数据
# 首先将四个文件分别赋值:control1,control2,rep1,rep2
>control1 <- read.table("~/disk2/data/rna-seq/matrix/SRR3589959.count", sep="\t", col.names = c("gene_id","control1"))
>control2 <- read.table("~/disk2/data/rna-seq/matrix/SRR3589961.count", sep="\t", col.names = c("gene_id","control2"))
>rep1 <- read.table("~/disk2/data/rna-seq/matrix/SRR3589960.count", sep="\t", col.names = c("gene_id","treat1"))
>rep2 <- read.table("~/disk2/data/rna-seq/matrix/SRR3589962.count", sep="\t",col.names = c("gene_id","treat2"))
# . 数据整合
# 将四个矩阵按照gene_id进行合并,并赋值给raw_count
>raw_count <- merge(merge(control1, contol2, by="gene_id"), merge(rep1,rep2, by="gene_id"))
# 去掉前五行
>raw_count_filter <- raw_count[-:-, ]
# 因为我们无法在EBI数据库上直接搜索找到ENSMUSG00000024045.5这样的基因,只能是ENSMUSG00000024045的整数,没有小数点,所以需要进一步替换为整数的形式。
# 第一步将匹配到的.以及后面的数字连续匹配并替换为空,并赋值给ENSEMBL
>ENSEMBL <- gsub("\\.\\d*", "", raw_count_filter$gene_id)
# 将ENSEMBL重新添加到raw_count_filt1矩阵
> row.names(raw_count_filter) <- ENSEMBL
# . 查看数据整体情况
> summary(raw_count_filter)
# . 将raw_count_filter写入csv文件(可用Excel文件)
> write.csv(raw_count_filter,"d:/cs_file/r_file/file-lyx0623/H_N_rawcount.csv",row.names=FALSE)
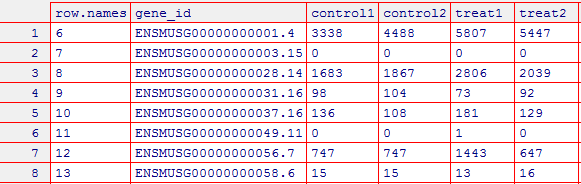

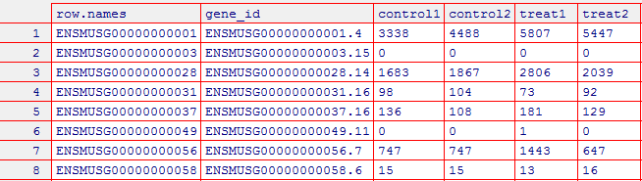
理论知识
关于比对:
】想知道已知基因的表达情况:选择alignment-free工具(例如salmon, sailfish)
】想找noval isoforms:alignment-based工具(如HISAT2, STAR)
】转录本定量:需要考虑更多的因素
1、基因表达定量有3个水平:基因水平、转录本水平、外显子使用水平

基因水平:
Htseq-count等工具作用:根据read和基因位置的overlap,判断该read属于哪个基因
转录本水平:
Cufflinks等工具处理的难题:转录本亚型(isoforms)之间通常是有重叠的,当二代测序读长低于转录本长度时,如何进行区分?这些工具大多采用的都是expectation maximization(EM)。好在我们有三代测序。
外显子使用水平:与基因水平类似。为了更好的计数,需要提供无重叠的外显子区域的gtf文件,用于分析差异外显子使用的DEXSeq提供了一个Python脚本(dexseq_prepare_annotation.py)执行这个任务。
2、reads计数后得到的基因定量结果(count matrix),在进行不同维度的比较时需要进行不同的处理(标准化):
【】比较同一个样本(within-sample)不同基因之间的表达情况---主要考虑转录本长度的影响
(因为转录本越长,那么检测的片段也会更多,直接比较等于让小孩和大人进行赛跑)
【】比较不同样本(across-sample)同一个基因的表达情况---主要考虑测序深度的影响
(测序深度越高,检测到的概率越大)
标准化的算法:RPKM(SE), FPKM(PE),TPM, TMM,RSEM等等。
一般,标准化之后才能进行差异表达分析(某些软件要求原始数据,未经标准化)
htseq参数说明
用法:htseq-count [options] alignment_file gff_file
alignment_file:比对结果文件,可以是sam格式或bam格式,可以通过后面介绍的-f参数指定,默认是sam格式。推荐使用按照name排序后的sam文件,这样的好处是消耗的内存更少,效率更高。关于输入文件排序详见参数-r。
gff_file: 包含单位信息的gff/GTF文件(gff文件格式),大多数情况下就是指注释文件; 由于GTF文件其实就是gff文件格式的变形,在这里同样可以传入GTF格式文件。
-f bam/sam: 指定输入文件格式,默认SAM
-r name/pos: 你需要利用samtool sort对数据根据read name或者位置进行排序,默认是name
-s yes/no/reverse: 数据是否来自于strand-specific assay。DNA是双链的,所以需要判断到底来自于哪条链。如果选择了no, 那么每一条read都会跟正义链和反义链进行比较。默认的yes对于双端测序表示第一个read都在同一个链上,第二个read则在另一条链上。
-a 最低质量, 剔除低于阈值的read
-m 模式 union(默认), intersection-strict and intersection-nonempty。一般而言就用默认的,作者也是这样认为的。
-i id attribute: 在GTF文件的最后一栏里,会有这个基因的多个命名方式(如下), RNA-Seq数据分析常用的是gene_id, 当然你可以写一个脚本替换成其他命名方式。
【转录组入门】6:reads计数的更多相关文章
- 【转录组入门】3:了解fastq测序数据
操作:需要用安装好的sratoolkit把sra文件转换为fastq格式的测序文件,并且用fastqc软件测试测序文件的质量 作业:理解测序reads,GC含量,质量值,接头,index,fastqc ...
- Storm入门2-单词计数案例学习
[本篇文章主要是通过一个单词计数的案例学习,来加深对storm的基本概念的理解以及基本的开发流程和如何提交并运行一个拓扑] 单词计数拓扑WordCountTopology实现的基本功能就是不停地读入 ...
- 弗雷塞斯 从生物学到生物信息学到机器学习 转录组入门(3):了解fastq测序数据
sra文件转换为fastq格式 1 fastq-dump -h --split-3 也就是说如果SRA文件中只有一个文件,那么这个参数就会被忽略.如果原文件中有两个文件,那么它就会把成对的文件按*_1 ...
- 转录组入门(3):了解fastq测序数据
sra文件转换为fastq格式 fastq-dump -h --split-3 也就是说如果SRA文件中只有一个文件,那么这个参数就会被忽略.如果原文件中有两个文件,那么它就会把成对的文件按*_1.f ...
- Bulk RNA-Seq转录组学习
与之对应的是single cell RNA-Seq,后面也会有类似文章. 参考:https://github.com/xuzhougeng/Learn-Bioinformatics/ 作业:RNA-s ...
- RNA-seq数据综合分析教程 AKAP95
https://blog.csdn.net/l_yivs?t=1 RNA-seq数据综合分析教程 2 4,055 A+ 所属分类:Transcriptomics 收 藏 2 RNA-se ...
- 【转载】Spark性能优化指南——高级篇
前言 数据倾斜调优 调优概述 数据倾斜发生时的现象 数据倾斜发生的原理 如何定位导致数据倾斜的代码 查看导致数据倾斜的key的数据分布情况 数据倾斜的解决方案 解决方案一:使用Hive ETL预处理数 ...
- 【转】【技术博客】Spark性能优化指南——高级篇
http://mp.weixin.qq.com/s?__biz=MjM5NjQ5MTI5OA==&mid=2651745207&idx=1&sn=3d70d59cede236e ...
- NOIP2017 国庆郑州集训知识梳理汇总
第一天 基础算法&&数学 day1难度测试 如果要用一个词来形容上午的测试,那真是体无完肤. 成绩: 题目 成绩 评价 T1 50 一般 T2 10 大失所望 T3 0 差 基础算法 ...
随机推荐
- [转]EOS智能合约 & 私链激活 & 基本操作
链接:https://www.jianshu.com/p/90dea623ffdf 简介 本篇文章,将跟大家介绍eos私链的激活.基础智能合约的安装,以及为大家演示转账等基础操作.还没有安装eos私链 ...
- 1个多商户、多平台版 微信小程序(多商户、多平台版),影城行业、影业连锁 多商户、多平台版微信小程序。(基于多平台版,支持在业务上 可给 每个单独影城 分发定制单独的小程序版本)
1个 影城行业 微信小程序(多商户.多平台版), 影业连锁 多商户.多平台版微信小程序.(基于多平台版,支持在业务上 可给 每个单独影城 分发定制单独的小程序版本) 资讯QQ: 876635409 ...
- 关于QQ屏蔽某些文件上传一些有意思的事
起因是因为某鱼事件... 资源倒是找了,可是因为TX的屏蔽文件上传.文件通过几次改名之后也没能上传成功(想想也没那么简单). 然后某神就写了上面那串代码实现上传.具体思想是:绕过文件摘要算法(hash ...
- padding填充属性
内边距属性: 设置元素的内容与边框之间的距离. 分4个方向(上右下左): padding-top padding-right padding-bottom padding-left 说明:值不能为负值 ...
- webservice接口和http接口介绍---更新版
web service(SOAP)与HTTP接口的区别什么是web service? soap请求是HTTP POST的一个专用版本,遵循一种特殊的xml消息格式Content-type设置为: te ...
- 冰水挑战 HDU - 6495
题目链接:http://acm.hdu.edu.cn/showproblem.php?pid=6495 题解:DP!!! dp[i][j] 表示前i个挑战,接受了j个剩余的最大体力,最后输出体力大于0 ...
- web爬虫,requests请求
requests请求,就是用yhthon的requests模块模拟浏览器请求,返回html源码 模拟浏览器请求有两种,一种是不需要用户登录或者验证的请求,一种是需要用户登录或者验证的请求 一.不需要用 ...
- 软件开发者路线图梗概&书摘chapter5
恒久学习:整个职业生涯,反馈回路,了解弱点 1.提高带宽:多维度.高效获取知识 博客.关注twitter动态.订阅邮件列表.加入本地用户组.技术大会.联系书的作者.在线教程 从信息的海洋中回到实际软件 ...
- [Python]_ELVE_centos7安装Python3.7.1与Python2共存
注:该博客转载至PengYunjing博客,加以改动. #0x01 安装依赖环境 yum -y install zlib-devel bzip2-devel openssl-devel ncurses ...
- Go 字符串连接+=与strings.Join性能对比
Go字符串连接 对于字符串的连接大致有两种方式: 1.通过+号连接 func StrPlus1(a []string) string { var s, sep string for i := 0; i ...
