DBSCAN算法及sklearn实现
基本概念:(Density-Based Spatial Clustering of Application with Noiso)
1.核心对象:
若某个点的密度达到算法设定的阈值则其为核心点。(即r领域内的点数量不小于minPts)
2.ε-领域的距离阈值:
设定的半径r
3.直接密度可达:
若某点p在点q的r领域内,且q是核心点则p-q直接密度可达
4.密度可达:
若有一个点的序列q0、q1、 ...qk,对任意qi-qi-q是直接密度可达的,则称从q0到qk密度可达,这实际上是直接密度可达的"传播"。
5.密度相连:
若从某核心点p出发,点q和点k都是密度可达的,则称点q和点k是密度相连的
6.边界点:
属于某一类的非核心点,不能发展下线了
7.直接密度可达:
若某点p在点q的r领域内,且q是核心点则p-q直接密度可达
8.噪声点:
不属于任何一个类簇的点,从任何一个核心点出发都是密度不可达的
9.可视化展示:
A:核心对象
B,C:边界点
N:离群点

工作流程:
参数D:
输入数据集
参数ε:
指定半径
MinPts:
密度阈值

半径ε,可以根据K距离来设定:找突变点
K距离:
给定数据集P={p(i);i=0,1...n},计算点P(i)到集合D的子集S中所有点之间的距离,距离按照从小到大的顺序排序,d(k)就被称为k-距离。
MinPts:
k-距离中的k值,一般取得小一些,多次尝试,这儿有个聚类可视化好玩的网址点击这里,可以感受下,挺好玩的。
优势:
不需要指定簇个数
可以发现任意形状的簇
擅长找到离群点(检测任务)
劣势:
高维数据有些困难(可以做降维)
参数难以选择(参数对结果影响非常大)
Sklearn中效率很慢(数据消减策略)
kmeans-dbcan聚类对比
- # beer dataset
- import pandas as pd
- beer = pd.read_csv('data.txt', sep=' ')
- beer
- X = beer[["calories","sodium","alcohol","cost"]]
# K-means clustering
- from sklearn.cluster import KMeans
- km = KMeans(n_clusters=3).fit(X)
- km2 = KMeans(n_clusters=2).fit(X)
- km.labels_
- array([0, 0, 0, 0, 0, 0, 0, 0, 1, 1, 0, 1, 0, 0, 0, 2, 0, 0, 2, 1])
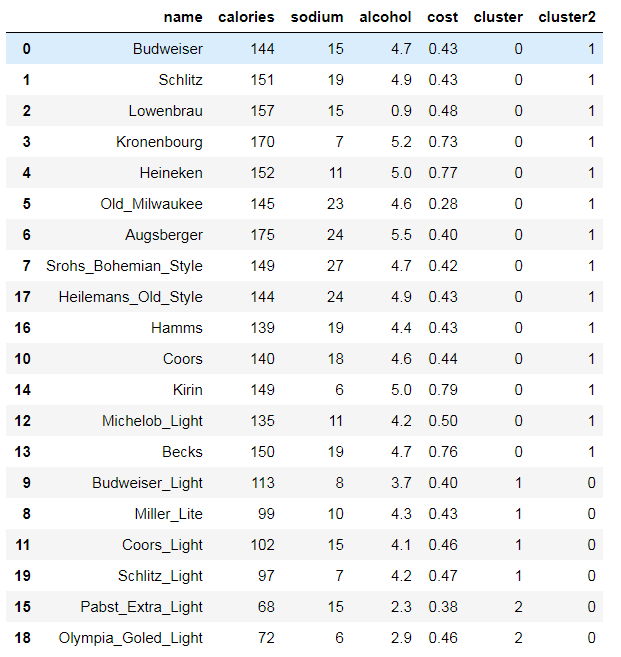
- beer['cluster'] = km.labels_
- beer['cluster2'] = km2.labels_
- beer.sort_values('cluster')
- from pandas.tools.plotting import scatter_matrix
- %matplotlib inline
- cluster_centers = km.cluster_centers_
- cluster_centers_2 = km2.cluster_centers_
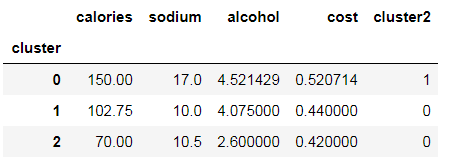
- beer.groupby("cluster").mean()
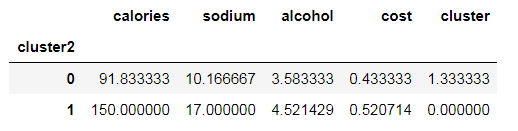
- beer.groupby("cluster2").mean()
- centers = beer.groupby("cluster").mean().reset_index()
- %matplotlib inline
- import matplotlib.pyplot as plt
- plt.rcParams['font.size'] = 14
- import numpy as np
- colors = np.array(['red', 'green', 'blue', 'yellow'])
- plt.scatter(beer["calories"], beer["alcohol"],c=colors[beer["cluster"]])
- plt.scatter(centers.calories, centers.alcohol, linewidths=3, marker='+', s=300, c='black')
- plt.xlabel("Calories")
- plt.ylabel("Alcohol")
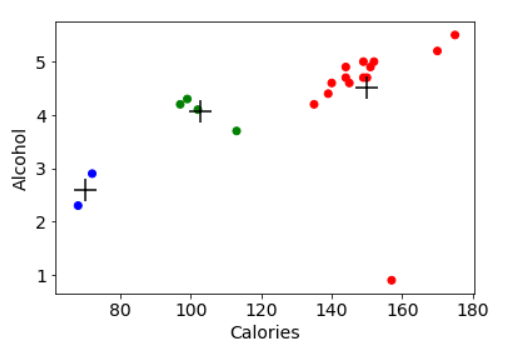
- scatter_matrix(beer[["calories","sodium","alcohol","cost"]],s=100, alpha=1, c=colors[beer["cluster"]], figsize=(10,10))
- plt.suptitle("With 3 centroids initialized")
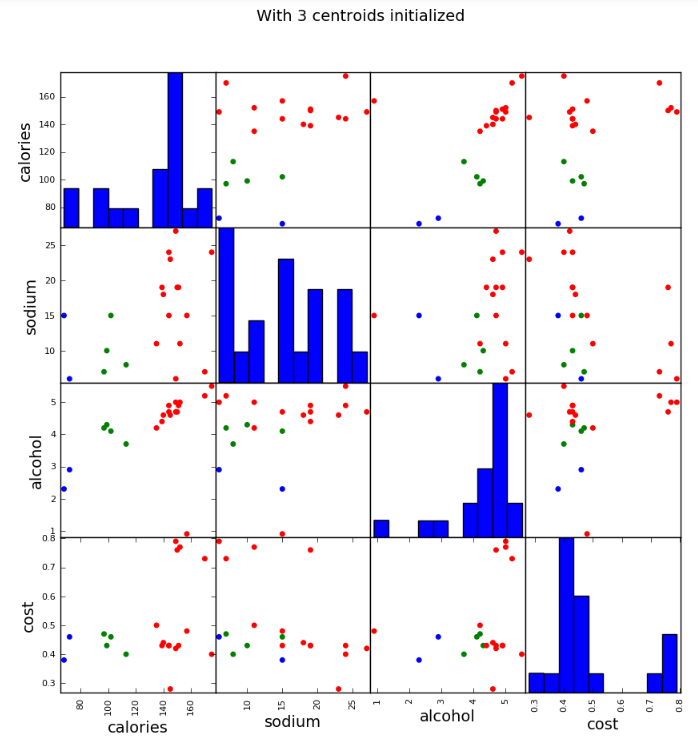
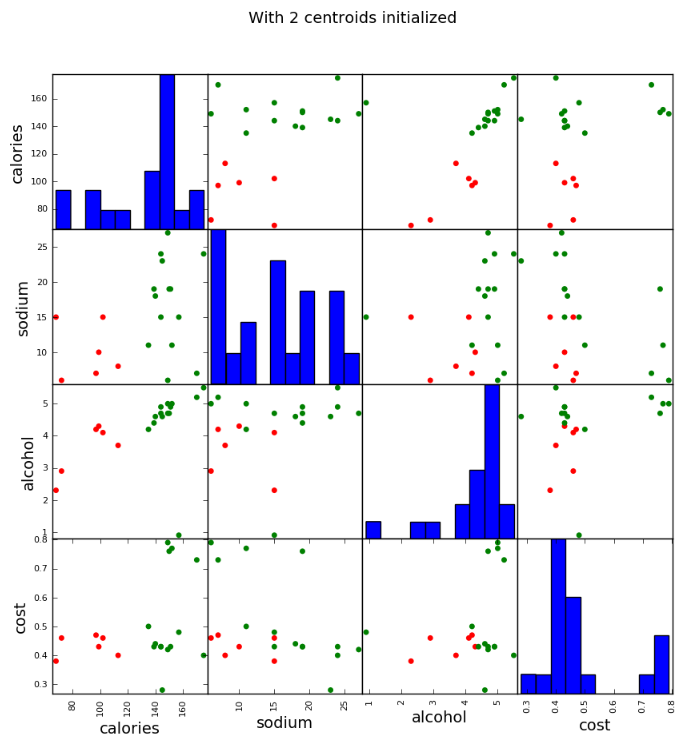
- scatter_matrix(beer[["calories","sodium","alcohol","cost"]],s=100, alpha=1, c=colors[beer["cluster2"]], figsize=(10,10))
- plt.suptitle("With 2 centroids initialized")
- from sklearn.preprocessing import StandardScaler
- scaler = StandardScaler()
- X_scaled = scaler.fit_transform(X)
- X_scaled
- array([[ 0.38791334, 0.00779468, 0.43380786, -0.45682969],
- [ 0.6250656 , 0.63136906, 0.62241997, -0.45682969],
- [ 0.82833896, 0.00779468, -3.14982226, -0.10269815],
- [ 1.26876459, -1.23935408, 0.90533814, 1.66795955],
- [ 0.65894449, -0.6157797 , 0.71672602, 1.95126478],
- [ 0.42179223, 1.25494344, 0.3395018 , -1.5192243 ],
- [ 1.43815906, 1.41083704, 1.1882563 , -0.66930861],
- [ 0.55730781, 1.87851782, 0.43380786, -0.52765599],
- [-1.1366369 , -0.7716733 , 0.05658363, -0.45682969],
- [-0.66233238, -1.08346049, -0.5092527 , -0.66930861],
- [ 0.25239776, 0.47547547, 0.3395018 , -0.38600338],
- [-1.03500022, 0.00779468, -0.13202848, -0.24435076],
- [ 0.08300329, -0.6157797 , -0.03772242, 0.03895447],
- [ 0.59118671, 0.63136906, 0.43380786, 1.88043848],
- [ 0.55730781, -1.39524768, 0.71672602, 2.0929174 ],
- [-2.18688263, 0.00779468, -1.82953748, -0.81096123],
- [ 0.21851887, 0.63136906, 0.15088969, -0.45682969],
- [ 0.38791334, 1.41083704, 0.62241997, -0.45682969],
- [-2.05136705, -1.39524768, -1.26370115, -0.24435076],
- [-1.20439469, -1.23935408, -0.03772242, -0.17352445]])
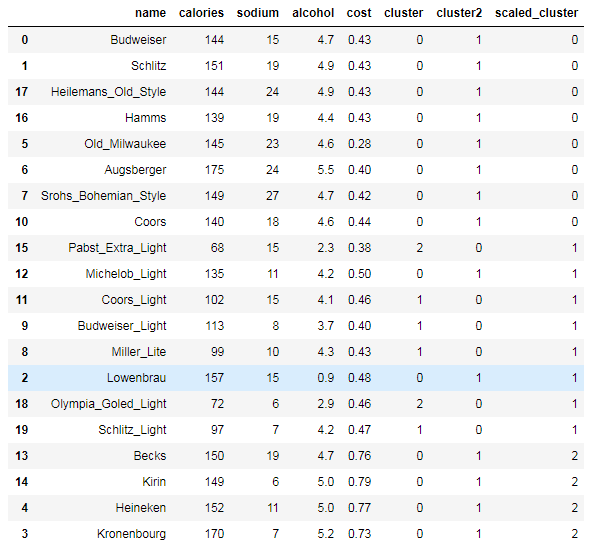
- km = KMeans(n_clusters=3).fit(X_scaled)
- beer["scaled_cluster"] = km.labels_
- beer.sort_values("scaled_cluster")
- beer.groupby("scaled_cluster").mean()

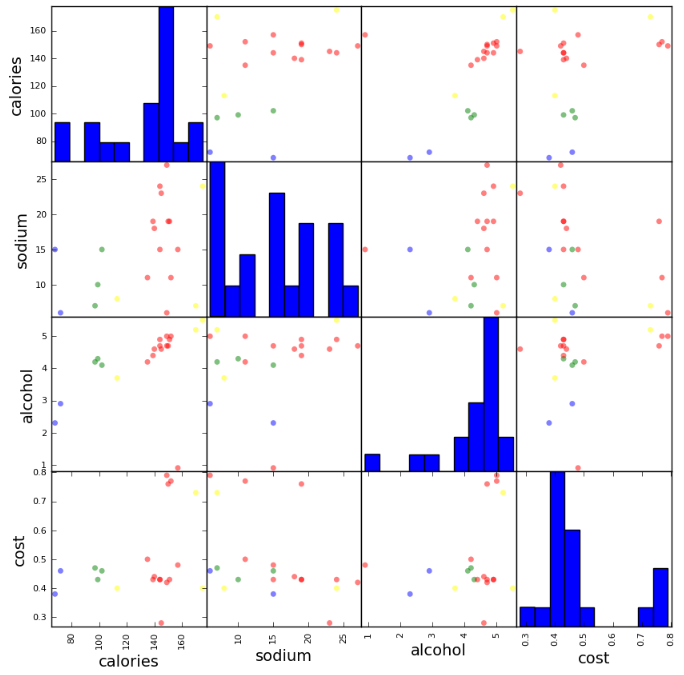
- pd.scatter_matrix(X, c=colors[beer.scaled_cluster], alpha=1, figsize=(10,10), s=100)


- from sklearn import metrics
- score_scaled = metrics.silhouette_score(X,beer.scaled_cluster)
- score = metrics.silhouette_score(X,beer.cluster)
- print(score_scaled, score)
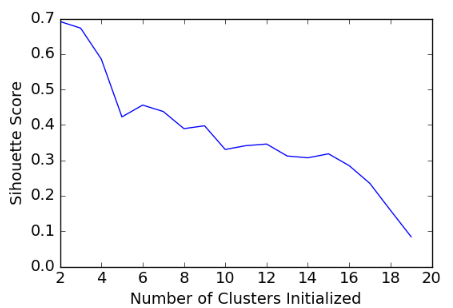
- scores = []
- for k in range(2,20):
- labels = KMeans(n_clusters=k).fit(X).labels_
- score = metrics.silhouette_score(X, labels)
- scores.append(score)
- scores
- [0.69176560340794857,
- 0.67317750464557957,
- 0.58570407211277953,
- 0.42254873351720201,
- 0.4559182167013377,
- 0.43776116697963124,
- 0.38946337473125997,
- 0.39746405172426014,
- 0.33061511213823314,
- 0.34131096180393328,
- 0.34597752371272478,
- 0.31221439248428434,
- 0.30707782144770296,
- 0.31834561839139497,
- 0.28495140011748982,
- 0.23498077333071996,
- 0.15880910174962809,
- 0.084230513801511767]
- plt.plot(list(range(2,20)), scores)
- plt.xlabel("Number of Clusters Initialized")
- plt.ylabel("Sihouette Score")
# DBSCAN clustering
- from sklearn.cluster import DBSCAN
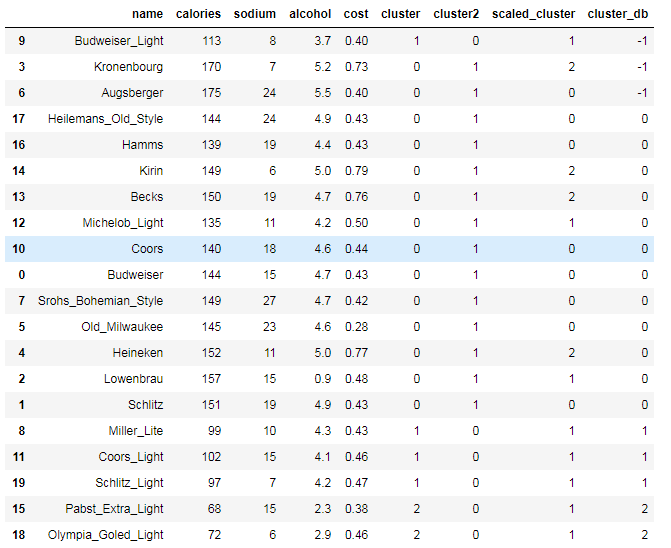
- db = DBSCAN(eps=10, min_samples=2).fit(X)
- labels = db.labels_
- beer['cluster_db'] = labels
- beer.sort_values('cluster_db')
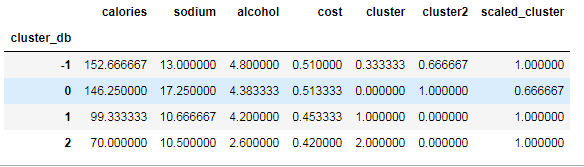
- beer.groupby('cluster_db').mean()
DBSCAN算法及sklearn实现的更多相关文章
- 挑子学习笔记:DBSCAN算法的python实现
转载请标明出处:https://www.cnblogs.com/tiaozistudy/p/dbscan_algorithm.html DBSCAN(Density-Based Spatial Clu ...
- 机器学习--聚类系列--DBSCAN算法
DBSCAN算法 基本概念:(Density-Based Spatial Clustering of Applications with Noise) 核心对象:若某个点的密度达到算法设定的阈值则其为 ...
- 机器学习 - 算法 - 聚类算法 K-MEANS / DBSCAN算法
聚类算法 概述 无监督问题 手中无标签 聚类 将相似的东西分到一组 难点 如何 评估, 如何 调参 基本概念 要得到的簇的个数 - 需要指定 K 值 质心 - 均值, 即向量各维度取平均 距离的度量 ...
- Python机器学习笔记:K-Means算法,DBSCAN算法
K-Means算法 K-Means 算法是无监督的聚类算法,它实现起来比较简单,聚类效果也不错,因此应用很广泛.K-Means 算法有大量的变体,本文就从最传统的K-Means算法学起,在其基础上学习 ...
- 【转】常用聚类算法(一) DBSCAN算法
原文链接:http://www.cnblogs.com/chaosimple/p/3164775.html#undefined 1.DBSCAN简介 DBSCAN(Density-Based Spat ...
- 常用聚类算法(一) DBSCAN算法
1.DBSCAN简介 DBSCAN(Density-Based Spatial Clustering of Applications with Noise,具有噪声的基于密度的聚类方法)是一种基于密度 ...
- 基于密度的聚类之Dbscan算法
一.算法概述 DBSCAN(Density-Based Spatial Clustering of Applications with Noise)是一个比较有代表性的基于密度的聚类算法.与划分和层次 ...
- DBSCAN算法
简单的说就是根据一个根据对象的密度不断扩展的过程的算法.一个对象O的密度可以用靠近O的对象数来判断.学习DBSCAN算法,需要弄清楚几个概念: 一:基本概念 1.:对象O的是与O为中心,为半径的空间, ...
- 数据挖掘算法:DBSCAN算法的C++实现
(期末考试快到了,所以比较粗糙,请各位读者理解..) 一. 概念 DBSCAN是一种产生划分聚类的基于密度的聚类算法,簇的个数由算法自动地确定.低密度区域中的点被视为噪声而忽略,因此DBSCAN ...
随机推荐
- Qt在控制台输出中文的解决办法(转载)
转载网址:https://blog.csdn.net/qq_22512533/article/details/75408984 按下快捷键Win+R,输入regedit打开注册变编辑器,依次找到 HK ...
- postman生成格式化时间
方法一: var moment = require('moment'); var data = moment().format(" YYYYMMDDHHmmss"); consol ...
- lua学习笔记2--table
table是lua中的一种"数据/代码结构",可以用俩创建不同的"数据类型"lua语言中的数组其实就是table类型 array = {, , , , } pr ...
- 奥比中光Astra Pro的使用(1)
在ubuntu上的使用 首先下载SDK以及OpenNI安装包,下载地址: 解压两个安装包 切换目录到AstraSDK-Linux下的install目录,并输入命令:sudo sh ./install. ...
- 使用TensorFlow训练SSD(二):数据的准备
在进行模型的训练之前,需要准备好相关的数据,相关的数据还需要进行标注.这篇博客将使用labelImg标注工具来进行数据的处理. 首先可从https://github.com/tzutalin/labe ...
- Go语言实例化结构体——为结构体分配内存并初始化
转自: http://c.biancheng.net/view/66.html 结构体的定义只是一种内存布局的描述,只有当结构体实例化时,才会真正地分配内存.因此必须在定义结构体并实例化后才能使用结构 ...
- 第五周课程总结&实验报告(四)
第五周课程总结 本周主要学习了 1.抽象类 抽象类的定义格式 abstract class抽象类名称{ 属性; 访问权限返回值类型方法名称(参数){ //普通方法 [return返回值]; } 访问权 ...
- sql server凭据
转自:https://blog.csdn.net/kk185800961/article/details/52469170 凭据是包含连接到 SQL Server 外部资源所需的身份验证信息(凭据)的 ...
- Oracle创建表空间、创建用户,给用户分配表空间以及可操作权限
创建表空间一共可分为四个步骤 具体脚本如下: 第1步:创建临时表空间 create temporary tablespace yd_temp tempfile 'D:\oracledata ...
- JS实现点击查看密码功能,再次点击隐藏密码!
<table border='1'> <tr> <td>aaaa</td> <td onclick="myFunction(this.i ...
