数据准备<5>:变量筛选-实战篇
在上一篇文章《数据准备<4>:变量筛选-理论篇》中,我们介绍了变量筛选的三种方法:基于经验的方法、基于统计的方法和基于机器学习的方法,本文将介绍后两种方法在Python(sklearn)环境下的具体实现。
1.环境介绍
版本:python2.7
工具:Spyder
开发人:hbsygfz
2.数据集介绍
数据集:sklearn中自带的cancer数据集,可参考官方介绍
from sklearn.datasets import load_breast_cancer
cancer = load_breast_cancer()
data = cancer.data
feature_names = cancer.feature_names
target = cancer.target
target_names = cancer.target_names
print("数据集大小:{}".format(data.shape))
print("特征:{}".format(feature_names))
print("目标变量:{}".format(target_names))
Out[1]:
数据集大小:(569, 30)
特征:['mean radius' 'mean texture' 'mean perimeter' 'mean area'
'mean smoothness' 'mean compactness' 'mean concavity'
'mean concave points' 'mean symmetry' 'mean fractal dimension'
'radius error' 'texture error' 'perimeter error' 'area error'
'smoothness error' 'compactness error' 'concavity error'
'concave points error' 'symmetry error' 'fractal dimension error'
'worst radius' 'worst texture' 'worst perimeter' 'worst area'
'worst smoothness' 'worst compactness' 'worst concavity'
'worst concave points' 'worst symmetry' 'worst fractal dimension']
目标变量:['malignant' 'benign']
由上可知,cancer数据集是一个(569,30)大小的数据集,共有30个特征,569条样本,目标变量为二分类,取值分别为'malignant'和'benign'。
从数据上看,发现这30个特征均为连续型数据。结合官方介绍,了解到目标变量的分布及含义如下:
3.代码实现
3.1 基于统计的方法
(0)准备工作
## 1.数据集导入
dataDf = pd.DataFrame(data,columns=list(feature_names))
targetSer = pd.Series(target)
targetSer.name = 'is_benign'
dataset = pd.concat([dataDf,targetSer],axis=1) #合并特征数据和目标变量数据
discreteColList = [] #离散型变量
continuousColList = list(feature_names) #连续型变量
targetCol = 'is_benign' #目标变量
## 2.数据预处理
# 定义连续变量分箱函数
def binCreate(df,bins):
colList = df.columns
resDf = pd.DataFrame(columns=colList)
m,n = df.shape
referSer = pd.Series(range(m))
referSer.name = 'rank'
lableSer = pd.qcut(referSer, bins, labels=range(bins))
lableSer.name = 'bin'
lableDF = pd.concat([referSer,lableSer], axis=1) #顺序与箱号合并
for col in colList:
rankSer = df[col].rank(method='min')
rankSer.name = 'rank'
rankDF = pd.concat([df[col],rankSer], axis=1)
binsDF = pd.merge(rankDF, lableDF, on='rank', how='left')
resDf[col] = binsDF['bin']
return resDf
# 定义区间(类别)分布统计函数
def binDistStatistic(df,tag):
colList = list(df.columns) #转成列表
colList.remove(tag) #删除目标变量
resDf = pd.DataFrame(columns=['colName','bin','binAllCnt','binPosCnt','binNegCnt','binPosRto','binNegRto'])
for col in colList:
allSer = df.groupby(col)[tag].count() #计算样本数
allSer = allSer[allSer>0] #剔除无效区间
allSer.name = 'binAllCnt' #定义列名
posSer = df.groupby(col)[tag].sum() #计算正样本数
posSer = posSer[allSer.index] #剔除无效区间
posSer.name = 'binPosCnt' #定义列名
tmpDf = pd.concat([allSer,posSer], axis=1) #合并统计结果
tmpDf = tmpDf.reset_index() #行索引转为一列
tmpDf = tmpDf.rename(columns={col:'bin'}) #修改区间列列名
tmpDf['colName'] = col #增加字段名称列
tmpDf['binNegCnt'] = tmpDf['binAllCnt'] - tmpDf['binPosCnt'] #计算负样本数
tmpDf['binPosRto'] = tmpDf['binPosCnt'] * 1.0000 / tmpDf['binAllCnt'] #计算正样本比例
tmpDf['binNegRto'] = tmpDf['binNegCnt'] * 1.0000 / tmpDf['binAllCnt'] #计算负样本比例
tmpDf = tmpDf.reindex(columns=['colName','bin','binAllCnt','binPosCnt','binNegCnt','binPosRto','binNegRto']) #索引重排
resDf = pd.concat([resDf,tmpDf]) #结果追加
rows, cols = df.shape
posCnt = df[tag].sum()
resDf['allCnt'] = rows #总体样本数
resDf['posCnt'] = posCnt #总体正样本数
resDf['negCnt'] = rows - posCnt #总体负样本数
resDf['posRto'] = posCnt * 1.0000 / rows #总体正样本比例
resDf['negRto'] = (rows - posCnt) * 1.0000 / rows #总体负样本比例
resDf['binPosCov'] = resDf['binPosCnt'] / resDf['posCnt']
resDf['binNegCov'] = resDf['binNegCnt'] / resDf['negCnt']
return resDf
# 定义区间(类别)属性统计函数
def binAttrStatistic(df,cont,disc,bins):
m,n = df.shape
referSer = pd.Series(range(m))
referSer.name = 'rank'
lableSer = pd.qcut(referSer, bins, labels=range(bins))
lableSer.name = 'bin'
lableDF = pd.concat([referSer,lableSer], axis=1) #顺序与箱号合并
resDf = pd.DataFrame(columns=['colName','bin','minVal','maxVal','binInterval'])
for col in cont:
rankSer = df[col].rank(method='min')
rankSer.name = 'rank'
rankDF = pd.concat([df[col],rankSer], axis=1)
binsDF = pd.merge(rankDF, lableDF, on='rank', how='left')
minSer = binsDF.groupby('bin')[col].min()
minSer.name = 'minVal'
maxSer = binsDF.groupby('bin')[col].max()
maxSer.name = 'maxVal'
tmpDf = pd.concat([minSer,maxSer], axis=1)
tmpDf = tmpDf.reset_index()
tmpDf['colName'] = col
tmpDf['binInterval'] = tmpDf['minVal'].astype('str') + '-' + tmpDf['maxVal'].astype('str')
tmpDf = tmpDf.reindex(columns=['colName','bin','minVal','maxVal','binInterval'])
tmpDf = tmpDf[tmpDf['binInterval']!='nan-nan']
resDf = pd.concat([resDf,tmpDf])
for col in disc:
binSer = pd.Series(df[col].unique())
tmpDf = pd.concat([binSer,binSer], axis=1)
tmpDf['colName'] = col
tmpDf.rename(columns={0:'bin',1:'binInterval'}, inplace = True)
tmpDf = tmpDf.reindex(columns=['colName','bin','minVal','maxVal','binInterval'])
resDf = pd.concat([resDf,tmpDf])
return resDf
# 定义结果合并函数
def binStatistic(df,cont,disc,tag,bins):
binResDf = binCreate(df[cont], bins) # 连续变量分箱
binData = pd.concat([binResDf,df[disc],df[tag]], axis=1) #合并离散变量与目标变量
binDistStatResDf = binDistStatistic(binData,tag) #对分箱后数据集进行分布统计
binAttrStatResDf = binAttrStatistic(df,cont,disc,bins) #区间(类别)大小统计
binStatResDf = pd.merge(binDistStatResDf, binAttrStatResDf, left_on=['colName','bin'], right_on=['colName','bin'], how='left')
resDf = binStatResDf.reindex(columns=['colName','bin','binInterval','minVal','maxVal','binAllCnt','binPosCnt','binNegCnt','binPosRto','binNegRto','allCnt','posCnt','negCnt','posRto','negRto','binPosCov','binNegCov'])
return resDf
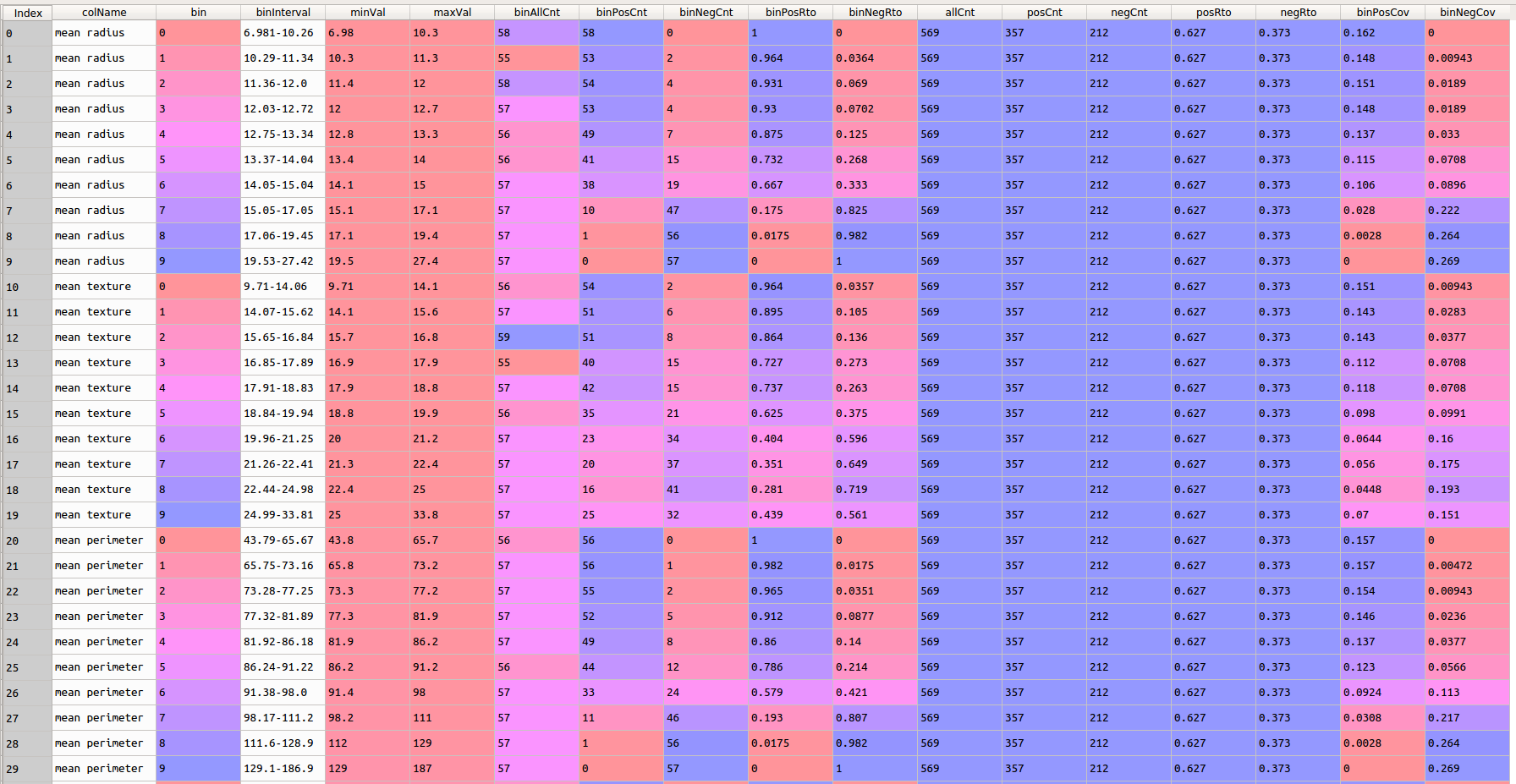
上述代码,首先将数据集中的连续变量进行等频分箱离散化,然后计算各分组中的正、负样本的比例以及占其总体的比例,方便后续预测能力指标的计算。调用:
binStatisticResDf = binStatistic(dataset, continuousColList, discreteColList, targetCol, 10)
结果如下:
(1)信息增益
##信息增益计算
import math
def entropyVal(prob):
if (prob == 0 or prob == 1):
entropy = 0
else:
entropy = -(prob * math.log(prob,2) + (1-prob) * math.log((1-prob),2))
return entropy
def gain(df,cont,disc,tag,bins):
binDf = binStatistic(df,cont,disc,tag,bins)
binDf['binAllRto'] = binDf['binAllCnt'] / binDf['allCnt'] #计算各区间样本占比
binDf['binEnty'] = binDf['binAllRto'] * binDf['binPosRto'].apply(entropyVal) #计算各区间信息熵
binDf['allEnty'] = binDf['posRto'].apply(entropyVal) #计算总体信息熵
tmpSer = binDf['allEnty'].groupby(binDf['colName']).mean() - binDf['binEnty'].groupby(binDf['colName']).sum() #计算信息增益=总体信息熵-各区间信息熵加权和
tmpSer.name = 'gain'
resSer = tmpSer.sort_values(ascending=False) #按信息增益大小降序重排
return resSer
gainSer = gain(dataset, continuousColList, discreteColList, targetCol, 10)
结果如下:
gainSer
Out[11]:
colName
worst perimeter 0.684679
worst radius 0.660968
worst area 0.660008
worst concave points 0.638077
mean concave points 0.624494
mean perimeter 0.560532
mean area 0.556175
mean radius 0.549344
mean concavity 0.522874
area error 0.511074
worst concavity 0.463328
radius error 0.365562
perimeter error 0.355755
worst compactness 0.321361
mean compactness 0.312030
concavity error 0.217127
concave points error 0.198744
mean texture 0.187825
worst texture 0.182411
worst smoothness 0.152196
worst symmetry 0.147642
compactness error 0.135060
mean smoothness 0.115767
mean symmetry 0.098459
worst fractal dimension 0.098175
mean fractal dimension 0.042941
fractal dimension error 0.042878
texture error 0.017587
smoothness error 0.016914
symmetry error 0.016343
Name: gain, dtype: float64
(2)基尼指数
##基尼指数计算
def giniVal(prob):
gini = 1 - pow(prob,2) - pow(1-prob,2)
return gini
def gini(df,cont,disc,tag,bins):
binDf = binStatistic(df,cont,disc,tag,bins)
binDf['binAllRto'] = binDf['binAllCnt'] / binDf['allCnt'] #计算各区间样本占比
binDf['binGini'] = binDf['binAllRto'] * binDf['binPosRto'].apply(giniVal) #计算各区间信息熵
binDf['allGini'] = binDf['posRto'].apply(giniVal) #计算总体信息熵
tmpSer = binDf['allGini'].groupby(binDf['colName']).mean() - binDf['binGini'].groupby(binDf['colName']).sum() #计算信息增益=总体信息熵-各区间信息熵加权和
tmpSer.name = 'gini'
resSer = tmpSer.sort_values(ascending=False) #按信息增益大小降序重排
return resSer
giniSer = gini(dataset, continuousColList, discreteColList, targetCol, 10)
结果如下:
giniSer
Out[12]:
colName
worst perimeter 0.354895
worst radius 0.342576
worst area 0.341825
worst concave points 0.335207
mean concave points 0.329700
mean area 0.301404
mean perimeter 0.300452
mean radius 0.297503
mean concavity 0.289159
area error 0.276011
worst concavity 0.256282
radius error 0.207248
perimeter error 0.197484
worst compactness 0.184931
mean compactness 0.182082
concavity error 0.118407
concave points error 0.114959
mean texture 0.110607
worst texture 0.106525
worst smoothness 0.092690
worst symmetry 0.091039
compactness error 0.078588
mean smoothness 0.067543
worst fractal dimension 0.061877
mean symmetry 0.059676
mean fractal dimension 0.027288
fractal dimension error 0.026794
texture error 0.010803
smoothness error 0.010417
symmetry error 0.010157
Name: gini, dtype: float64
(3)区分度
##区分度计算
def lift(df,cont,disc,tag,bins):
binDf = binStatistic(df,cont,disc,tag,bins)
binDf['binLift'] = binDf['binPosRto'] / binDf['posRto'] #区间提升度=区间正样本比例/总体正样本比例
tmpSer = binDf['binLift'].groupby(binDf['colName']).max() #变量区分度=max(区间提升度)
tmpSer.name = 'lift'
resSer = tmpSer.sort_values(ascending=False) #按区分度大小降序重排
return resSer
liftSer = lift(dataset, continuousColList, discreteColList, targetCol, 10)
结果如下:
liftSer
Out[13]:
colName
area error 1.593838
mean radius 1.593838
mean concavity 1.593838
perimeter error 1.593838
radius error 1.593838
mean concave points 1.593838
mean area 1.593838
worst area 1.593838
concavity error 1.593838
worst concave points 1.593838
worst perimeter 1.593838
worst radius 1.593838
mean perimeter 1.593838
worst concavity 1.565875
mean compactness 1.565376
concave points error 1.536915
compactness error 1.536915
mean texture 1.536915
worst compactness 1.536915
mean smoothness 1.508453
worst texture 1.508453
worst smoothness 1.479992
mean symmetry 1.454027
worst fractal dimension 1.398103
worst symmetry 1.366146
fractal dimension error 1.280762
mean fractal dimension 1.258293
smoothness error 1.230331
texture error 1.223840
symmetry error 1.209118
Name: lift, dtype: float64
(4)信息值(IV)
##信息值(IV)计算
def iv(df,cont,disc,tag,bins):
binDf = binStatistic(df,cont,disc,tag,bins)
binDf['binPosCovAdj'] = (binDf['binPosCnt'].replace(0,1)) / binDf['posCnt'] #调整后区间正样本覆盖率(避免值为0无法取对数)
binDf['binNegCovAdj'] = (binDf['binNegCnt'].replace(0,1)) / binDf['negCnt'] #调整后区间负样本覆盖率(避免值为0无法取对数)
binDf['woe'] = binDf['binPosCovAdj'].apply(lambda x:math.log(x,math.e)) - binDf['binNegCovAdj'].apply(lambda x:math.log(x,math.e))
binDf['iv'] = binDf['woe'] * (binDf['binPosCovAdj'] - binDf['binNegCovAdj'])
tmpSer = binDf.groupby('colName')['iv'].sum()
tmpSer.name = 'iv'
resSer = tmpSer.sort_values(ascending=False)
return resSer
ivSer = iv(dataset, continuousColList, discreteColList, targetCol, 10)
结果如下:
ivSer
Out[14]:
colName
worst perimeter 5.663336
worst area 5.407202
worst radius 5.391269
worst concave points 5.276160
mean concave points 5.117567
mean perimeter 4.643066
mean area 4.507951
mean radius 4.460431
area error 4.170720
mean concavity 3.999623
worst concavity 3.646313
perimeter error 2.777306
radius error 2.694609
worst compactness 2.320652
mean compactness 2.223346
concavity error 1.508040
concave points error 1.368055
mean texture 1.263312
worst texture 1.212502
worst smoothness 0.972226
worst symmetry 0.971215
compactness error 0.916664
mean smoothness 0.772058
mean symmetry 0.617936
worst fractal dimension 0.596305
fractal dimension error 0.253930
mean fractal dimension 0.244841
texture error 0.099411
smoothness error 0.087521
symmetry error 0.083463
Name: iv, dtype: float64
这里,我们对比上述4个指标计算的重要性排序结果。
gainRankSer = gainSer.rank(ascending=False,method='min') #并列排名取最小排名
giniRankSer = giniSer.rank(ascending=False,method='min') #并列排名取最小排名
liftRankSer = liftSer.rank(ascending=False,method='min') #并列排名取最小排名
ivRankSer = ivSer.rank(ascending=False,method='min') #并列排名取最小排名
resDf = pd.concat([gainRankSer,giniRankSer,liftRankSer,ivRankSer],axis=1)
resDf2 = resDf.sort_values(by='gain',ascending=True) #使用gain排名进行重排
结果如下:
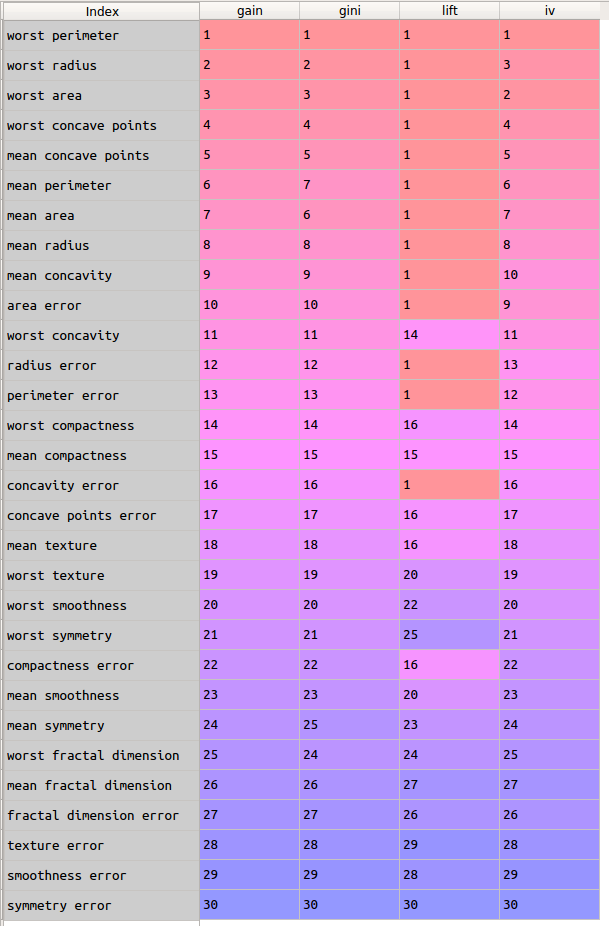
通过颜色深浅,可以很清晰的看到:
1)信息增益、基尼指数降低值的评估结果基本没有差别,二者与信息值(IV)在极个别的特征上有较小的差别;
2)区分度与其他三个指标,整体上差别不大,但是其对部分变量的评估结果相同,不能像其他三个指标那样这些变量的预测能力区分开(如表中预测能力前10名的变量)。
综上所述,在实际工程实践中,可以将多个指标综合起来看,以确定最终的显著变量范围,而不是仅使用一个指标来进行判断和筛选。
3.2 基于机器学习的方法
(0)准备工作
为了更能体现基于机器学习的变量筛选的效果,向cancer数据集中增加噪声特征。
#增加噪声数据
import numpy as np
rng = np.random.RandomState(33) #设定种子数,保证生成的随机数相同
noise = rng.normal(size=(len(data),50)) #基于这个随机数产生一个50个维度的正态分布数据
noiseDf = pd.DataFrame(noise)
dataNoiseDf = pd.concat([dataDf,noiseDf],axis=1) #与噪声数据合并
#划分样本集
from sklearn.model_selection import train_test_split
X_train, X_test, y_train, y_test = train_test_split(dataNoiseDf, targetSer, random_state=33, test_size=0.40) #划分训练集和测试集
(1)单一模型
SelectFromModel是sklearn中用于基于模型的特征选择的类,它可以选出重要性程度大于给定阈值的所有特征。
##基于单一算法
from sklearn.feature_selection import SelectFromModel
from sklearn.ensemble import RandomForestClassifier
select1 = SelectFromModel(RandomForestClassifier(n_estimators=100, random_state=0), threshold='median') #使用中位数作为阈值,选出40个显著特征
select1.fit(X_train, y_train)
X_train_l1 = select1.transform(X_train)
print("X_train.shape:{}".format(X_train.shape))
print("X_train_l1.shape:{}".format(X_train_l1.shape))
#可视化判定结果,黑色为显著特征,白色为非显著特征
import matplotlib.pyplot as plt
mask1 = select1.get_support()
print("Effective Features:{}".format(list(mask1)))
plt.matshow(mask1.reshape(1,-1), cmap='gray_r')
plt.xlabel("Feature Index")
结果如下:
Out[20]:
X_train.shape:(341, 80)
X_train_l1.shape:(341, 40)
Effective Features:[True, True, True, True, True, True, True, True, True, True, True, False, True, True, True, True, True, True, True, True, True, True, True, True, True, True, True, True, True, True, True, True, False, True, False, True, False, False, False, False, False, False, True, True, False, True, False, False, False, False, False, False, False, False, False, False, False, False, True, False, False, False, False, True, False, False, False, False, False, False, True, False, False, False, False, False, False, False, False, True]

从图中可以清晰看到,只有1个原始特征没有被选择,并且大多数噪声特征都被剔除掉。
(2)迭代
RFE(recursive feature elimination,递归特征消除)是sklearn中一种基于迭代的特征选择类,它从所有特征开始构建模型,并根据模型舍弃最不重要的特征,然后使用除了被舍弃的特征之外的全部特征构建一个新模型,如此反复,直到仅剩下预设数量的特征为止。
##基于迭代
from sklearn.feature_selection import RFE
select2 = RFE(RandomForestClassifier(n_estimators=100, random_state=42), n_features_to_select=40) #直接设定最后要保留的特征个数为40个
select2.fit(X_train, y_train)
X_train_l2 = select2.transform(X_train)
#可视化判定结果,黑色为显著特征,白色为非显著特征
mask2 = select2.get_support()
print("Effective Features:{}".format(list(mask2)))
plt.matshow(mask2.reshape(1,-1), cmap='gray_r')
plt.xlabel("Feature Index")
结果如下:
Out[22]:
Effective Features:[True, True, True, True, True, True, True, True, True, True, True, False, True, True, True, False, True, True, False, False, True, True, True, True, True, True, True, True, True, True, False, False, False, True, True, True, True, False, False, False, False, True, False, True, False, False, False, False, False, False, False, False, False, False, False, True, False, True, False, True, False, False, False, True, False, True, False, False, False, True, True, False, False, False, False, False, False, False, False, True]

从图中可以清晰看到,有4个原始特征没有被选择,并且大多数噪声特征都被剔除掉,相比上一种基于单模型的效果要差一点。
4. 参考与感谢
[1] Python机器学习基础教程
[2] Python数据分析与数据化运营
[3] sklearn.datasets.load_breast_cancer
[4] sklearn.feature_selection.SelectFromModel
[5] sklearn.feature_selection.RFE
数据准备<5>:变量筛选-实战篇的更多相关文章
- 数据准备<4>:变量筛选-理论篇
在上一篇文章<数据准备<3>:数据预处理>中,我们提到降维主要包括两种方式:基于特征选择的降维和基于维度转换的降维,其中基于特征选择的降维通俗的讲就是特征筛选或者变量筛选,是指 ...
- Systemd 入门教程:实战篇
Systemd 入门教程:实战篇 上一篇文章,介绍了 Systemd 的主要命令,这篇文章主要介绍如何使用 Systemd 来管理我们的服务,以及各项的含义: 一.开机启动 对于那些支持 System ...
- VB6基本数据库应用(五):数据的查找与筛选
同系列的第五篇,上一篇在:http://blog.csdn.net/jiluoxingren/article/details/9633139 数据的查找与筛选 第4篇发布到现在已经过了4天,很抱歉,学 ...
- Ceres Solver: 高效的非线性优化库(二)实战篇
Ceres Solver: 高效的非线性优化库(二)实战篇 接上篇: Ceres Solver: 高效的非线性优化库(一) 如何求导 Ceres Solver提供了一种自动求导的方案,上一篇我们已经看 ...
- 《大数据实时计算引擎 Flink 实战与性能优化》新专栏
基于 Flink 1.9 讲解的专栏,涉及入门.概念.原理.实战.性能调优.系统案例的讲解. 专栏介绍 扫码下面专栏二维码可以订阅该专栏 首发地址:http://www.54tianzhisheng. ...
- 如何编写高质量的 JS 函数(4) --函数式编程[实战篇]
本文首发于 vivo互联网技术 微信公众号 链接:https://mp.weixin.qq.com/s/ZoXYbjuezOWgNyJKmSQmTw作者:杨昆 [编写高质量函数系列],往期精彩内容: ...
- 算法---FaceNet在Tf下的实战篇
FaceNet---Tensorflow下的下的实战篇 @WP20190225 ===============目录=============== 一.FaceNet算法简介 二.FaceNet配置与使 ...
- SAS数据挖掘实战篇【五】
SAS数据挖掘实战篇[五] SAS--预测模型 6.1 测模型介绍 预测型(Prediction)是指由历史的和当前的数据产生的并能推测未来数据趋势的知识.这类知识可以被认为是以时 间为关键属性的关联 ...
- SAS数据挖掘实战篇【四】
SAS数据挖掘实战篇[四] 今天主要是介绍一下SAS的聚类案例,希望大家都动手做一遍,很多问题只有在亲自动手的过程中才会有发现有收获有心得. 1 聚类分析介绍 1.1 基本概念 聚类就是一种寻找数据之 ...
随机推荐
- util.promisify 的那些事儿
util.promisify是在node.js 8.x版本中新增的一个工具,用于将老式的Error first callback转换为Promise对象,让老项目改造变得更为轻松. 在官方推出这个工具 ...
- Struts2笔记2--动态方法调用和Action接收请求方式
动态方法调用(在请求的时候,再明确具体的响应方法,配置的时候不明确): LoginAction类中有两个方法some和second 1. 动态方法的调用(修改常量struts.enable.Dynam ...
- 你的组织使用了 windows defender 应用程序控制来阻止此应用
=============================================== 2018/2/8_第1次修改 ccb_warlock === ...
- nodejs 接收上传的图片
1.nodejs接收上传的图片主要是使用formidable模块,服务器是使用的express搭建. 引入formidable var formidable = require('./node_mod ...
- java基础18 String字符串和Object类(以及“equals” 和 “==”的解析)
一.String字符串 问:笔试题:new String("abc")创建了几个对象?答:两个对象,一个对象是 位于堆内存,一个对象位于字符串常量池 class Demo17 { ...
- Gitflow工作流
什么是Gitflow工作流 Gitflow工作流定义了一个围绕项目发布的严格分支模型.虽然比功能分支工作流复杂几分,但提供了用于一个健壮的用于管理大型项目的框架. Gitflow工作流没有用超出功能分 ...
- python_Appium测试环境搭建
Android环境搭建 移动端Appium环境部署比Web的selenium环境稍微复杂一些,如用python编写测试用例脚本或者开发测试框架以及UI自动化操作方法是一样的,基本是通用.因两者都是基于 ...
- LongAdder类学习笔记
优秀原文 LongAdder | LongAccumulator简介 源码阅读:全方位讲解LongAdder 说到LongAdder,不得不提的就是AtomicLong.AtomicLong是JDK1 ...
- js中的call,apply,bind区别
在JavaScript中,call.apply和bind是Function对象自带的三个方法,这三个方法的主要作用是改变函数中的this指向. call.apply.bind方法的共同点和区别:app ...
- bash常用快捷键和命令
在使用Linux的时候,最常见的终端解释器就是bash了.bash下有很多技巧,我知道这么几个: 0.关于按键模式bash默认的按键模式是emacs风格的.你也可以通过set -i vi设定为vi风格 ...
