安装生物信息学软件-MetaPhlAn2
上周20161021-20161028的任务还没有搞完,所以今天来填坑(微笑脸)
××××××××××××××××××××我是萌萌哒分割线×××××××××××××××××××××××××××××××
本文参考:https://bitbucket.org/biobakery/biobakery/wiki/metaphlan2
MetaPhlAn2是使用宏基因组鸟枪法测序数据描绘微生物群落组成的工具。

ps: 与系统发育相关的图的表示

安装:
1. 下载并解压 2. 修改/etc/profile,添加路径 3. 直接使用metaphlan2.py
4. 使用这种命令时要给出bowtie2的路径--bowtie2_exe<bowtie2>.
××××××××××××××××××××××××××啦啦啦××××××××××××××××××××××××××××
帮助信息:metaphlan2.py -h | less使用上下箭头滚动查看,q退出。
实验内容:
Step 0 : 下载数据(右键,copy link address)
mkdir mytest
cd mytest
wget https://bitbucket.org/biobakery/biobakery/raw/tip/demos/biobakery_demos/data/metaphlan2/input/SRS014459-Stool.fasta.gz
Step 1: 使用单个reads文件作为输入
metaphlan2.py SRS014476-Supragingival_plaque.fasta.gz --input_type fasta > SRS014476-Supragingival_plaque_profile.txt
bowtie2out.txt: intermediate mapping results to unique sequence markers
可以用这个命令查看(不过我觉得用gedit也可以啊==除非是命令行)
less -S SRS014476-Supragingival_plaque.fasta.gz.bowtie2out.txt
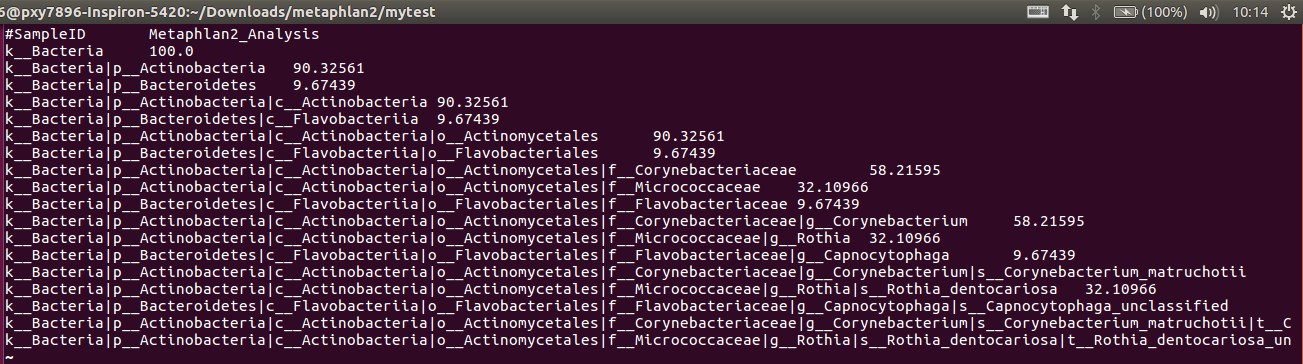
profile.txt:最终的结果
解释:
1. 这就是对分类的描述:Kingdom: k__, Phylum: p__, Class: c__, Order: o__, Family: f__, Genus: g__, Species: s__
2. 数字是百分比
3. OTU equivalents can be extracted by using only the species-level s__ clades from this file (again, making
sure to include clades unclassified at this level). Step 2: 多线程处理--nproc命令
metaphlan2.py SRS014459-Stool.fasta.gz --input_type fasta --nproc 4 > SRS014459-Stool_profile.txt
Step 3: 多输入处理
for f in *.fasta.gz
do
metaphlan2.py $f --input_type fasta --nproc 4 > ${f%.fasta.gz}_profile.txt
done Step 4: 合并结果(使用utils/merge_metaphlan_tables.py脚本)
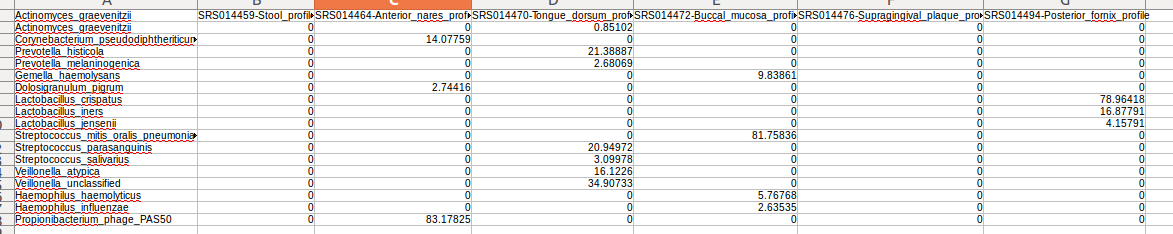
/home/pxy7896/Downloads/metaphlan2/utils/merge_metaphlan_tables.py *_profile.txt > merged_abundance_table.txt
可以用LibreOffice Calc查看。。。因为数据多,有点丑。大概如下图,前面是分类信息,后面是几个文件的数量信息。

一定要看官方手册!!!!wiki的tutorial虽然好!!!但是!!不全!!MetaPhlAn2也提供了画图的脚本,画出来也挺萌的啊!!!我干嘛要去折腾别的画图!!!
心好累!!!下次再补这个的画图好了,如果用的到的话!!!
https://bitbucket.org/biobakery/metaphlan2#markdown-header-basic-usage
Step 5: 数据可视化,heatmap
(1) 安装brew
sudo apt install linuxbrew-wrapper
(2) 安装hclust2
brew tap biobakery/biobakery
brew install hclust2
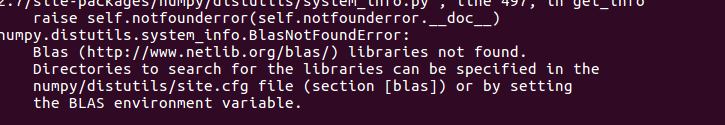
安装失败。。。问题诡异。。。所以我决定先安装R语言包,用里面的函数heatmap什么的来画。
安装R参见:
http://www.cnblogs.com/pxy7896/p/6012895.html
=============================装完R之后再来装就神奇的成功了===================================
(3) 产生物种species丰度表(只有物种层面的)
使用命令:grep -E "(s__)|(^ID)" merged_abundance_table.txt | grep -v "t__" | sed 's/^.*s__//g' > merged_abundance_table_species.txt
命令解释:
1. grep是按指定模式输出的一个软件(库),参考用户手册:http://www.gnu.org/software/grep/manual/grep.pdf
2.

3. 画图
错误1: 找不到.py,加路径:/home/pxy7896/.linuxbrew/Cellar/hclust2/1.0.0-277c0d6
错误2:
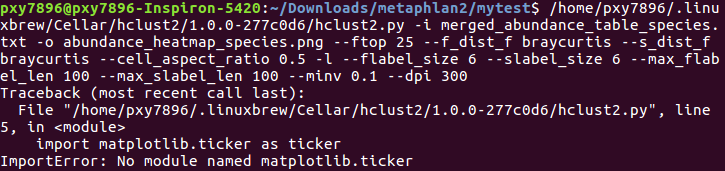
安装matplotlib
sudo apt-get install python-matplotlib
然后发现没有scipy。。。同样安装。。
然后发现没有pandas。。。同样安装。。
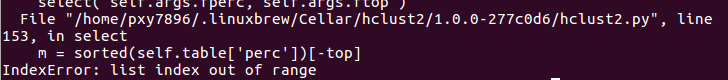
这个问题。。。我还没解决。。。。
所以直接用官网给的数据吧(输入文件存为test.txt)。。。。感觉这个坑早晚要填。立flag
/home/pxy7896/.linuxbrew/Cellar/hclust2/1.0.0-277c0d6/hclust2.py -i test.txt -o abundance_heatmap_species.png --ftop 25 --f_dist_f braycurtis --s_dist_f braycurtis --cell_aspect_ratio 0.5 -l --flabel_size 6 --slabel_size 6 --max_flabel_len 100 --max_slabel_len 100 --minv 0.1 --dpi 300
参数解释~
select the top 25 features, use Bray-Curtis as the distance measure both between samples and between features (microbes), set the ratio between the width/height of cells to 0.5, use a log scale for assigning heatmap colors, set the sample and feature label size to 6, set the max sample and feature label length to 100, select the minimum value to display as 0.1, and select an image resolution of 300

Step 6: 数据v可视化,GraPhlAn
安装:

(1) 产生输入文件:
/home/pxy7896/.linuxbrew/Cellar/graphlan/0.9.7/bin/export2graphlan.py --skip_rows 1,2 -i merged_abundance_table.txt --tree merged_abundance.tree.txt --annotation merged_abundance.annot.txt --most_abundant 100 --abundance_threshold 1 --least_biomarkers 10 --annotations 5,6 --external_annotations 7 --min_clade_size 1
real奇怪。。。我明明已经改过环境路径了,还是找不到export2graphlan.py(微笑脸)
所以只好写一串了。最后产生

命令解释: skip rows 1 and 2, select the top 100 most abundance clades to highlight, set a minimum abundance threshold for clades to be annotated, extract a minimum of 10 biomarkers, select taxonomic levels 5 and 6 to be annotated within the tree, select taxonomic level 7 to be used in the external legend, and set the minimum size of clades annotated as biomarkers to 1.
(2) 画cladogram
/home/pxy7896/.linuxbrew/Cellar/graphlan/0.9.7/bin/graphlan_annotate.py --annot merged_abundance.annot.txt merged_abundance.tree.txt merged_abundance.xml
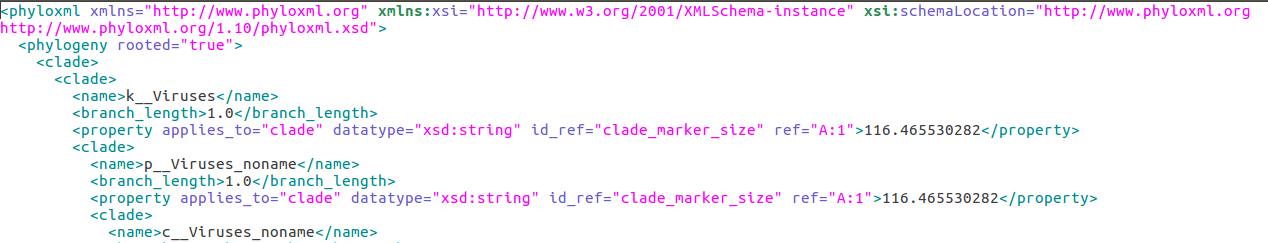
/home/pxy7896/.linuxbrew/Cellar/graphlan/0.9.7/bin/graphlan.py --dpi 300 merged_abundance.xml merged_abundance.png --external_legends
现在就出现了两个说明图片。。。和一张很好看的图~


不同颜色表示不同的分组,节点越大,丰度越高,从内而外表示门纲目科属,其中部分biomarker在树图中直接标出,其他的字母所代表的具体物种,在左侧的legend中标出。
安装生物信息学软件-MetaPhlAn2的更多相关文章
- 安装生物信息学软件-HUMAnN2
先挖坑 因为这个软件需要memory>16G,所以应该要安装在服务器上
- 安装生物信息学软件-bowtie2
好吧,这是本周(2016.10.21-28)的学习任务之一:安装bowtie2并学习其使用方法&参数设置 所以,啃文档咯,官方文档Version 2.2.9 http://bowtie-bio ...
- 安装生物信息学软件-R
主要参考http://www.3fwork.com/b211/000091MYM021616/ Step 1 : sudo gedit /etc/apt/sources.list 添加到末尾 deb ...
- 安装生物信息学软件-Samtools
装完Bowtie2,官方文档给出的栗子说可以玩一玩samtools,所以我入个坑 参考这篇http://m.010lm.com/roll/2016/0620/2343389.html Step 1: ...
- 安装生物信息学软件-Biopython
其实好多东西装过好多次,然而每次都要翻文档,经常掉进前面掉进过的坑...所以这里重新写一份指南,以防下次再装又忘了(魂淡我并不想再装了啊不要立flag) 1. 安装biopython 1.1 因为bi ...
- 单机静默安装GI软件并创建ASM实例和ASM磁盘组
环境:RHEL 6.4 + Oracle 11.2.0.4 需求:单机静默安装GI软件并创建ASM实例和ASM磁盘组,为后续迁移数据库文件到ASM做准备 1. 安装配置GI软件 2. 创建ASM实例 ...
- Linux删除apt-get安装的软件
我们都知道安装软件最简单的方法是apt-get install,但是卸载就不常用了,如何卸载呢?1.sudo apt-get remove xxxxxsudo apt-get autoremove2. ...
- Linux 中如何卸载已安装的软件(转载)
Linux 中如何卸载已安装的软件. Linux软件的安装和卸载一直是困扰许多新用户的难题.在Windows中,我们可以使用软件自带的安装卸载程序或在控制面板中的“添加/删除程序”来实 ...
- 安装ubuntu和安装ubuntu后要安装的软件列表
安装ubuntu 老毛桃进入win pe修复启动项 在win pe下面制作ubuntu的安装盘,只用的软件是ultraISO 参考博客下面的分区的那篇文章 http://jingyan.baidu. ...
随机推荐
- phpcms无法读取index.html的解决步骤
代码如下: phpcms\modules\content\classes\html.class.php 查找 复制代码 代码如下: /** * 更新首页 */ public function inde ...
- mysql查看存储过程
查询数据库中的存储过程 方法一: select `name` from mysql.proc where db = 'your_db_name' and `type` = 'PROCEDURE' 方法 ...
- zigbee学习之路(五):定时器1(查询方式)
一.前言 今天,我们来学习几乎所有单片机都有的功能,定时器的使用,定时器对单片机来说是相当重要的,有了它,单片机就可以进行一些复杂的工作. 二.原理与分析 谈到定时器的控制,我们最先想到的是要给它赋初 ...
- 【SSO单点系列】(3):CAS4.0 登录页验证码的添加
2016.08.23 更新 注意:这个教程只适合4.0版本的,4.1以及以上的版本的已经不试用了, 后面几篇有人提到过 源码网盘链接更新了下 : 链接: http://pan.baidu.com/s/ ...
- (6) 深入理解Java Class文件格式(五)
前情回顾 本专栏的前几篇博文, 对class文件中的常量池进行了详细的解释. 前文讲解了常量池中的7种数据项, 它们分别是: CONSTANT_Utf8_info CONSTANT_NameAndTy ...
- 理解GRUB2工作原理及配置选项与方法
GRUB2是借鉴GRUB改写到更加安全强大到多系统引导程序,现在大部分较新的Linux发行版都是使用GRUB2作为引导程序的.GRUB2采用了模块化设计,使得GRUB2核心更加精炼,使用更加灵活,同时 ...
- Linux 各文件夹介绍
http://www.cnblogs.com/amboyna/archive/2008/02/16/1070474.html linux下的文件结构,看看每个文件夹都是干吗用的/bin 二进制可执行命 ...
- 特征的SID表、M表、P表、Q表、X表、Y表、T表
声明:原创作品,转载时请注明文章来自SAP师太技术博客( 博/客/园www.cnblogs.com):www.cnblogs.com/jiangzhengjun,并以超链接形式标明文章原始出处,否则将 ...
- python走起之第四话
本节大纲: 一:双层装饰器:一个函数可以被多层装饰器进行装饰,函数渲染(编译)从下到上,函数执行从上到下.如下程序: 1 #!/usr/bin/env python 2 #-*-coding:utf- ...
- iOS - Project 项目
1.项目流程 1.1 分析项目的架构 iOS 常见的几种架构 标签式 Tab Menu 列表式 List Menu 抽屉式 Drawer 瀑布式 Waterfall 跳板式 Springborad 陈 ...