全网最好看的单细胞umap图绘制教程
全网最好看的单细胞umap图绘制教程
作者按
大家或许都曾被Nature, Science上的单细胞umap图吸引过,不免心生崇拜。在这里,我们将介绍一种简单方便的顶刊级umap图可视化
全文字数|预计阅读时间: 2000|5min
——Starlitnightly(星夜)
环境加载
我们先导入一些必须的依赖包
import omicverse as ov
import scanpy as sc
import matplotlib.pyplot as plt
ov.ov_plot_set()
____ _ _ __
/ __ \____ ___ (_)___| | / /__ _____________
/ / / / __ `__ \/ / ___/ | / / _ \/ ___/ ___/ _ \
/ /_/ / / / / / / / /__ | |/ / __/ / (__ ) __/
\____/_/ /_/ /_/_/\___/ |___/\___/_/ /____/\___/
Version: 1.6.3, Tutorials: https://omicverse.readthedocs.io/
读取数据
我们选取了一个结直肠癌的数据作为演示。
adata=ov.read('crc_50000.h5ad')
adata
AnnData object with n_obs × n_vars = 50000 × 25121
obs: 'n_genes', 'doublet_score', 'predicted_doublet', 'n_genes_by_counts', 'total_counts', 'total_counts_mt', 'pct_counts_mt', 'leiden', 'scsa_celltype', 'major_celltype', 'scsa_true_celltype'
var: 'gene_ids', 'n_cells', 'mt', 'n_cells_by_counts', 'mean_counts', 'pct_dropout_by_counts', 'total_counts', 'highly_variable', 'means', 'dispersions', 'dispersions_norm'
uns: 'dendrogram_leiden', 'hvg', 'leiden', 'leiden_colors', 'leiden_sizes', 'log1p', 'major_celltype_colors', 'neighbors', 'paga', 'pca', 'rank_genes_groups', 'scrublet', 'scsa_celltype_colors', 'umap'
obsm: 'X_pca', 'X_umap'
obsp: 'connectivities', 'distances'
首先我们先尝试一下最基础的umap图绘制可视化
fig, ax = plt.subplots(figsize=(4,4))
ov.pl.embedding(adata,
basis="X_umap",
color=['major_celltype'],
title='',
show=False,
size=10,
frameon='small',
ax=ax
)
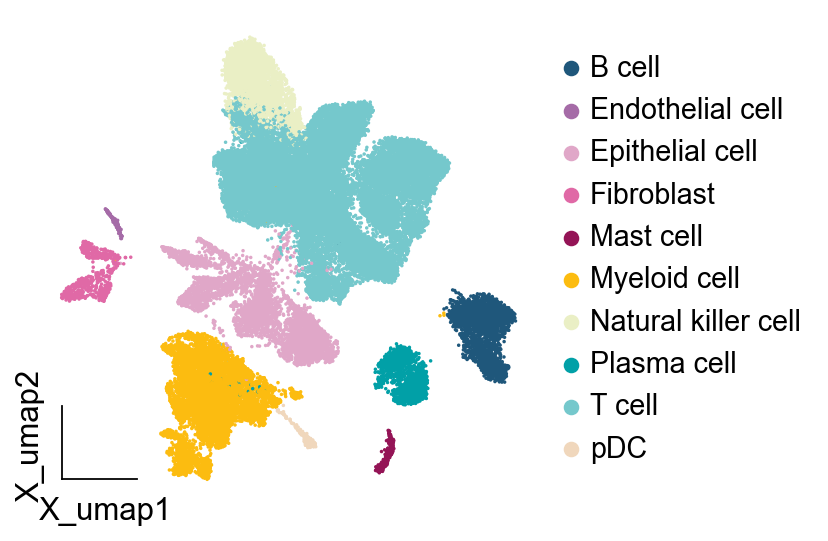
细胞可视化顺序
我们发现效果平平无奇对吧,接下来,我们首先需要修改的是,细胞可视化的顺序,将免疫细胞放到一块,将其他细胞放到另一块
adata.obs['major_celltype'].cat.categories
Index(['B cell', 'Endothelial cell', 'Epithelial cell', 'Fibroblast',
'Mast cell', 'Myeloid cell', 'Natural killer cell', 'Plasma cell',
'T cell', 'pDC'],
dtype='object')
new_order=['B cell', 'Plasma cell','T cell', 'Natural killer cell','Myeloid cell','pDC','Mast cell',
'Endothelial cell', 'Epithelial cell', 'Fibroblast',
]
adata.obs['major_celltype']=adata.obs['major_celltype'].cat.reorder_categories(new_order)
此时我们再可视化,会发现细胞的排序变成了我们指定的顺序
fig, ax = plt.subplots(figsize=(4,4))
ov.pl.embedding(adata,
basis="X_umap",
color=['major_celltype'],title='',
show=False,
frameon='small',
size=10,
ax=ax
)
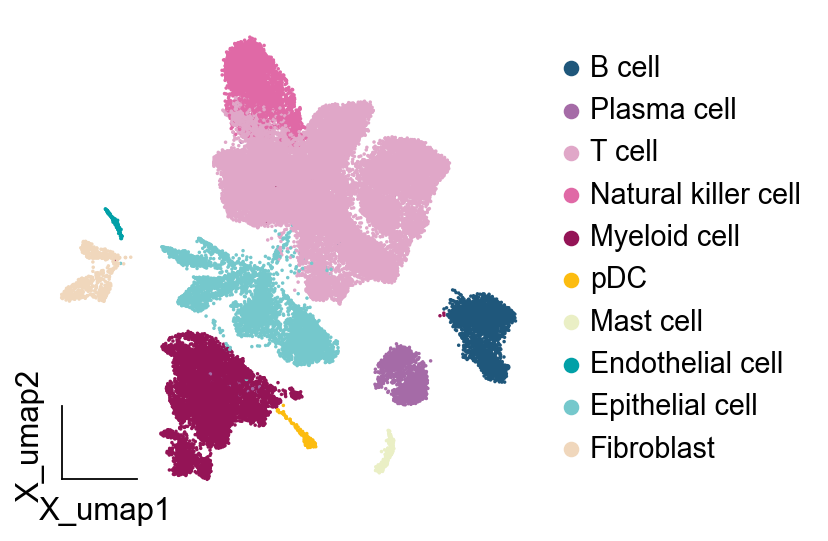
接下来我们再指定细胞类型的颜色,这里有两个方法:
- 第一个方法是我们使用颜色条顺序来指定
- 第二个方法是直接指定细胞类型的颜色
我们先尝试第一个方法,我们希望B细胞是蓝色,T细胞是红色,髓系细胞是绿色,其他细胞是黄色,那么我们根据数量指定颜色条。
需要注意的是,我们在omicverse中提供了5种顶刊级别的颜色条调用
- 红色:
ov.pl.red_color - 蓝色:
ov.pl.blue_color - 橙色:
ov.pl.orange_color - 绿色:
ov.pl.green_color - 紫色:
ov.pl.purple_color
如果你有其他的颜色需求也可以自行指定,颜色条是list格式,例如palette=['#000000','#FFFFFF']
fig, ax = plt.subplots(figsize=(4,4))
ov.pl.embedding(adata,
basis="X_umap",
color=['major_celltype'],title='',
show=False,
size=10,
frameon='small',
palette=ov.pl.blue_color[4:6]+ov.pl.purple_color[1:3]+ov.pl.green_color[:3]+ov.pl.orange_color[:],
ax=ax
)
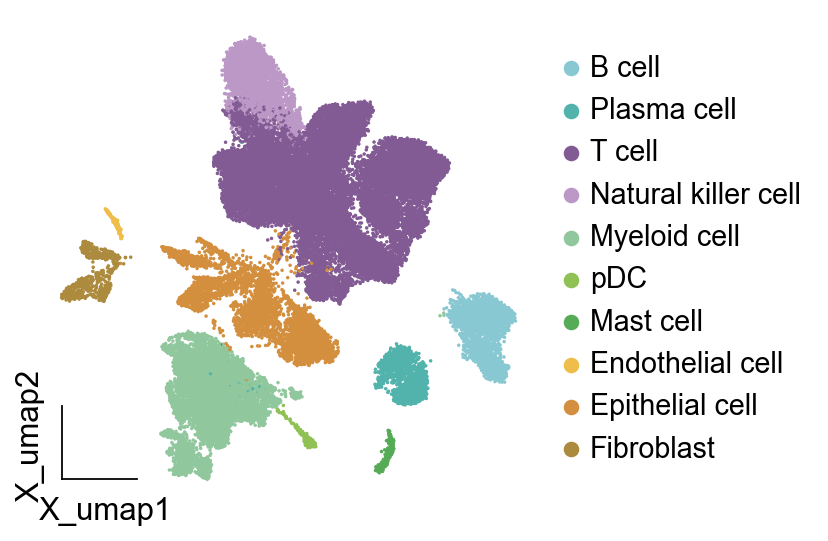
我们发现颜色变成了我们指定的颜色类型
第二种方法也很简单,我们指定每一种细胞类型的颜色然后进行可视化就好了
color_dict={
'B cell':ov.pl.blue_color[4],
'Plasma cell':ov.pl.blue_color[5],
'T cell':ov.pl.purple_color[0],
'Natural killer cell':ov.pl.purple_color[1],
'Myeloid cell':ov.pl.green_color[0],
'pDC':ov.pl.green_color[1],
'Mast cell':ov.pl.green_color[2],
'Endothelial cell':ov.pl.red_color[0],
'Epithelial cell':ov.pl.red_color[1],
'Fibroblast':ov.pl.red_color[2]
}
fig, ax = plt.subplots(figsize=(4,4))
ov.pl.embedding(adata,
basis="X_umap",
color=['major_celltype'],title='',
show=False,
frameon='small',
size=10,
palette=color_dict,
ax=ax
)
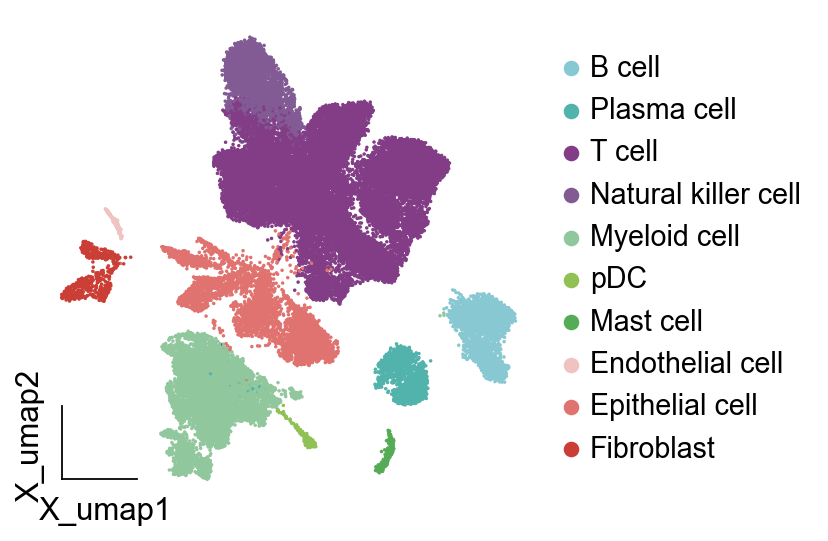
细胞标签优化
我们接下来需要优化的是细胞类型的展示,我们上面绘制的细胞的标签在右边,对于读者而言,需要一个一个去颜色对应的。在Nature最新的出版需求中,希望作者们考虑到色弱等人士的需求,因此我们可以直接在细胞umap图上绘制细胞类型。
需要注意的是,scanpy默认的细胞类型在umap图上的可视化可能出现遮挡,我们在omicverse中提供了新的函数ov.pl.embedding_adjust
from matplotlib import patheffects
fig, ax = plt.subplots(figsize=(4,4))
ov.pl.embedding(adata,
basis="X_umap",
color=['major_celltype'],title='',
show=False, legend_loc=None, add_outline=False,
frameon='small',legend_fontoutline=2,ax=ax
)
ov.pl.embedding_adjust(
adata,
basis="X_umap",
groupby='major_celltype',
ax=ax,
adjust_kwargs=dict(arrowprops=dict(arrowstyle='-', color='black')),
text_kwargs=dict(fontsize=12 ,weight='bold',
path_effects=[patheffects.withStroke(linewidth=2, foreground='w')] ),
)
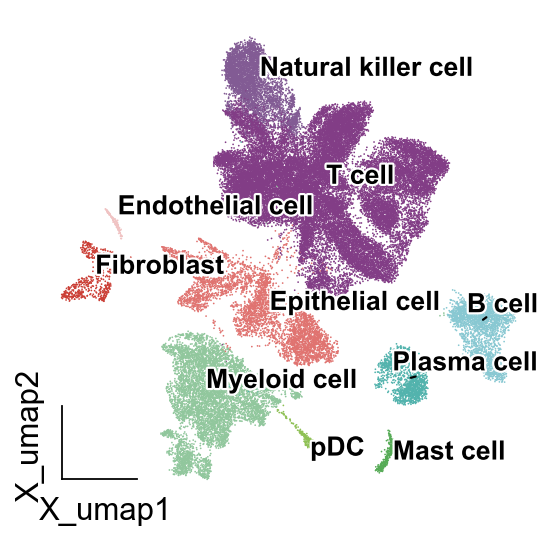
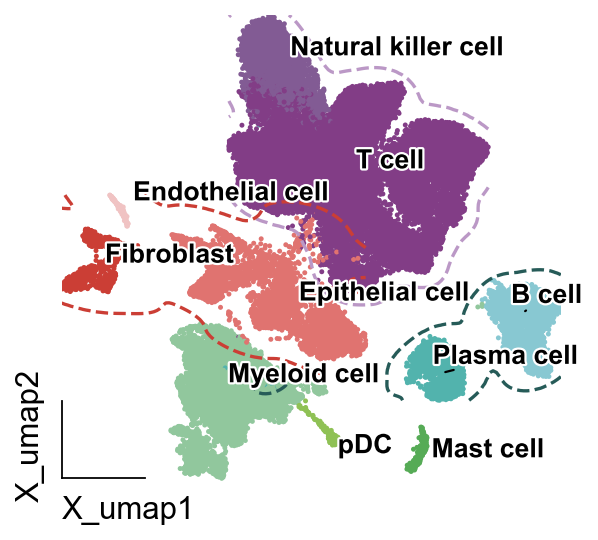
同类细胞轮廓
我们在很多顶刊中会发现,同类细胞往往作者喜欢用轮廓线圈起来,因此,我们在omicverse中也提供了类似的实现。
from matplotlib import patheffects
fig, ax = plt.subplots(figsize=(4,4))
ov.pl.embedding(adata,
basis="X_umap",
color=['major_celltype'],title='',
show=False, legend_loc=None, add_outline=False,
frameon='small',legend_fontoutline=2,ax=ax
)
ov.pl.embedding_adjust(
adata,
basis="X_umap",
groupby='major_celltype',
ax=ax,
adjust_kwargs=dict(arrowprops=dict(arrowstyle='-', color='black')),
text_kwargs=dict(fontsize=12 ,weight='bold',
path_effects=[patheffects.withStroke(linewidth=2, foreground='w')] ),
)
ov.pl.contour(ax=ax,adata=adata,
basis="X_umap",
groupby='major_celltype',clusters=['T cell','Natural killer cell'],
contour_threshold=0.02,colors=ov.pl.red_color[2],linestyles='dashed')
ov.pl.contour(ax=ax,adata=adata,
basis="X_umap",
groupby='major_celltype',clusters=['Plasma cell','B cell'],
contour_threshold=0.01,colors=ov.pl.blue_color[6],linestyles='dashed')
ov.pl.contour(ax=ax,adata=adata,
basis="X_umap",
groupby='major_celltype',clusters=['Endothelial cell', 'Epithelial cell', 'Fibroblast'],
contour_threshold=0.002,colors=ov.pl.orange_color[2],linestyles='dashed')
fig.savefig('umap-ct_major.png',dpi=300,bbox_inches='tight')
fig.savefig('umap-ct_major.pdf',dpi=300,bbox_inches='tight')
以上就是本期教程的全部内容了,如果觉得OmicVerse对你的论文有帮助,不要忘记应用哦,更多单细胞的可视化方法与教程请参考: https://starlitnightly.github.io/omicverse/Tutorials-plotting/t_visualize_single/
全网最好看的单细胞umap图绘制教程的更多相关文章
- Matlab绘图基础——利用axes(坐标系图形对象)绘制重叠图像 及 一图多轴(一幅图绘制多个坐标轴)
描述 axes在当前窗口中创建一个包含默认属性坐标系 axes('PropertyName',propertyvalue,...)创建坐标系时,同时指定它的一些属性,没有指定的使用DefaultAxe ...
- 矢量图绘制工具Svg-edit调整画布的大小
矢量图绘制工具Svg-edit调整画布的大小 ------------------------------ ------------------------
- Matlab 语谱图(时频图)绘制与分析
Matlab 语谱图(时频图)绘制与分析 语谱图:先将语音信号作傅里叶变换,然后以横轴为时间,纵轴为频率,用颜色表示幅值即可绘制出语谱图.在一幅图中表示信号的频率.幅度随时间的变化,故也称" ...
- matplotlib点线 坐标刻度 3D图绘制(六)
plot语句中支持除X,Y以外的参数,以字符串形式存在,来控制颜色.线型.点型等要素,语法形式为: plt.plot(X, Y, 'format', ...) 1 点和线的样式 颜色 参数color或 ...
- 使用JavaScript制作一个好看的轮播图
目录 使用JavaScript制作出好看的轮播图效果 准备材料 1.图片若干张(包括轮播图和按钮的图片) 2.将按钮的图片应用到按钮上的CSS样式文件 3.实现轮播和点击跳转的JavaScript代码 ...
- UML类图绘制
UML图简介 含义:UML-Unified Modeling Language 统一建模语言,又称标准建模语言.是用来对软件密集系统进行可视化建模的一种语言 主要模型: 功能模型:从用户的角度展示系统 ...
- D3力布图绘制--节点自己连自己的实现
案例分析 先看下实现的效果图 实现方法 本篇是在之前写的博文 D3力布图绘制--节点间的多条关系连接线的方法 基础上加修改的,这里放上修改的代码,其他的一样 // DATA var nodes = [ ...
- D3力布图绘制--节点跑掉,单曲线弯曲问题记录
D3力布图绘制中遇到的交互问题,频繁操作数据后,会出现节点跑掉和单曲线弯曲的问题 问题描述 在id指向都正常的情况下出现以下2种状况: 单曲线弯曲 节点跑掉 经排查,是数据重复导致的问题 线条也是一样 ...
- MATLAB之心形图绘制
一.静态心形图绘制 (1)效果展示 (2)静态心形原始代码 clc; clear all; ; % 均布三位坐标 x=-:; y=-:; z=-:; [x,y,z]=meshgrid(x,y,z); ...
- 面向对象的照妖镜——UML类图绘制指南
1.前言 感受 在刚接触软件开发工作的时候,每次接到新需求,在分析需求后的第一件事情,就是火急火燎的打开数据库(DBMS),开始进行数据表的创建工作.然而这种方式,总是会让我在编码过程中出现实体类设计 ...
随机推荐
- Vue cli之安装
1.安装node.js Node.js是一个新的后端(后台)语言,它的语法和JavaScript类似,所以可以说它是属于前端的后端语言,后端语言和前端语言的区别: 运行环境:后端语言一般运行在服务器端 ...
- 必应每日一图url(可直接使用)
必应每日一图url 首先放出地址,后面是一堆心路历程(一堆废话),只为链接的可以不用看 https://baotangguo.cn:8081/ 最初 博客园装饰的时候(虽然是抄的),想着上面背景图 ...
- mp4封装格式与MPEG4Extractor
首先来看mp4的封装格式,mp4数据都被放在一个个的箱子当中,也就是box,box的字节序为网络字节序,也就是大端存储,box由header和body组成,header指明box的大小和类型,body ...
- 获得centos7网络yum源
获得centos73网络yum源 wget http://mirrors.163.com/.help/CentOS7-Base-163.repo 网易 wget -O /etc/yum.repos.d ...
- react 高阶函数
HOC(Higher Order Components)就是一个函数,传给它一个组件,它返回一个新的组件. 高阶组件:就相当于手机壳,通过包装组件,增强组件功能. 实现步骤: 首先创建一个函数 指定函 ...
- String Record
T1. P5840 算法:ACAM+BIT+树链剖分 自然地,我们会对 \(s_i\) 建 ACAM,然后建出一颗 fail 树. 此时我们考虑集合内加入一个新的字符串.每一个匹配到的点我们都会给从这 ...
- C#.NET FRAMEWORK ASP.NET MVC 获取客户端IP
C#.NET FRAMEWORK ASP.NET MVC 获取客户端IP 工具类: using System; namespace CommonUtils { public static class ...
- 为什么说 Mybatis 是半自动 ORM 映射工具?它与全自动的区别在哪里?
Hibernate 属于全自动 ORM 映射工具,使用 Hibernate 查询关联对象或者关联集合对象时,可以根据对象关系模型直接获取,所以它是全自动的.而 Mybatis 在查询关联对象或关联集合 ...
- OpenCV + sklearnSVM 实现手写数字分割和识别
这学期机器学习考核方式以大作业的形式进行考核,而且只能使用一些传统的机器学习算法. 综合再三,选择了自己比较熟悉的MNIST数据集以及OpenCV来完成手写数字的分割和识别作为大作业. 1. 数据集准 ...
- 中国电信登录RSA算法+分析图文
Tips:当你看到这个提示的时候,说明当前的文章是由原emlog博客系统搬迁至此的,文章发布时间已过于久远,编排和内容不一定完整,还请谅解` 中国电信登录RSA算法+分析图文 日期:2016-9-30 ...
