HDU1560 DNA sequence(IDA*)题解
DNA sequence
For example, given "ACGT","ATGC","CGTT" and "CAGT", you can make a sequence in the following way. It is the shortest but may be not the only one.
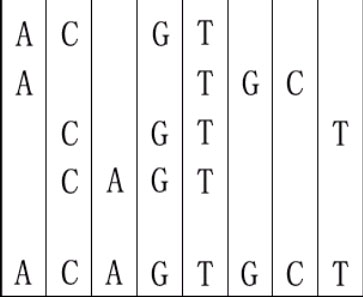
思路:
刚开始BFS就爆内存了。
新思路是给dfs加一个最小限制,超过限制就返回,然后不断加大限制直到符合,那么此时dfs的答案也是最小的。这里有几个要剪枝的地方:一是超过限制剪枝;二是预估值+当前值超过限制也要剪枝。
一开始看的那些题解都看不懂的我emmmm......orz
借鉴题解:链接
Code:
#include<cstdio>
#include<cstring>
#include<cstdlib>
#include<cctype>
#include<queue>
#include<cmath>
//#include<map>
#include<iostream>
#include<algorithm>
#define INF 0x3f3f3f3f
const int N=810;
using namespace std;
char str[10][10],dna[4]={'A','C','G','T'};
int n,deep,ans,len[10];
void dfs(int step,int pos[]){ //step为当前长度
if(step>deep) return; //超过深度返回
int maxdeep=0;
for(int i=0;i<n;i++){
maxdeep=max(len[i]-pos[i],maxdeep); //maxdeep为预估剩余深度
}
if(step+maxdeep>deep) return; //当前长度加预估剩余深度大于deep,剪枝
if(maxdeep==0){ //所有串都满足
ans=deep;
return;
}
int temp[10],flag;
for(int i=0;i<4;i++){
flag=0;
for(int j=0;j<n;j++){
if(str[j][pos[j]]==dna[i]){
temp[j]=pos[j]+1;
flag=1;
}
else temp[j]=pos[j];
}
if(flag){
dfs(step+1,temp);
}
if(ans) return;
}
}
int main(){
int t;
scanf("%d",&t);
while(t--){
deep=0;
scanf("%d",&n);
for(int i=0;i<n;i++){
scanf("%s",str[i]);
len[i]=strlen(str[i]);
deep=max(deep,len[i]); //找出最长的作为第一次深搜最小深度
}
ans=0;
int pos[10]; //表示第i组验证到第pos[i]个
memset(pos,0,sizeof(pos));
while(1){
dfs(0,pos);
if(ans) break;
deep++; //加深迭代深度,重新DFS
}
printf("%d\n",ans);
}
return 0;
}
HDU1560 DNA sequence(IDA*)题解的更多相关文章
- HDU1560 DNA sequence —— IDA*算法
题目链接:http://acm.hdu.edu.cn/showproblem.php?pid=1560 DNA sequence Time Limit: 15000/5000 MS (Java/Oth ...
- HDU1560 DNA sequence IDA* + 强力剪枝 [kuangbin带你飞]专题二
题意:给定一些DNA序列,求一个最短序列能够包含所有序列. 思路:记录第i个序列已经被匹配的长度p[i],以及第i序列的原始长度len[i].则有两个剪枝: 剪枝1:直接取最长待匹配长度.1900ms ...
- Hdu1560 DNA sequence(IDA*) 2017-01-20 18:53 50人阅读 评论(0) 收藏
DNA sequence Time Limit : 15000/5000ms (Java/Other) Memory Limit : 32768/32768K (Java/Other) Total ...
- HDU1560 DNA sequence
题目: The twenty-first century is a biology-technology developing century. We know that a gene is made ...
- HDU 1560 DNA sequence (IDA* 迭代加深 搜索)
题目地址:http://acm.hdu.edu.cn/showproblem.php?pid=1560 BFS题解:http://www.cnblogs.com/crazyapple/p/321810 ...
- HDU 1560 DNA sequence(IDA*)
题目链接:http://acm.hdu.edu.cn/showproblem.php?pid=1560 题目大意:给出n个字符串,让你找一个字符串使得这n个字符串都是它的子串,求最小长度. 解题思路: ...
- HDU 1560 DNA sequence(DNA序列)
HDU 1560 DNA sequence(DNA序列) Time Limit: 15000/5000 MS (Java/Others) Memory Limit: 32768/32768 K ...
- hdu 1560 DNA sequence(迭代加深搜索)
DNA sequence Time Limit : 15000/5000ms (Java/Other) Memory Limit : 32768/32768K (Java/Other) Total ...
- POJ2278 DNA Sequence —— AC自动机 + 矩阵优化
题目链接:https://vjudge.net/problem/POJ-2778 DNA Sequence Time Limit: 1000MS Memory Limit: 65536K Tota ...
随机推荐
- Building a Space Station---poj2031(最小生成树)
题目链接:http://poj.org/problem?id=2031 n个球型的cell,如果任意两个球表面没有接触或者没有包含关系,就选择最近的表面建立通道: 所以用maps[i][j]表示i,j ...
- lmathlib文件
lua的math库是通过lua扩展而来,主要对系统math库进行了一次封装,以供lua使用,一般可以从lmathlib文件开始阅读源码. //绝对值 static int math_abs (lua_ ...
- 【python-opencv】对象测量
opencv 中轮廓特征包括: 如面积,周长,质心,边界框等 *弧长与面积测量 *多边形拟合 *获取轮廓的多边形拟合结果 python-opencv API提供方法: cv2.moments()用来计 ...
- grunt学习二
1. 新建文件和文件目录 mkdir grunt-in-action cd grunt-in-action cd grunt-in-action mkdir grunt-empty cd grunt- ...
- idong常用js总结
1.判断屏幕高度 $(document).ready(function() { $("#left").height($(window).height()); $(&qu ...
- Unity无法创建新工程
1.解决方法 重新登录一遍账号
- java的poi技术读取Excel数据
这篇blog主要是讲述java中poi读取excel,而excel的版本包括:2003-2007和2010两个版本, 即excel的后缀名为:xls和xlsx. 读取excel和MySQL相关: ja ...
- CRM项目总结-封装PortletURLUtil
package com.ebizwindow.crm.utils; import java.security.Key; import java.util.List; import javax.port ...
- centos安装samba
yum -y install samba samba-client samba-swat /etc/init.d/smb start chkconfig --level 35 smb on cp -p ...
- 教你玩转产品管理系统iClap(PC端功能篇)
之前和大家介绍了iClap的基础功能, 这一次针对PC端右侧的工具栏再做一个详细的介绍 随着版本的更新迭代,陆续会有更多工具和功能推出! 导航 为项目成员提供网址浏览访问导航服务,帮助项目成员快速查找 ...
