DNA序列对齐问题
一、问题描述
该问题在算法导论中引申自求解两个DNA序列相似度的问题。
可以从很多角度定义两个DNA序列的相似度,其中有一种定义方法就是通过序列对齐的方式来定义其相似度。
给定两个DNA序列A和B,对齐的方式是将空格分别插入到A和B序列中,得到具有相同长度的对齐后的序列C和D;空格可以插入到任意的位置(包括两端),但是相同位置不能同时为空格,也即是不存在C[i]和D[i]同时为空格的情况。然后为对齐后的序列的每个位置打分,总分为每个位置得分之和,具体的打分规则如下:
a、如果C[i] == D[i]且都不是空格,得3分;
b、如果C[i] != D[j]且都不是空格,得1分;
c、如果C[i] 或者D[i]是空格,得0分。
求给定原序列A和B的一个对齐方案,使得该对齐方案的总分最高。
例如,序列原序列A和B如下:
String strA = "GATC";
String strB = "ATCG";
则其中一个对齐方案如下:
GATC*
*ATCG
该方案总得分score=2*0+3*3 = 9分。
实际上这是最优的对齐方案,在所有的对齐方案中总得分最高为9分。
二、问题分析
为了用更加简单的方式来表示对齐的方案,我们尝试用一些特定的字符记号来表示对齐方案,对此,首先做一个约定,对于打分规则:
1、情况a用“=”字符标记;
2、情况b用“~”字符标记;
3、情况c用“*”字符标记,但是情况c实际上可以细分为两种情况:C[i]为空格时用“+”标记,D[i]为空格时用“-”号标记。这样用“+”和“-”细分的表示相比于统一用“*”来表示,本质的区别在于让对齐方案具有所谓的“方向性”,后面会看到这样的细分对于算法的实现有一定的好处。
有了这样的约定,可以将一个对齐方案用这些字符表示出来,该字符串称之为一个对齐规则字符串R。
例如上面的例子中,对齐规则就可以用字符串“-===+”来表示。
可以推断,任何两个原序列的对齐规则字符串R的长度必然满足:
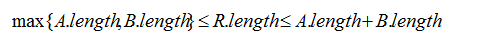
只要能够求得最优对齐方案的对齐规则字符串,就可以计算出最高分数,还可以还原出各自的对齐序列。
考察该问题的最优子结构性质,与最长公共子序列思考的角度比较类似,
用C(i,j)表示序列A[0]...A[i]和序列B[0]...B[j]的最优对齐方案的得分,不难得出其初始条件和递推求解式:

用R(i,j)表示序列A[0]...A[i]和序列B[0]...B[j]的最优对齐方案的对齐规则字符串,结合上面的递推求解式,不难推出对齐规则字符串的运算规则:

三、算法实现
package agdp;
public class Alignment {
//根据对齐骨规则生成相应的对齐后的字符串
private static String[] generate(String base,String ...origin){
int num = origin.length;
String[] align = new String[num];
for (int i = 0; i < num; i++) {
if (origin[i].length() == base.length()) {
align[i] = origin[i];
}else {//base.length()只能是等于或者大于两个原字符串的长度
String tmp = "";
for (int j = 0,k = 0; j < base.length(); j++) {
if (base.charAt(j) == '+') {
if (i == 0) {tmp = tmp+"*";}
else{tmp = tmp+origin[i].charAt(k++);}
}else if(base.charAt(j) == '-'){
if (i == 0) {tmp = tmp+origin[i].charAt(k++);}
else{tmp = tmp+"*";}
}
else {
tmp = tmp+origin[i].charAt(k++);
}
}
align[i] = tmp;
}
}
return align;
}
public static String align(String strA,String strB){
int m = strA.length(),n = strB.length(),tmp;
//aux数组记录子问题的最有对齐方案的分数,也即子问题的最高分数。
int[][] aux = new int[m+1][n+1];
//rule数组记录对齐方案,分别用"+"、"-"、"="和"~"记录四种情况。
String[][] rule = new String[m+1][n+1];
//rule初始化
rule [0][0] = "";
for (int i = 1; i < m+1; i++) {
rule[i][0] = rule[i-1][0]+"-";
}
for (int i = 1; i < n+1; i++) {
rule[0][i] = rule[0][i-1]+"+";
}
for (int i = 1; i <= m; i++) {
for (int j = 1; j <= n; j++) {
if (strA.charAt(i-1) == strB.charAt(j-1)) {
aux[i][j] = aux[i-1][j-1]+3;
rule[i][j] = rule[i-1][j-1] + "=";//A[i]==B[j]:->"="
}else {
tmp = Math.max(Math.max(aux[i-1][j], aux[i][j-1]), aux[i-1][j-1]+1);
aux[i][j] = tmp;
if (tmp == aux[i-1][j-1]-1) {//A[i]!=B[j]且A[i]和 B[j]不为空字符:->"~"
rule[i][j] = rule[i-1][j-1]+"~";
}else if(tmp == aux[i-1][j]-2){//B[i]为空字符:->"-"
rule[i][j] = rule[i-1][j]+"-";
}else{
rule[i][j] = rule[i][j-1]+"+";//A[i]为空字符:->"+"
}
}
}
}
//格式化输出aux数组
for (int i = 0; i < m+1; i++) {
for (int j = 0; j < n+1; j++) {
System.out.format("%3d",aux[i][j]);
}
System.out.println();
}
//格式化输出rule数组
for (int i = 0; i < m+1; i++) {
for (int j = 0; j < n+1; j++) {
System.out.format("%-15s",rule[i][j]);
}
System.out.println();
}
//返回最优的对齐方法对应的规则
return rule[m][n];
}
//根据规则字符串计算分数
public static int getScore(String ruleStr){
int score = 0;
for (int i = 0; i < ruleStr.length(); i++) {
if (ruleStr.charAt(i) == '=') {
score += 3;
}else if (ruleStr.charAt(i) == '~') {
score += 1;
}
}
return score;
}
public static void main(String[] args) {
// TODO Auto-generated method stub
int[] scoreAry = {3,1,0,0};
// String strA = "GATCGGCAT";
// String strB = "CAATGTGAATC";
String strA = "GATC";
String strB = "ATCG";
// String strA = "GAC";
// String strB = "ATCG";
String ruleStr = align(strA, strB);
System.out.println(ruleStr);
int score = getScore(ruleStr);
System.out.println(score);
String[] alignStr = generate(ruleStr, strA,strB);
for(String str:alignStr){
System.out.println(str);
}
}
}
还是以原始序列“GATC”和“ATCG”为例:
其子问题的得分的计算如下:
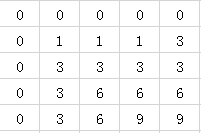
子问题的对齐规则字符串的计算如下:

需要特别注意的是,用“+”和“-”号来区分打分情况c后,对齐规则字符串是具有“方向性”的,也就是说对齐规则“-===+”是指从A->B方向的对齐规则。那如果需要B->A的对齐规则,只需要将对齐规则的字符串中+“和”-”相互替换即可。
实际上,从DNA序列对齐问题过渡到编辑距离问题是很比较自然的。本文也有意识的将这两个问题联系在一起,编辑距离问题见下一篇博文。
参考资料:
算法导论.第十五章 习题15-5
转载请注明原文出处:
http://www.cnblogs.com/qcblog/p/7820140.html
DNA序列对齐问题的更多相关文章
- [LeetCode] Repeated DNA Sequences 求重复的DNA序列
All DNA is composed of a series of nucleotides abbreviated as A, C, G, and T, for example: "ACG ...
- 利用Python【Orange】结合DNA序列进行人种预测
http://blog.csdn.net/jj12345jj198999/article/details/8951120 coursera上 web intelligence and big data ...
- 华为OJ平台——DNA序列
题目描述: 一个DNA序列由A/C/G/T四个字母的排列组合组成.G和C的比例(定义为GC-Ratio)是序列中G和C两个字母的总的出现次数除以总的字母数目(也就是序列长度).在基因工程中,这个比例非 ...
- 环状DNA序列
大意: 一个DNA序列是环状的,这意味着有N个碱基的序列有N种表示方法(假设无重复).而这N个序列有一种最小的表示,这个最小表示的意思是这个序列的字典序最小(字典序的意思是在字典中的大小 比如ABC& ...
- 简单DNA序列组装(非循环子图)
生物信息学原理作业第四弹:DNA序列组装(非循环子图) 原理:生物信息学(孙啸) 大致思想: 1. 这个算法理解细节理解比较困难,建议看孙啸的生物信息学相关章节. 2. 算法要求所有序列覆盖整个目标D ...
- DNA序列组装(贪婪算法)
生物信息学原理作业第四弹:DNA序列组装(贪婪算法) 原理:生物信息学(孙啸) 大致思想: 1. 找到权值最大的边: 2. 除去以最大权值边的起始顶点为起始顶点的边: 3. 除去以最大权值边为终点为终 ...
- DNA序列局部比对(Smith–Waterman algorithm)
生物信息原理作业第三弹:DNA序列局部比对,利用Smith–Waterman算法,python3.6代码实现. 实例以及原理均来自https://en.wikipedia.org/wiki/Smith ...
- 利用Needleman–Wunsch算法进行DNA序列全局比对
生物信息学原理作业第二弹:利用Needleman–Wunsch算法进行DNA序列全局比对. 具体原理:https://en.wikipedia.org/wiki/Needleman%E2%80%93W ...
- HDU 1560 DNA sequence(DNA序列)
HDU 1560 DNA sequence(DNA序列) Time Limit: 15000/5000 MS (Java/Others) Memory Limit: 32768/32768 K ...
随机推荐
- Knapsack I 竟然是贪心,证明啊。。。。
Knapsack I Time Limit: 2000/1000MS (Java/Others) Memory Limit: 128000/64000KB (Java/Others) SubmitSt ...
- 宝藏(树形DP)
这道题目是十分考验思维的,n^2应该还是比较好想的,主要是如何转移根的问题.转移根,在我看来应该是树形dp最难的一部分了, 一般学会如何转移根,也就差不多考验通吃树形dp了. 下面转一转大佬链接: ...
- JavaScript 实现发布消息后,距离当前时间的实现
某条消息发布后,距离当前时间多久的时间显示 //显示发布时间的函数 function pastTime(_createTime) { //var createTime = _createTime.su ...
- Java历程-初学篇 Day02变量,数据类型和运算符
一,数据类型 1,基础数据类型 整型 byte short int long 浮点型 float double 字符型 char 布尔类型 boolean 2,引用类型 String 字符串型 二,变 ...
- Python数据可视化利器Matplotlib,绘图入门篇,Pyplot介绍
Pyplot matplotlib.pyplot是一个命令型函数集合,它可以让我们像使用MATLAB一样使用matplotlib.pyplot中的每一个函数都会对画布图像作出相应的改变,如创建画布.在 ...
- HDU1421搬寝室(简单DP)
当然,还可以加滚动数组优化. #include<cstdio> #include<cstdlib> #include<iostream> #include<m ...
- EasyUI DataGrid使用示例
<%@ Page Language="C#" AutoEventWireup="true" CodeFile="EasyUIDemo.aspx. ...
- 初学者易上手的SSH-struts2 04值栈与ognl表达式
什么是值栈?struts2里面本身提供的一种存储机制,类似于域对象,值栈,可以存值和取值.,特点:先进后出.如果将它当做一个容器的话,而这个容器有两个元素,那么最上面的元素叫做栈顶元素,也就是所说的压 ...
- 微信公众号第三方 推送component_verify_ticket协议
整了一天,终于弄明白了 component_verify_ticket 怎么获取的了.在此先批一下微信公众号平台,文档又没写清楚,又没有客服,想搞哪样哈! 好,回归正题. 第一,先通过开发者资质认证, ...
- java将map拼接成“参数=值&参数=值”
Java将Map拼接成"参数=值&参数=值" 把一个map的键值对拼接成"参数=值&参数=值"即"username=angusbao& ...
