支持向量机SVM 参数选择
http://ju.outofmemory.cn/entry/119152
http://www.cnblogs.com/zhizhan/p/4412343.html
支持向量机SVM是从线性可分情况下的最优分类面提出的。所谓最优分类,就是要求分类线不但能够将两类无错误的分开,而且两类之间的分类间隔最大,前者是保证经验风险最小(为0),而通过后面的讨论我们看到,使分类间隔最大实际上就是使得推广性中的置信范围最小。推广到高维空间,最优分类线就成为最优分类面。
支持向量机是利用分类间隔的思想进行训练的,它依赖于对数据的预处理,即,在更高维的空间表达原始模式。通过适当的到一个足够高维的非线性映射  ,分别属于两类的原始数据就能够被一个超平面来分隔。如下图所示:
,分别属于两类的原始数据就能够被一个超平面来分隔。如下图所示:

空心点和实心点分别代表两个不同的类,H为将两类没有错误的区分开的分类面,同时,它也是一个最优的分类面。原因正如前面所述,当以H为分类面时,分类间隔最大,误差最小。而这里的  之间的距离margin就是两类之间的分类间隔。支持向量机将数据从原始空间映射到高维空间的目的就是找到一个最优的分类面从而使得分类间隔margin最大。而那些定义最优分类超平面的训练样本,也就是上图中过
之间的距离margin就是两类之间的分类间隔。支持向量机将数据从原始空间映射到高维空间的目的就是找到一个最优的分类面从而使得分类间隔margin最大。而那些定义最优分类超平面的训练样本,也就是上图中过  的空心点和实心点,就是支持向量机理论中所说的支持向量。显然,所谓支持向量其实就是最难被分类的那些向量,然而,从另一个角度来看,它们同时也是对求解分类任务最有价值的模式。
的空心点和实心点,就是支持向量机理论中所说的支持向量。显然,所谓支持向量其实就是最难被分类的那些向量,然而,从另一个角度来看,它们同时也是对求解分类任务最有价值的模式。
支持向量机的基本思想可以概括为:首先通过非线性变换将输入空间变换到一个高维空间,然后在这个新空间中求取最优线性分类面,而这种非线性变换是通过定义适当的内积函数来实现的。支持向量机求得的分类函数形式上类似于一个神经网络,其输出是若干中间层节点的线性组合,而每一个中间层节点对应于输入样本与一个支持向量的内积,因此也被叫做支持向量网络。如下图所示:

由于最终的判别函数中实际只包含于支持向量的内积和求和,因此判别分类的计算复杂度取决于支持向量的个数。
不难发现,支持向量机作为统计学习理论中的经典代表使用了与传统方法完全不同的思路,即不是像传统方法那样首先试图将原输入空间降维(即特征选择和特征变换),而是设法将输入空间升维,以求在高维空间中问题变得线性可分或接近线性可分。因为升维知识改变了内积运算,并没有使得算法的复杂性随着维数的增加而增加,而且在高维空间中的推广能力并不受到维数的影响。
另外,需要说明的是,支持向量机采用不同的内积函数,将导致不同的支持向量机算法
目前得到研究的内积函数主要有以下三类:
(1)采用多项式形式的内积函数;
(2)采用核函数形式的内积函数;
(3)采用S形函数作为内积函数;
libSVM是台湾大学林智仁教授等研究人员开发的一个用于支持向量机分类,回归分析及分布估计的c/c++开源库。另外,它也可以用于解决多类分类问题。 libSVM最新的版本是2011年4月发布的3.1版。林智仁教授设计开发该SVM库的目的是为了让其它非专业人士可以更加方便快捷的使用SVM这个统计学习工具。libSVM提供了一些简单易用的接口,从而使得用户可以方便的使用,而不必关心其内部复杂的数学模型和运算过程。libSVM的主要特点有:
(1)各种SVM的表达公式;
(2)有效的多类分类能力;
(3)交叉验证功能;
(4)各种核函数,包括预先计算得到的核矩阵;
(5)用于非平衡数据的加权svm;
(6)提供c++和java源代码;
(7)用于演示SVM分类与回归能力的GUI界面;
.....
很多初学者往往按照以下的步骤使用libSVM:
(1)将数据转换到libSVM指定的格式;
(2)随机选择一个核函数和一些参数;
(3)测试;
这种方法虽然可行,但却不一定能很快达到好的效果。为此,林智仁教授推荐按照以下的步骤来使用libSVM:
(1)将数据转换到libSVM指定的格式;
(2)对数据进行尺度操作(一般指数据的归一化);
(3)考虑RBF(径向基)核函数;
(4)利用交叉验证来得到最好的参数C和r;
(5)用最好的C和r来训练所有训练集合;
(6)测试;
之所以推荐首选径向基核函数,是由于该核可以将数据非线性地映射到高维空间,而且,它还能处理那种特征(数据)及其属性之间呈现非线性关系的情况,而线性核函数只是径向基核函数的一个特例。另外,相比而言,多项式核函数在高维空间有着更多的参数,从而使得模型更加复杂。同时,需要提醒的是,径向基核函数并非万能的,尤其当特征数据的数值本身比较大的时候,线性核函数要更实用一些。
任何人可以在http://www.csie.ntu.edu.tw/~cjlin/libsvm 来下载libSVM开源库。不过,按照开发者的要求,在使用之前,请务必阅读其copyright,并按照其要求进行相应的引用和说明。另外,在使用之前,强烈推荐大家阅读libSVM.zip里面的readme文件。该文件详细描述了libSVM的使用方法及注意事项。
libSVM介绍
鉴于libSVM中的readme文件有点长,而且,都是采用英文书写,这里,我把其中重要的内容提炼出来,并给出相应的例子来说明其用法,大家可以直接参考我的代码来调用libSVM库。
第一部分,利用libSVM自带的简易工具来演示SVM的两类分类过程。
(以下内容只是利用libSVM自带的一个简易的工具供大家更好的理解SVM,如果你对SVM已经有了一定的了解,可以直接跳过这部分内容)
首先,你要了解的是libSVM只是众多SVM实现版本中的其中之一。而SVM是一种进行两类分类的分类器,在libSVM最新版(libSVM3.1)里面,已经自带了简单的工具,可以对二分类进行演示。以windows平台为例,将libSVM.zip解压之后,有一个名为windows的子文件夹,里面有一个名为svm-toy.exe的可执行文件。直接双击,运行该可执行文件,显示如下的界面
点击第二个按钮“Run”,然后,在左上部分,用鼠标左键随机点几下,代表你选择的第一类模式的数据分布,下图是我随即点了几下的结果:

之后,点击“Change”,接着,用鼠标左键在窗口右下方随便点击几下,代表你选择的第二类模式的数据分布,如下图所示:
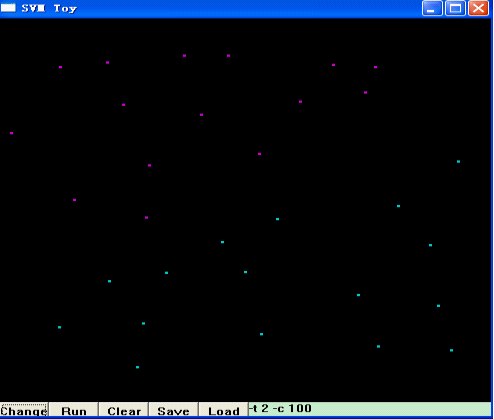
接着,点击“Run”,libSVM就帮你把这两类模式分开了,并用两种不同的颜色区域来代表两类不同的模式,如下图所示:

图中左上方紫色的区域,是第一类模式所在的区域,右下方的蓝色区域,是你选择的第二类模式所在的的区域,而两者的分界面,也就是SVM的最优分类面。当然,SVM是通过核函数将原始数据映射到高维空间,在高维空间进行线性分类。换句话说,在高维空间,这两类数据应该是线性可分的,即:最优分类面应该是一条直线,而这里看到的,是将高维空间分类的结果又映射回原始空间所呈现的分类结果,即:非线性的分类面。细心的朋友可能已经发现,在上述界面的右下角,有一个编辑框,里面写着“-t 2 -c 100”,显然,这是libSVM的一些参数,你也可以试着更改这些参数,来选择不同的核函数、不同的SVM类型等来达到最好的分类效果。
第二部分:libSVM中的小工具
libSVM中包含以下可执行程序文件(小工具):
(1)svm-scale:一个用于对输入数据进行归一化的简易工具
(2)svm-toy:一个带有图形界面的交互式SVM二分类功能演示小工具;
(3)svm-train:对用户输入的数据进行SVM训练。其中,训练数据是按照以下格式输入的:
<类别号> <索引1>:<特征值1> <索引2>:<特征值2>...
(4)svm-predict:根据SVM训练得到的模型,对输入数据进行预测,即分类。
第三部分:libSVM用法介绍:`
libSVM的所有函数申明及结构体定义均包含在libSVM.h文件当中,在使用过程中,你必须要包含该头文件,并且,对libSVM.cpp进行相应的链接。在对libSVM中的函数用法进行详细介绍之前,我们不妨先简单了解一下libSVM.h中一些结构体的含义。
struct svm_node
{
int index;
double value;
};该结构体,定义了一个“SVM节点”,即:索引i及其所对应的第i个特征值。这样n个相同类别号的SVM节点,就构成了一个SVM输入向量。即:一个SVM输入向量可以表示为如下的形式:类别标签 索引1:特征值1 索引2:特征值2 索引3:特征值3...我们可以将若干个这样的输入向量输入到libSVM进行训练,或者,输入一个类别标签未知的向量对其进行预测。struct svm_problem
{
int l;
double *y;
struct svm_node **x;
};该结构体中的l代表训练样本的个数;double型指针y代表l个训练样本中每个训练样本的类别号,也就是我们常说的“标签”;而"SVM节点"x,则是一个指针的指针(如果你对指针的指针不熟悉,完全可以把x理解为一个矩阵),x所指向的内容就是所有训练样本所有的特征值数据。
假如我们有下面的训练样本数据:
类别标签 特征值1 特征值2 特征值3 特征值4 特征值5
1 0 0.1 0.2 0 0
2 0 0.1 0.3 -1.2 0
1 0.4 0 0 0 0
2 0 0.1 0 1.4 0.5
1 -0.1 -0.2 0.1 1.1 0.1
那么,svm_problem结构体中的l=5(共有5个训练样本),y=[1,2,1,2,1];指针x所指向的内容可以视为5个行向量,每个行向量有5列,即:x指代一个5*5的矩阵,其值为:
(1,0)(2,0.1)(3,0.2)(4,0)(5,0)(-1,?)
(1,0)(2,0.1)(3,0.3)(4,-1.2)(5,0)(-1,?)
(1,0.4)(2,0)(3,0)(4,0)(5,0)(-1,?)
(1,0)(2,0.1)(3,0)(4,1.4)(5,0.5)(-1,?)
(1,-0.1)(2,-0.2)(3,0.1)(4,1.1)(5,0.1)(-1,?)
需要提醒的是,这里,每一行最后一列都是以“-1”开头,这是libSVM规定的特征值向量的结束标识;此外,索引应该按照升序方式进行排列。
enum { C_SVC, NU_SVC, ONE_CLASS, EPSILON_SVR, NU_SVR };//libSVM规定的SVM类型
enum { LINEAR, POLY, RBF, SIGMOID, PRECOMPUTED };//libSVM规定的核函数的类型
struct svm_parameter
{
int svm_type;//取值为前面提到的枚举类型中的值
int kernel_type;//取值为前面提到的枚举类型中的值
int degree; //用于多项式核函数/
double gamma;//用于多项式、径向基、S型核函数
double coef0;//用于多项式和S型核函数
/* 以下参数仅仅用于训练阶段 */
double cache_size; //核缓存大小,以MB为单位
double eps; //误差精度小于eps时,停止训练
double C; //用于C_SVC,EPSILON_SVR,NU_SVR
int nr_weight; //用于C_SVC
int *weight_label;//用于C_SVC
double* weight;//用于C_SVC
double nu;//用于NU_SVC,ONE_CLASS,NU_SVR
double p;//用于EPSILON_SVR
int shrinking; //等于1代表执行启发式收缩
int probability;//等于1代表模型的分布概率已知
};该结构体定义了libSVM中的用到的SVM参数。其中svm_type可以是C_SVC, NU_SVC, ONE_CLASS, EPSILON_SVR, NU_SVR中的任意一种,代表着SVM的类型;
C_SVC: C-SVM classification
NU_SVC: nu-SVM classification
ONE_CLASS: one-class-SVM
EPSILON_SVR: epsilon-SVM regression
NU_SVR: nu-SVM regression
kernel_type可以是LINEAR, POLY, RBF, SIGMOID中的一种,代表着核函数的类型;
LINEAR: u'*v,线性核函数;
POLY: (gamma*u'*v + coef0)^degree,多项式核函数;
RBF: exp(-gamma*|u-v|^2),径向基核函数;
SIGMOID: tanh(gamma*u'*v + coef0),S型核函数;
PRECOMPUTED: kernel values in training_set_file,自定义的核函数;
nr_weight, weight_label, and weight这三个参数用于改变某些类的惩罚因子。当输入数据不平衡,或者误分类的风险代价不对称的时候,这三个参数将会对样本训练起到非常重要的调节作用。
nr_weight是weight_label和weight的元素个数,或者称之为维数。Weight[i]与weight_label[i]之间是一一对应的,weight[i]代表着类别weight_label[i]的惩罚因子的系数是weight[i]。如果你不想设置惩罚因子,直接把nr_weight设置为0即可。为了防止错误的参数设置,你还可以调用libSVM提供的接口函数svm_check_parameter()来对输入参数进行检查。
在使用libSVM进行分类之前,你需要通过样本学习,构建一个SVM分类模型。该分类模型也可以理解为生成一些用于分类的“数据”。当然,构建的分类模型需要保存为文件,以便后续使用。用于libSVM训练的函数,其申明如下所示:
struct svm_model *svm_train(const struct svm_problem *prob, const struct svm_parameter *param);显然,该函数的输入,就是svm_problem结构体的prob指针所指向的内容。该结构体在前面已经介绍过,其内部,不仅包含了训练样本的个数,还包含每个训练样本的“标签”及该训练样本对应的特征数据。而svm_parameter类型的param指针则指定了libSVM所用到的诸如SVM类型,核函数类型,惩罚因子之类的参数。另外,该函数的返回值是一个svm_model结构体,该结构体的定义,在libSVM.cpp当中:
struct svm_model
{
svm_parameter param; //SVM参数设置
int nr_class; //类别数量,对于regression和ne-class SVM这两种情况,该值为2
int l; //支持向量的个数
svm_node **SV; //支持向量
double **sv_coef; //用于决策函数的支持向量系数
double *rho; //决策函数中的常数项
double *probA; // pariwise probability information
double *probB;
// for classification only
int *label; // 每个类类别标签
int *nSV; //每个类的支持向量个数
int free_sv; //如果svm_model已经通过svm_load_model创建,则该值为1;如果svm_model是通过svm_train创建的,该值为0
};需要提醒的是,libSVM支持多类分类问题,当有k个待分类问题时,libSVM构建k*(k-1)/2种分类模型来进行分类,即:libSVM采用一对一的方式来构建多类分类器,如下所示:
1 vs 2, 1 vs 3, ..., 1 vs k, 2 vs 3, ..., 2 vs k, ..., k-1 vs k。
用户在得到SVM分类模型之后,需要将其进行保存。在这里,libSVM已经提供了相应的函数接口:
int svm_save_model(const char *model_file_name, const struct svm_model *model);在调用训练函数之后,只需要指定保存位置,直接调用该函数,就可以进行相应的保存。
在对样本进行训练得到分类模型之后,就可以利用该分类模型对未知输入数据进行类别判断了,也就是我们常说的“预测”。用于libSVM预测的函数,其申明如下所示:
double svm_predict(const struct svm_model *model, const struct svm_node *x);该函数的第一个参数就是利用样本训练得到的SVM分类模型,第二个参数,是输入的未知模式的特征数据,即:得到了表征某一类别的特征数据,根据这些数据,来判断它所对应的类别标签。而SVM分类模型,可以由libSVM定义的下面这个接口函数来进行加载:
struct svm_model *svm_load_model(const char *model_file_name);
此外,在使用上述函数过程中,需要对svm_model及svm_parameter申请内存,而不使用它们的时候,用户需要调用以下两个函数进行内存释放:
void svm_destroy_model(struct svm_model *model);
void svm_destroy_param(struct svm_parameter *param);前面提到,很多人看到libSVM这么多的参数,估计要犯晕了。没关系,我之前把相关的libSVM参数已经讲解了一遍,这里,再给出libSVM的用法。如果你不想花时间去仔细研究libSVM,完全可以参照我的函数来直接调用libSVM完成你的工作。
首先是训练SVM得到模型;假设,有10个训练样本,每个训练样本,有12个特征值,即:每个训练样本的维数是12,也就是说,训练样本构成了一个10*12的矩阵(当然,所有数据应该被归一化到[-1,1]或[0,1]范围内),另外,所有训练样本,应该有一个明确的类别标签,以此来表明当前样本所属的类别。所以,完整的训练样本,应该是10*13的矩阵,这里,我们给出一个10*13的矩阵,并认为,它就是训练样本。每个训练样本是个行向量,而该矩阵的第一列则代表训练样本的“标签”。即:第一个训练样本,类别标签为“1”,第二个训练样本类别标签为“-1”。第一个训练样本的第一个特征值为0.708333,第二个特征值为1,第三个特征值为1...
double inputArr[10][13] =
{
,0.708333,1,1,-0.320755,-0.105023,-1,1,-0.419847,-1,-0.225806,0,1,
-1,0.583333,-1,0.333333,-0.603774,1,-1,1,0.358779,-1,-0.483871,0,-1,
,0.166667,1,-0.333333,-0.433962,-0.383562,-1,-1,0.0687023,-1,-0.903226,-1,-1,
-1,0.458333,1,1,-0.358491,-0.374429,-1,-1,-0.480916,1,-0.935484,0,-0.333333,
-1,0.875,-1,-0.333333,-0.509434,-0.347032,-1,1,-0.236641,1,-0.935484,-1,-0.333333,
-1,0.5,1,1,-0.509434,-0.767123,-1,-1,0.0534351,-1,-0.870968,-1,-1,
,0.125,1,0.333333,-0.320755,-0.406393,1,1,0.0839695,1,-0.806452,0,-0.333333,
,0.25,1,1,-0.698113,-0.484018,-1,1,0.0839695,1,-0.612903,0,-0.333333,
,0.291667,1,1,-0.132075,-0.237443,-1,1,0.51145,-1,-0.612903,0,0.333333,
,0.416667,-1,1,0.0566038,0.283105,-1,1,0.267176,-1,0.290323,0,1
};另外,我们给出一个待分类的输入向量,并用行向量形式表示:
double testArr[]=
{
.25,1,1,-0.226415,-0.506849,-1,-1,0.374046,-1,-0.83871,0,-1
};下面,给出完整的SVM训练及预测程序:
#include "stdafx.h"
#include "svm.h"
#include "iostream"
#include "fstream"
using namespace std;
double inputArr[10][13] =
{
, 0.708333, 1, 1, -0.320755, -0.105023, -1, 1, -0.419847, -1, -0.225806, 0, 1,
-1, 0.583333, -1, 0.333333, -0.603774, 1, -1, 1, 0.358779, -1, -0.483871, 0, -1,
, 0.166667, 1, -0.333333, -0.433962, -0.383562, -1, -1, 0.0687023, -1, -0.903226, -1, -1,
-1, 0.458333, 1, 1, -0.358491, -0.374429, -1, -1, -0.480916, 1, -0.935484, 0, -0.333333,
-1, 0.875, -1, -0.333333, -0.509434, -0.347032, -1, 1, -0.236641, 1, -0.935484, -1, -0.333333,
-1, 0.5, 1, 1, -0.509434, -0.767123, -1, -1, 0.0534351, -1, -0.870968, -1, -1,
, 0.125, 1, 0.333333, -0.320755, -0.406393, 1, 1, 0.0839695, 1, -0.806452, 0, -0.333333,
, 0.25, 1, 1, -0.698113, -0.484018, -1, 1, 0.0839695, 1, -0.612903, 0, -0.333333,
, 0.291667, 1, 1, -0.132075, -0.237443, -1, 1, 0.51145, -1, -0.612903, 0, 0.333333,
, 0.416667, -1, 1, 0.0566038, 0.283105, -1, 1, 0.267176, -1, 0.290323, 0, 1
};
double testArr[] =
{
.25, 1, 1, -0.226415, -0.506849,-1,-1,0.374046,-1,-0.83871,0,-1};voidDefaultSvmParam(struct svm_parameter *param ){
param->svm_type = C_SVC;
param->kernel_type = RBF;
param->degree =3;
param->gamma =0;/* 1/num_features */
param->coef0 =0;
param->nu =0.5;
param->cache_size =100;
param->C =1;
param->eps =1e-3;
param->p =0.1;
param->shrinking =1;
param->probability =0;
param->nr_weight =0;
param->weight_label = NULL;
param->weight = NULL;}voidSwitchForSvmParma(struct svm_parameter *param,char ch,char*strNum,int nr_fold,int cross_validation ){switch( ch ){case's':{
param->svm_type = atoi( strNum );break;}case't':{
param->kernel_type = atoi( strNum );break;}case'd':{
param->degree = atoi( strNum );break;}case'g':{
param->gamma = atof( strNum );break;}case'r':{
param->coef0 = atof( strNum );break;}case'n':{
param->nu = atof( strNum );break;}case'm':{
param->cache_size = atof( strNum );break;}case'c':{
param->C = atof( strNum );break;}case'e':{
param->eps = atof( strNum );break;}case'p':{
param->p = atof( strNum );break;}case'h':{
param->shrinking = atoi( strNum );break;}case'b':{
param->probability = atoi( strNum );break;}case'q':{break;}case'v':{
cross_validation =1;
nr_fold = atoi( strNum );if( nr_fold <2){
cout <<"nr_fold should > 2!!! file: "<< __FILE__ <<" function: ";
cout << __FUNCTION__ <<" line: "<< __LINE__ << endl;}break;}case'w':{++param->nr_weight;
param->weight_label =(int*) realloc( param->weight_label,sizeof(int)* param->nr_weight );
param->weight =(double*) realloc( param->weight,sizeof(double)* param->nr_weight );
param->weight_label[param->nr_weight -1]= atoi( strNum );
param->weight[param->nr_weight -1]= atof( strNum );break;}default:{break;}}}voidSetSvmParam(struct svm_parameter *param,char*str,int cross_validation,int nr_fold ){DefaultSvmParam( param );
cross_validation =0;char ch =' ';int strSize = strlen( str );for(int i =0; i < strSize; i++){if( str[i]=='-'){
ch = str[i +1];int length =0;for(int j = i +3; j < strSize; j++){if( isdigit( str[j])){
length++;}else{break;}}char*strNum =newchar[length +1];int index =0;for(int j = i +3; j < i +3+ length; j++){
strNum[index]= str[j];
index++;}
strNum[length]='/0';SwitchForSvmParma( param, ch, strNum, nr_fold, cross_validation );delete strNum;}}}voidSvmTraining(char*option ){struct svm_parameter param;struct svm_problem prob;struct svm_model *model;struct svm_node *x_space;int cross_validation =0;int nr_fold =0;int sampleCount =10;int featureDim =12;
prob.l = sampleCount;
prob.y =newdouble[sampleCount];
prob.x =newstruct svm_node*[sampleCount];
x_space =newstruct svm_node[(featureDim +1)* sampleCount];SetSvmParam(¶m, option, cross_validation, nr_fold );for(int i =0; i < sampleCount; i++){
prob.y[i]= inputArr[i][0];}int j =0;for(int i =0; i < sampleCount; i++){
prob.x[i]=&x_space[j];for(int k =1; k <= featureDim; k++){
x_space[i * featureDim + k].index = k;
x_space[i * featureDim + k].value = inputArr[i][k];}
x_space[(i +1)* featureDim].index =-1;
j =(i +1)* featureDim +1;}
model = svm_train(&prob,¶m );constchar* model_file_name ="C://Model.txt";
svm_save_model( model_file_name, model );
svm_destroy_model( model );
svm_destroy_param(¶m );delete[] prob.y;delete[] prob.x;delete[] x_space;}intSvmPredict(constchar* modelAdd ){struct svm_node *testX;struct svm_model* testModel;
testModel = svm_load_model( modelAdd );int featureDim =12;
testX =newstruct svm_node[featureDim +1];for(int i =0; i < featureDim; i++){
testX[i].index = i +1;
testX[i].value = testArr[i];}
testX[featureDim].index =-1;double p = svm_predict( testModel, testX );
svm_destroy_model( testModel );delete[] testX;if( p >0.5){return(1);}else{return(-1);}}int _tmain(int argc, _TCHAR* argv[]){SvmTraining("-c 100 -t 1 -g 4 -r 1 -d 4");int flag =SvmPredict("c://model.txt");
cout <<"flag = "<< flag << endl;
system("pause");return(0);}以上文章来源: http://blog.csdn.net/carson2005/article/details/6539218
LibSVM使用指南
本文包含以下几个部分:
- 支持向量机--SVM简介
- LibSVM的安装
- LibSVM的使用
- LibSVM参数调优
- Java版LibSVM库函数的调用
SVM简介
在进行下面的内容时我们认为你已经具备了数据挖掘的基础知识。
SVM是新近出现的强大的数据挖掘工具,它在文本分类、手写文字识别、图像分类、生物序列分析等实际应用中表现出非常好的性能。SVM属于监督学习算法,样本以属性向量的形式提供,所以输入空间是R n 的子集。

图1
如图1所示,SVM的目标是找到两个间距尽可能大的边界平面来把样本本点分开,以”最小化泛化误差“,即对新的样本点进行分类预测时,出错的几率最小。落在边界平面上的点称为支持向量。Vapnik证明如果可以找到一个较小的支持向量集,就可以保证得到很好的泛化能力----也就是说支持向量的个数越少越好。
数据点通常在输入空间是线性不可分的,我们把它映射到更高维的特征空间,使其线性可分----这里的映射函数我们称之为核函数。特征空间的超平面对应输入空间的一个非线性的分离曲面,因此称为非线性分离器。
线性SVM分类器的输出是u=w*x-b。w是分类平面的法矢,x是输入向量,b是常量,u代表分类。即SVM的目的就是计算出w和b。最大化margin(两个分类平面之间的距离)等价于求下面的二次优化问题:

对于非线性分类器就要把x映射到特征空间,同时考虑误差ε的存在(即有些样本点会越过分类边界),上述优化问题变为:

从输入空间是映射到特征空间的函数称为核函数,LibSVM中使用的默认核函数是RBF(径向基函数radial basis function),即

这样一来就有两个参数需要用户指定:c和gamma。实际上在LibSVM中用户需要给出一个c和gamma的区间,LibSVM采用交叉验证cross-validation accuracy的方法确定分类效果最好的c和gamma。
举个例子说明什么是交叉验证,假如把训练样本集拆成三组,然后先拿 1 跟 2 来 train model 并 predict 3 以得到正确率; 再来拿 2 跟 3 train 并 predict 1 ,最后 1,3 train 并 predict 2 ,最后取预测精度最高的那组c和gamma。
有时属于不同分类的样本数是不平衡的,所以有人提出QP(二次优化)的目标函数应该为:

LibSVM中允许用户指定权重值C+和C-。
对于文本分类采用最简单的线性分类器即可,因为输入的文档向量矩阵高度稀疏,可以认为不需要映射到特征空间,在输入空间就线性可分, 这样我们就不需要使用核函数了----然而我的实践证明这个结论并不总是正确的。
LibSVM的安装
- Linux上下载 tar.gz包
- 解压后就可直接使用java版的了
但要求电脑上装有1.5版本以上的java,并且设置好了$classpath全局变量。
LibSVM的使用
我们只讲Linux下Java版的使用,在有的VM上,java版的libsvm运行速度可逼近C++版的运行速度。
cd /path/to/libsvm-3.1/java
java -classpath libsvm.jar svm_scale <arguments>
java -classpath libsvm.jar svm_train <arguments>
java -classpath libsvm.jar svm_predict <arguments>
java -classpath libsvm.jar svm_toy
LibSVM要求处理的文件数据都满足如下格式:
rlabel1 index1:value1 index2:value2 …...
rlabel2 index1:value1 index2:value2 …...
下面的脚本是我用于转换成LibSVM要求的文件格式用的,作个备份:
|
libsvm在存储中存储数据时默认采用的是float,而不是double。当你原始数据精度要求很高时这确实是个问题。
rlabel表示分类,为一个数字。Index从1开始递增,表示输入向量的序号,value是输入向量相应维度上的值,如果value为0,该项可以不写。下面是一个示例文件:
0 1:3.2 3:1.6
1 1:1.5 2:4.2 3:0.5
0 1:5.1 2:1.6 3:2.0
1 2:5.4
svm_scale用于把输入向量按列进行规范化(或曰缩放)。
Usage: svm-scale [options] data_filename
options:
-l lower : x scaling lower limit (default -1)
-u upper : x scaling upper limit (default +1)
-y y_lower y_upper : y scaling limits (default: no y scaling)
-s save_filename : save scaling parameters to save_filename
-r restore_filename : restore scaling parameters from restore_filename
举个例子,比如我运行:java svm_scale -l 0 -u 1 -s range train > train.scale则输入文件是train,输出文件是 train.scale,把输入向量的各列都缩放到[0,1]的范围内,range文件中保存了相关的缩放信息。
Train文件原来的内容:
1 1:0 2:0
1 1:3 2:4
1 1:5 2:9
1 1:12 2:1
1 1:8 2:7
0 1:9 2:8
0 1:6 2:12
0 1:10 2:8
0 1:8 2:5
0 1:14 2:8
range文件自动生成:
x
0.000000000000000 1.000000000000000
1 0.000000000000000 14.00000000000000
2 0.000000000000000 12.00000000000000
train.scale文件自动生成:
1.0
1.0 1:0.21428571428571427 2:0.3333333333333333
1.0 1:0.35714285714285715 2:0.75
1.0 1:0.8571428571428571 2:0.08333333333333333
1.0 1:0.5714285714285714 2:0.5833333333333334
0.0 1:0.6428571428571429 2:0.6666666666666666
0.0 1:0.42857142857142855 2:1.0
0.0 1:0.7142857142857143 2:0.6666666666666666
0.0 1:0.5714285714285714 2:0.4166666666666667
0.0 1:1.0 2:0.6666666666666666
然后我再运行:java svm_scale -r range test > test.scale意思是说从range文件中读取缩放信息运用于test文件,输出test.scale文件。
向量规范化后我们train一下训练样本,以生成支持向量。
运行:java svm_train -s 0 -c 5 -t 2 -g 0.5 -e 0.01 train.scale
对于文本分类svm_train中有几个选项会用到:
-s svm_type : set type of SVM (default 0)
0 -- C-SVC
-t kernel_type : set type of kernel function (default 2)
0 -- linear: u'*v
2 -- radial basis function: exp(-gamma*|u-v|^2)
-g gamma : set gamma in kernel function (default 1/num_features) num_features是输入向量的个数
-c cost : set the parameter C of C-SVC, epsilon-SVR, and nu-SVR (default 1)
-m cachesize : set cache memory size in MB (default 100) 使用多少内存
-e epsilon : set tolerance of termination criterion (default 0.001)
-h shrinking : whether to use the shrinking heuristics, 0 or 1 (default 1)
-wi weight : set the parameter C of class i to weight*C, for C-SVC (default 1) 当各类数量不均衡时为每个类分别指定C
-v n: n-fold cross validation mode交叉验证时分为多少组
-q : quiet mode (no outputs)
会生成train.scale.model文件,内容如下:
svm_type c_svc
kernel_type rbf
gamma 0.5
nr_class 2
total_sv 9
rho -0.5061570424019811
label 1 0
nr_sv 4 5
SV
2.7686973549711875 1:0.21428571428571427 2:0.3333333333333333
5.0 1:0.35714285714285715 2:0.75
5.0 1:0.8571428571428571 2:0.08333333333333333
5.0 1:0.5714285714285714 2:0.5833333333333334
-5.0 1:0.6428571428571429 2:0.6666666666666666
-2.4351637665059895 1:0.42857142857142855 2:1.0
-5.0 1:0.7142857142857143 2:0.6666666666666666
-5.0 1:0.5714285714285714 2:0.4166666666666667
-0.3335335884651968 1:1.0 2:0.6666666666666666
nr_class代表训练样本集有几类,rho是判决函数的常数项b,nr_sv是各个类中落在边界上的向量个数,SV下面枚举了所有的支持向量,每个支持向量前面都有一个数字,代表什么我现在也不清楚。
当train C-SVM时会有类似下面的输出:
optimization finished, #iter = 219
nu = 0.431030
obj = -100.877286, rho = 0.424632
nSV = 132, nBSV = 107
Total nSV = 132
obj是对SSVM问题的优化目标函数的值。rho是决策函数中的常数项b。nSV是支持向量的个数,nBSV是边界支持向量的个数(i.e., alpha_i = C)。
如果“自由支持向量”个数很多,很可能是因为过拟合了。如果输入数据的attribute在一个很大的范围内分布,最好scale一下。
采用时默认的核函数RBF是比较好的,if RBF is used with model selection, then there is no need to consider the linear kernel.
如果预测的准确率太低,如何提高一下?使用python目录下的grid.py进行模型选择以找到比较好的参数。
grid.py是一种用于RBF核函数的C-SVM分类的参数选择程序。用户只需给定参数的一个范围,grid.py采用交叉验证的方法计算每种参数组合的准确度来找到最好的参数。
Usage: grid.py [-log2c begin,end,step] [-log2g begin,end,step] [-v fold]
[-svmtrain pathname] [-gnuplot pathname] [-out pathname] [-png pathname]
[additional parameters for svm-train] dataset
The program conducts v-fold cross validation using parameter C (and gamma)= 2^begin, 2^(begin+step), ..., 2^end.
首先sudo apt-get install gnuplot
然后编译C++版本的LibSVM,生成svm-train二进制可执行文件。
cd /path/to/libsvm-3.1
make
举个例子就都明白了:python grid.py -log2c -5,5,1 -log2g -4,0,1 -v 5 -svmtrain /home/orisun/develop/libsvm-3.1/svm-train -m 500 traincev_svmfmt_part1
-m 500是使用svm_train时可以使用的参数。
最后输出两个文件:dataset.png绘出了交叉验证精度的轮廓图,dataset.out对于每一组log2(c)和log2(gamma)对应的CV精度值。
如果训练时间过长,你可能需要:
1.指定更大有cache size。(-m)
2.使用更宽松的stopping tolerance。(-e)
当使用一个很大有-e时,你可能需要检查一下-h 0 (no shrinking) or -h 1 (shrinking)哪个更快。
3.如果上面的方法还不行就需要裁剪训练集。使用tools目录下的subset.py来随机获得训练集的一个子集。
Usage: subset.py [options] dataset number [output1] [output2]
This script selects a subset of the given data set.
options:
-s method : method of selection (default 0)
0 -- stratified selection (classification only)
1 -- random selection
output1 : the subset (optional)
output2 : the rest of data (optional)
If output1 is omitted, the subset will be printed on the screen.
当迭代次数很高时使用shrinking是有帮助的,而当使用一个很大的-e时,迭代次数会减少,最好把shrinking关掉。
当指定一个很大-m时Linux会报"段错误“,很可能是内存溢出了。对于32位的机子最大的可编址内存是4G。同时Linux系统按照3:1来划分用户空间:核空间,所以用户空间只有最大只有3G,而可动态分配的内存最大只有2G。当你使用一个接近2G的-m时内存就会耗尽。
解决办法:
1.换64位的机子。
2.如果你的机子支持Intel's PAE (Physical Address Extension),你可以在Linux内核中开启HIGHMEM64G选项,这样核空间和用户空间的划分就是4G:4G。
3.安装"tub”软件,它可以消除动态分配内存只有2G的限制。tub可以在这里获取 http://www.bitwagon.com/tub.html。
在svm_train的过程中如果不想看到中间输出可以使用-q选项。
如果你是在编程代码中使用libsvm库,可以这样:
1.声明一个空的输出函数:void print_null(const char *s) {}
2.把它赋给libsvm库中的输出函数:svm_print_string = &print_null;
在处理多类分类问题时,libsvm采用的是one-against-one,而不是one-against-the rest,实际上后者的性能要好,而之所以采用前者仅仅是因为它快。
交叉验证是为了选择更好的参数,做完交叉验证后并不会输出model文件,此时你需要re-train the whole data without the -v option。
如果你有多核/共享内存的计算机,libsvm还允许你采用OpenMP进行并行编程。
预测时如果开启-b则会耗费更长的时间,并且开启-b和提高预测的准确率并没有绝对的关系。
最后可以预测分类了,运行:java svm_predict test.scale train.scale.model result
test.scale 是待预测的文件, train.scale.model是利用训练文本集生成的model文件,最终会生成result文件,内容如下:
1.0
1.0
0.0
1.0
0.0
0.0
0.0
0.0
1.0
0.0
由于在 test.scale中我已标记了正确的rlable,所以 svm_predict还会报告正确率Accuracy = 70.0% (7/10) (classification)。在实际的分类问题中,我们当然是无法提前知道待分类文件中的rlabel中,可以任意标记一个数字,这时候还会给出Accuracy ,不过它是毫无意义的。
在使用svm_toy时只支持3种颜色,最大分类数是3。如果想有更多分类,需要修改原代码svm-toy.cpp。如果直接从文件中load数据,要求向量是2维的,并且每一维都在(0,1),同时rlabel只能是1、2、3(甚至不能是1.0、2.0、3.0)。
下面是使用svn_toy的一个截图:

图2
LibSVM库函数的调用
库函数在"libsvm"包中。
在Java版中以下函数可以调用:
public class svm {
public static final int LIBSVM_VERSION=310;
public static svm_model svm_train(svm_problem prob, svm_parameter param);
public static void svm_cross_validation(svm_problem prob, svm_parameter param, int nr_fold, double[] target);
public static int svm_get_svm_type(svm_model model);
public static int svm_get_nr_class(svm_model model);
public static void svm_get_labels(svm_model model, int[] label);
public static double svm_get_svr_probability(svm_model model);
public static double svm_predict_values(svm_model model, svm_node[] x, double[] dec_values);
public static double svm_predict(svm_model model, svm_node[] x);
public static double svm_predict_probability(svm_model model, svm_node[] x, double[] prob_estimates);
public static void svm_save_model(String model_file_name, svm_model model) throws IOException
public static svm_model svm_load_model(String model_file_name) throws IOException
public static String svm_check_parameter(svm_problem prob, svm_parameter param);
public static int svm_check_probability_model(svm_model model);
public static void svm_set_print_string_function(svm_print_interface print_func);
}
注意在Java版中svm_node[]的最后一个元素的索引不是-1.
用户可以自定义自己的输出格式,通过:
your_print_func = new svm_print_interface()
{
public void print(String s)
{
// your own format
}
};
svm.svm_set_print_string_function(your_print_func);
文章来源: http://www.cnblogs.com/zhangchaoyang/articles/2189606.html
LIBSVM回归详细操作步骤
用gridregression.py搜索最优参数的方法如下:
python gridregression.py -svmtrain “F:\Programing\Matlab\libsvm\windows\svm-train.exe” -gnuplot “C:\Program Files\gnuplot\bin\gnuplot.exe” -log2c -10,10,1 -log2g -10,10,1 -log2p -10,10,1 -v 10 -s 3 -t 2 list.scale > gridregression_feature.parameter
python gridregression.py -svmtrain ../svm-train -gnuplot /usr/local/bin/gnuplot -log2c -10,10,1 -log2g -10,10,1 -log2p -10,10,1 -v 10 -s 3 -t 2 list.scale > gridregression_feature.parameter
注意:-svmtrain是给出svmtrain.exe所在路径,一定要是完整的全路径-gnuplot是给出pgnuplot.exe所在路径。这里要用pgnuplot.exe这种命令行形式的,不要用wgnupl32.exe,这个是图形界面的。
-log2c是给出参数c的范围和步长
-log2g是给出参数g的范围和步长
-log2p是给出参数p的范围和步长
上面三个参数可以用默认范围和步长
-s选择SVM类型,也是只能选3或者4
-t是选择核函数
-v 10 将训练数据分成10份做交叉验证,默认为5。
其实只要修改一个文件(gridregression.py)的路径就可以了,其他网上说的两个文件(grid.py和easy.py)的路径可以不做修改,因为回归根本没有用到。修改的地方是绿色的两行路径,写成实际路径就可以了。网上下载下来的一般都是r”…svm-…”所以要改,修改后如下图。
改完之后,首先把你的数据集包括data2和test2(这是原始的)放到C:\libsvm-2.88\windows下。
现在要做的就是真正意义上的第一步,归一化处理,这一步要做,希望不要为了简便不做,这样预测出来不准。具体在dos下调完路径后,执行下面两句,分别是归一化数据集和测试集后产生新的两个文件data和test。
至于路径的问题,很多不涉及计算机专业的网友很痛苦,经常会不知所措。不过我再多嘴一次:先输入字母cd,大小写都无所谓了,然后空格,之后就是你想要让dos去运行的地址(路径)。可以老老实实的手打,也可以复制C:\libsvm-2.88\windows,然后粘贴,dos框下Ctrl+V无效,只能鼠标右击选择粘帖。最后按一回车键就把路径调到C:\libsvm-2.88\windows下了。
现在C:\libsvm-2.88\windows中多了两个文件,其实还有一个scale,不用管他,不起作用!
现在是通过gridregression.py函数进行参数寻优,把路径调好,注意调到C:\Python25下了。输入下面的语句python C:\libsvm-2.88\python\gridregression.py -svmtrain C:\libsvm-2.88\windows\svm-train.exe -gnuplot C:\gnuplot\bin\pgnuplot.exe -log2c -10,10,1 -log2g -10,10,1 -log2p -10,10,1 -v 10 -s 3 -t 2 C:\libsvm-2.88\windows\data.txt > gridregression_data.parameter,可能你要等很长时间,我的数据很多搞了一个晚上。
有天早上一个朋友说你昨晚这么晚回来,早上怎么7点就上线了啊qq,汗,电脑一夜跑这玩意儿。。。扯远了。
PS.命令很长可以选择上述方法复制后,在dos框中右击粘贴完成。当然有热心的新浪网友的建议:(这么长的命令在dos下敲很容易出错的,建议大家在相应目录下建立一个bat文件,如“py.bat",把相应命令拷贝过去,直接运行"py",这样就省事多了。希望博主把这方法加到你的博文里。),我还没有试过。
训练完后,在C:\Python25中会有一个gridregression_data.parameter文件,里面就是自动寻优的结果,主要也是为了这个东西,搞了我老半天nnd。
打开它,下面最后一行分别为c,g,p,mse。其中mse没有用,其实这个值越小越好。
P.S. 有网友回复说:data训练时的mse=88.1545,而test测试时mse=401.938(他的数据结果),其实data训练的mse不是越小越好,过小以后,会形成过学习情况。其实严格意义上说是test的mse越小越好,因为它越小,最终的预测精度越高。另外,还可以通过squared correlation coefficient判断,它越接近1,也说明预测的精度越高。这是我做过many数据试验的吐血结果,望与大家分享。
把刚才的3个参数用来进行训练svm模型。把路径重新调回C:\libsvm-2.88\windows,输入语句训练,会在C:\libsvm-2.88\windows里产生一个data.txt.model文件。训练好了紧接着对test文件预测,输入第二条语句,得出结果在out里面。
最后打开out和test文件比较一下结果差多少,自己去计算咯。
到此已经实现了libsvm软件做回归预测的全过程,个人认为已经很详尽,比网上任何帖子都清楚哈哈。哪里不清楚的希望大家一起讨论~~
支持向量机SVM 参数选择的更多相关文章
- [吴恩达机器学习笔记]12支持向量机5SVM参数细节
12.支持向量机 觉得有用的话,欢迎一起讨论相互学习~Follow Me 参考资料 斯坦福大学 2014 机器学习教程中文笔记 by 黄海广 12.5 SVM参数细节 标记点选取 标记点(landma ...
- 支持向量机(SVM)利用网格搜索和交叉验证进行参数选择
上一回有个读者问我:回归模型与分类模型的区别在哪?有什么不同,我在这里给他回答一下 : : : : 回归问题通常是用来预测一个值,如预测房价.未来的天气情况等等,例如一个产品的实际价格为500元,通过 ...
- libsvm的安装,数据格式,常见错误,grid.py参数选择,c-SVC过程,libsvm参数解释,svm训练数据,libsvm的使用详解,SVM核函数的选择
直接conda install libsvm安装的不完整,缺几个.py文件. 第一种安装方法: 下载:http://www.csie.ntu.edu.tw/~cjlin/cgi-bin/libsvm. ...
- 【IUML】支持向量机SVM
从1995年Vapnik等人提出一种机器学习的新方法支持向量机(SVM)之后,支持向量机成为继人工神经网络之后又一研究热点,国内外研究都很多.支持向量机方法是建立在统计学习理论的VC维理论和结构风险最 ...
- [转]支持向量机SVM总结
首先,对于支持向量机(SVM)的简单总结: 1. Maximum Margin Classifier 2. Lagrange Duality 3. Support Vector 4. Kernel 5 ...
- 机器学习:Python中如何使用支持向量机(SVM)算法
(简单介绍一下支持向量机,详细介绍尤其是算法过程可以查阅其他资) 在机器学习领域,支持向量机SVM(Support Vector Machine)是一个有监督的学习模型,通常用来进行模式识别.分类(异 ...
- libsvm参数选择
以前接触过libsvm,现在算在实际的应用中学习 LIBSVM 使用的一般步骤是: 1)按照LIBSVM软件包所要求的格式准备数据集: 2)对数据进行简单的缩放操作: 3)首要考虑选用RBF 核函数: ...
- 以图像分割为例浅谈支持向量机(SVM)
1. 什么是支持向量机? 在机器学习中,分类问题是一种非常常见也非常重要的问题.常见的分类方法有决策树.聚类方法.贝叶斯分类等等.举一个常见的分类的例子.如下图1所示,在平面直角坐标系中,有一些点 ...
- 机器学习之支持向量机—SVM原理代码实现
支持向量机—SVM原理代码实现 本文系作者原创,转载请注明出处:https://www.cnblogs.com/further-further-further/p/9596898.html 1. 解决 ...
随机推荐
- # 20165225 《Java程序设计》第一周学习总结
20165225 <Java程序设计>第一周学习总结 1.视频与课本中的学习: 首先是为了运行和开发Java分别安装了JRE和JDK,具体做法在老师给的<Java2 实用教程(第五版 ...
- 使用axios加入进度条
思路:(安慰剂按钮)首先当触发按钮时,设置拦截器,启动进度条从0开始到100满(html进度条用数值value来控制,默认为0),设置进度条的配置函数然后在后端返回函数中启动停止精度条的函数,为了保持 ...
- 微信6.7.4 ios12 软键盘收回时页面不回弹,导致光标位置错乱,再次点击输入框区域时无法focus
https://developers.weixin.qq.com/community/develop/doc/00044ae90742f8c82fb78fcae56800 https://blog.c ...
- Orchard Core 版本冲突 The type 'FormTagHelper' exists in both 'Microsoft.AspNetCore.Mvc.TagHelpers, Version=2.1.1.0, Culture=neutral, PublicKeyToken=adb9793829ddae60' and...
最近老大让我看Orchard Core,这是一个CMS系统.可以先参考大佬的文章:https://www.cnblogs.com/shanyou/archive/2018/09/25/9700422. ...
- 洛谷P3121 审查(黄金)Censoring(Gold) [USACO15FEB] AC自动机
正解:AC自动机 解题报告: 传送门! 啊我好呆啊其实就挺模板题的,,,只是要一个栈搞一下,,,然后我就不会了,,,是看了题解才get的,,,QAQ 然后写下解法趴QwQ 首先看到多串匹配不难想到AC ...
- 004-Quartz存储与持久化-基于quartz.properties的配置
一.概述 Quartz提供两种基本作业存储类型.第一种类型叫做RAMJobStore,第二种类型叫做JDBC作业存储.在默认情况下Quartz将任务调度的运行信息保存在内存中,这种方法提供了最佳的性能 ...
- MYSQL: set names utf8是什么意思?
set names utf8 是用于设置编码,可以再在建数据库的时候设置,也可以在创建表的时候设置,或只是对部分字段进行设置,而且在设置编码的时候,这些地方最好是一致的,这样能最大程度上避免数据记录出 ...
- react-router v4 使用 history 控制路由跳转
问题 当我们使用react-router v3的时候,我们想跳转路由,我们一般这样处理 我们从react-router导出browserHistory. 我们使用browserHistory.push ...
- 适用于 Windows 7 SP1、Windows Server 2008 R2 SP1 和 Windows Server 2008 SP2 的 .NET Framework 4.5.2 仅安全更新说明:2017 年 9 月 12 日
https://support.microsoft.com/zh-cn/help/4040960/description-of-the-security-only-update-for-the-net ...
- eclipse签名使用的key文件(android生成keystore)
命令行(或终端)生成keystore文件 在命令行(或终端)输入命令: keytool -genkey -alias Gallery.keystore -keyalg RSA -validit ...
