(原创)遗传算法C++实现
本文没有对遗传算法的原理做过多的解释 基础知识可以参考下面的博客:
http://blog.csdn.net/u010451580/article/details/51178225
本实验用到的变异用到下面网址上的方法,当然这个网址也很好的阐释了CVRP的解决方案:
https://image.hanspub.org/Html/10-2620135_14395.htm
本文所用交叉算法是部分交叉映射PMX,PMX基础知识请参考这个博客:
http://blog.csdn.net/u012750702/article/details/54563515
选择采用的是锦标赛法可参考下面的锦标赛博客的讲解:
http://www.cnblogs.com/legend1130/archive/2016/03/29/5333087.html
遗传算法实验
要求:
车辆路径问题(VRP)是运筹学领域一个经典的组合优化问题,可以描述为:一定数量的客户,各自有不同数量的货物需求,配送中心向客户提供货物,由一个车队负责分送货物,组织适当的行车路线,目标是使得客户的需求得到满足,达到总配送路程最短的目的。(Cvrp 带有容量限制的)要求按GA算法思想设计VRPLIB中算例eil51的求解算法,并利用计算机语言实现设计的算法。实验数据网址:http://comopt.ifi.uni-heidelberg.de/software/TSPLIB95/vrp/
本程序可以调节是否可用交叉和变异,及其参数值,以及锦标赛法选取的后代数目可在参数设置区进行调节(默认0.1倍种群大小) ,仅仅将下面的两个数据文件与GA.cpp文件放在一个工程目录下即可运行代码。
capacity.txt
position.txt
下面是实现的代码(Windows----IDE----DEVC++)
/*
Name: GA算法实现 车辆的CVRP问题
Copyright:
Author: GCJ NEW NEU Labotatry
Date: 12/11/17 20:49
Description: 本文件和附加的参数capacity.txt和data.txt文档放在一个工程下即可,
然后修改readTxt()函数内部的路径即可运行成功 */ #include <iostream>
#include"time.h"
#include<fstream>
#include<math.h>
#include<stdlib.h> /*--------------------------------参数配置区---------------------------------------------------*/ #define CLIENT_NUM 50 //客户数量 为50 1个配送中心
#define CAPACITY 160 //车的容量为160
#define Population_size 50 //种群大小
#define iterations 50 //迭代次数 #define ISGA_crossover 1 //是否可交叉 1:交叉 0:不交叉
#define PC 0.7 //配置交叉率 #define ISmutate 1 //是否可变异 1: 变异 0:不变异
#define PM 0.1 //变异率 #define IsChampionShip 1 //锦标赛参数是否可调节(默认值为0.1倍的种群大小) 1:可调 0:不可调 /*--------------------------------宏配置区---------------------------------------------------*/ #define Min(x,y) ( ( (x) < (y) ) ?(x):(y) )
#define Max(x,y) ( ( (x) > (y) ) ?(x):(y) )
#define f(x) (x -1) //用f宏 作为index 因为在找商店的序号跟二维数组之间相差1 所以用f表示两者之间的映射 /*---锦标赛参数设置区---*/
#ifdef ChampionShip
double championShip = 0.2; //自己可随意设置成0-1之间的小数 但是最好不要超过0.5
#endif typedef int ElementType;
using namespace std;
ElementType **Distance; //存储商店之间的距离
ElementType * Capacity; //存储车容量
typedef struct _rand{
int flag;
ElementType num;
}Rand; class Chromosome
{
public:
Chromosome();
Chromosome(int len ); //length表示染色体的长度
virtual~Chromosome(); //析构函数
Chromosome(const Chromosome&a); //自定义拷贝构造函数
const Chromosome &operator =(const Chromosome & o ) ; void initialize() ; //初始化染色体 调用newRandom函数 产生1- length 的随机数
int newRandom(int low,int high ); //随机产生 0- num 个不重复的数字
void evaluate();
#if ISmutate
void mutate(); //采用逆转变异算子
#endif
//查看染色体内容的调试函数
void toprint(){
int i;
// cout<<"染色体内容"<<endl;
for( i = ; i<CLIENT_NUM ; i++){
cout<<codespace[i]<<" ";
}
}
void printpath(); //打印最后的车辆安排路径
ElementType getFitness(){return this->fitness;} //返回染色体适应值
int getLength(){ return length; } //获取染色体长度
int getCar(){return carNum; } //获取车的数量
int *codespace = NULL; //编码空间 代表2-51商店的标号 private:
int length; //染色体的长度
ElementType fitness; //方便之后的数据的扩展
int carNum; //车数量
};
typedef struct _Cross{
ElementType one;
ElementType two;
int flag1 ; //标记找到的one
int flag2 ; //标记找到的two
_Cross():flag1(),flag2(){}
}Cross; //部分交叉映射需要用到的结构 记录映射关系
class GA
{
public:
GA(){};
GA(int popnum,int max); //popnum 种群大小,max表示迭代次数
virtual~GA();
GA(const GA&o); //自定义拷贝构造函数 把指针的情况考虑进去了
const GA &operator =(const GA & o ); //自定义赋值函数 //成员函数
void initializePop(); //初始化种群
void GArun(); //运行GA算法
void Insert_Sort (Chromosome * list,int len); //种群中按照适应值从小到大排序
Chromosome &slectChromosome(Chromosome* pop); //选择好的染色体用锦标赛法 返回引用的原因是 不需要拷贝构造 #if ISGA_crossover
void crossoverChromosome( Chromosome &one,Chromosome &two);//染色体交叉 然后修补不符合编码规则的个体
#endif int search(Cross *a,int num,ElementType b,ElementType *c,int opt); //在Cross型的数组中寻找b 然后寻找后的结果存到c中 void printBestChromosome(){ cout<<endl<<"Best适应值: "<<best_chr.getFitness();
cout<<endl<<"Best车数量: "<<best_chr.getCar()<<endl<<"Best染色体序列:\n "<<endl; best_chr.toprint();
}
void printDebug(int i){ cout<<"正常输出"<<i<<endl;} //测试代码bug
void BubbleSort(int *list,int len) //用来最后检测当前的染色体是不是顺序排列的
{
int i,j,temp;
for(i=;i<len;i++)
for(j=;j<len-i-;j++)
{
if(list[j]>list[j+])
{
temp=list[j];
list[j]=list[j+];
list[j+]=temp;
}
}
}
//成员变量
int length; //表示染色体的长度 此时没有用到
int popsize; //种群的规模
int max_gen; //种群的迭代次数
int elite_num;
Chromosome *old_pop; //老的种群
Chromosome *new_pop; //新产生的种群
Chromosome *pool_pop; //种群池
Chromosome good_chr; //好的染色体即 个体
Chromosome best_chr; //最好的个体
}; /*-------------------------------------------Chromosome(染色体)成员函数实现区-------------------------------------------------*/ Chromosome::Chromosome():fitness(),length(CLIENT_NUM),carNum(){
int i;
codespace = new int[length ];
for(i = ;i< length ;i++ ){
codespace[i] = ; //默认染色体的值为0
}
}
/*
Description: 申请空间并初始化染色体 适应值默认为0 可选择染色体的长度
*/
Chromosome::Chromosome( int len):length(len),fitness(),carNum() {
int i;
codespace = new int[len ];
for(i = ;i< length ;i++ ){
codespace[i] = ; //默认染色体的值为0
}
}
/*
Description: 拷贝构造函数
*/
Chromosome::Chromosome(const Chromosome&a){ //自定义拷贝构造函数 把指针的情况考虑进去了
int i;
codespace = new int[a.length];
for(i = ;i< a.length ;i++ ){
codespace[i] = a.codespace[i];
}
fitness = a.fitness;
length = a.length;
carNum = a.carNum;
}
/*
Description: 赋值构造函数
*/
const Chromosome& Chromosome::operator =(const Chromosome & o ) {
int i;
for(i = ; i< length;i++) //仅仅赋值
this->codespace[i] =o.codespace[i];
this->fitness = o.fitness;
this->carNum = o.carNum;
return *this;
}
/*
Description: 析构函数
*/
Chromosome::~Chromosome(){
delete codespace; //释放申请的资源
}
/*
Description: 随机产生 0- num 个不重复的数字 上下限 返回产生的序列 high 表示产生的最大数字 low最低数字
*/
int Chromosome::newRandom(int low,int high ) {
Rand newrand[high - low +]; //定义排列数字的那么大的数组
int record[high - low +]; //记录产生的随机数
int i,j=, rand1;
int index;
int count = high- low +; //low - high 间的数字 目前剩下没有选取到的 数量
for( i =;i< high-low +;i++){
newrand[i].num = low + i; //先产生low - high顺序排列的数组 之后 用随机数的方式产生一个任意随机的排列
newrand[i].flag = ; //表示这个元素没有被随机的取到
}
//随机产生0- num不重复的核心
while( (count !=) && ( rand1 = rand()%(count)+ )){
count --; //剩余数量--
for( i = ,index = ; i < high-low + ; i++){ //如果被标记了 就继续寻找
if( newrand[i].flag )
continue;
index ++; //表示 遍历有效元素的个数
if( index == rand1 ){
record[j] = newrand[i].num;
codespace[j] = newrand[i].num;
newrand[i].flag = ; //表示标记过了
j++;
}
}/*end for*/
}/* end while*/
}
/*
Description: 染色体初始化
*/
void Chromosome:: initialize() {
newRandom(,length+); //产生1-length 随机数 1-50
}
/*
Description: 对染色体的适值更新
*/
void Chromosome::evaluate() {
int i ,carCount = ,sumCapacity= ;
fitness = Distance[ ][f(codespace[]) ];
for( i =; i<length; i++ ){
if(sumCapacity <= CAPACITY ){
sumCapacity += Capacity[ f(codespace[i]) ]; //2- 51 对应的 商店存储量 }
else{
i--;
sumCapacity = ;
carCount++;
fitness += Distance[ ][ f(codespace[i]) ];
}
}
//出来后 最后的车肯定不满 所以需要加上最后一个商店到0的距离
fitness += Distance[][f(codespace[i-])];
for( i = ; i< length - ; i ++)
fitness += Distance[ Min( f(codespace[i]),f(codespace[i+]) )][ Max( f(codespace[i]),f(codespace[i+]) ) ];
carNum = carCount;
}
/*
Description: 染色体变异 50个 即产生0-49即可选择哪里变异
*/
#if ISmutate
void Chromosome::mutate(){
int rand1,rand2;
double rand3;
int i,j;
ElementType * temp;
int temp2,temp3;
rand3 = (rand()%)/9.0; //产生0-1小数
if(rand3 < PM){
//产生变异
rand1 = (rand()%length);
rand2 = (rand()%length);
while( (rand1 == rand2)) {
rand2 = (rand()%length);
}
temp3 = Max(rand1,rand2);
temp2 = Min(rand1,rand2);
temp = new int[temp3 - temp2+]; for( j=temp3-temp2 ,i =temp2; i<=temp3; j--,i++ ){
temp[j] = codespace[i];
}
//交换
for(j = ,i =temp2;i<=temp3;j++,i++){
codespace[i] = temp[j];
}
delete temp;
}/*end if*/
}
#endif
/*
Description: 打印路径
*/
void Chromosome::printpath(){
int i ,carCount = ,sumCapacity= ,j=,k=,l=;
fitness = Distance[ ][f(codespace[]) ];
printf("\n第1辆车:\n");
for( j=,i =; i<length; i++ ){ if(sumCapacity <= CAPACITY ){
sumCapacity += Capacity[ f(codespace[i]) ]; //2- 51 对应的 商店存储量
cout<<codespace[i]<<" ";
}
else{
i--;
printf("\n\n第%d辆车:\n ",carCount+);
j++;
sumCapacity = ;
carCount++;
fitness += Distance[ ][ f(codespace[i]) ];
}
}
//出来后 最后的车肯定不满 所以需要加上最后一个商店到0的距离
fitness += Distance[][f(codespace[i-])];
for( i = ; i< length - ; i ++)
fitness += Distance[ Min( f(codespace[i]),f(codespace[i+]) )][ Max( f(codespace[i]),f(codespace[i+]) ) ];
carNum = carCount; } /*-------------------------------------------GA成员函数实现区----------------------------------------------------------*/ GA::GA(int popnum,int max):popsize(popnum),length(CLIENT_NUM),max_gen(max),elite_num(),old_pop(NULL),new_pop(NULL),pool_pop(NULL){
old_pop = new Chromosome[popsize];
new_pop = new Chromosome[popsize];
pool_pop = new Chromosome[popsize+elite_num] ;
}
/*
Description: 拷贝构造函数
*/
GA::GA(const GA& o){
int i;
if(old_pop == NULL){
old_pop = new Chromosome[popsize];
new_pop = new Chromosome[popsize];
pool_pop = new Chromosome[popsize+elite_num] ;
}
for( i = ;i<o.popsize;i++){
old_pop[i] = o.old_pop[i];
new_pop[i] = o.new_pop[i];
pool_pop[i] = o.pool_pop[i];
}
for( ;i<o.popsize+o.elite_num;i++){
pool_pop[i] = o.pool_pop[i];
}
length = o.length;
popsize = o.popsize;
max_gen = o.max_gen;
elite_num = o.elite_num;
Chromosome good_chr = o.good_chr;
Chromosome best_chr = o.best_chr;
}
/*
Description: 赋值函数
*/
const GA& GA::operator =(const GA&o){
int i;
for( i = ;i<o.popsize;i++){
old_pop[i] = o.old_pop[i];
new_pop[i] = o.new_pop[i];
pool_pop[i] = o.pool_pop[i];
}
for( ;i<o.popsize+o.elite_num;i++){
pool_pop[i] = o.pool_pop[i];
}
length = o.length;
popsize = o.popsize;
max_gen = o.max_gen;
elite_num = o.elite_num;
Chromosome good_chr = o.good_chr;
Chromosome best_chr = o.best_chr;
}
/*
Description: 析构函数
*/
GA::~GA(){
delete[]old_pop;
delete[]new_pop;
delete[]pool_pop;
}
/*
Description: 初始化种群
*/
void GA::initializePop(){
int i;
for( i = ;i<popsize;i++){
old_pop[i].initialize();
old_pop[i].evaluate();
}
}
/*
Description: 种群中按照适应值从小到大排序
*/
void GA::Insert_Sort (Chromosome * pop,int num)
{
//进行N-1轮插入过程
int i,k;
for(i=; i<num; i++){
//首先找到元素a[i]需要插入的位置 int j=;
while( (pop[j].getFitness()< pop[i].getFitness() ) && (j <i ) )
j++; //将元素插入到正确的位置
if(i != j){ //如果i==j,说明a[i]刚好在正确的位置
Chromosome temp = pop[i];
for(k = i; k > j; k--){
pop[k] = pop[k-];
}
pop[j] = temp;
}
}
}
/*
Description: 选择好的染色体用锦标赛法 默认以popsize *0.1 (可配置成可调) 百分之10的个体里 进行选择最好的个体
*/ //在选择之前 因为已经排好序了 号码越小,代表个体越好
Chromosome& GA::slectChromosome(Chromosome* pop){
int i;
int rand1,rand2;
ElementType small = popsize-;
#if ISChampionShip
int num = popsize *championShip;
#else
int num = popsize * 0.1;
#endif for( i = ; i< num;i++){
rand1 = rand()%popsize;
if( rand1 < small)
small = rand1;
}
return pop[small];
}
/*
Description: 在Cross型的数组中寻找b 然后寻找后的结果存到c中 为交叉做准备
*/
int GA::search( Cross *a,int num,ElementType b,ElementType *c,int opt){
int i;
if(opt == )//从1里面找
{
for( i = ; i < num;i++){ if( (a[i].flag1 == )&&( b == a[i].one ) ){
*c = i;//记录找到的位置
a[i].flag1 =;
return ;
}
} }
else if(opt == ){
//从2里面找
for( i = ; i < num;i++){
if( (a[i].flag2 == )&&( b == a[i].two ) ){
//找到了就做标记
a[i].flag2 = ;
*c = i;//记录找到的位置
return ;
}
}
}
return ;
}
/*
Description: 交叉 然后修补不符合编码规则的个体 采用部分映射交叉 如果两代的个体相同 那么就必须产生另一种算法
*/
#if ISGA_crossover
void GA::crossoverChromosome( Chromosome &one,Chromosome &two){
int rand1,rand2; //rand1 小 rand2 大
int i,j,k;
Cross * temp;
Cross * table ; //保存映射的表
int count= ; //记录存在几个映射关系
int temp2,temp3;
int emp2,emp3;
int lastcount = ; int opt = ; //开始选择2
//产生随机数选择交叉点
rand1 = (rand()%length);
rand2 = (rand()%length);
while( rand1 == rand2) {
rand2 = (rand()%length);
} temp2 = Min(rand1,rand2);
temp3 = Max(rand1,rand2);
emp2 = temp2;//新加变量
emp3 = temp3;
temp = new Cross[temp3 - temp2+]; //记录部分映射的位置元素 进行映射关系表的建立
table = new Cross[temp3 - temp2 +]; //记录映射表 因为映射关系表 <= 此时申请的空间 for( j= ,i =temp2; i<=temp3; j++,i++ ){
temp[j].one = one.codespace[i];
temp[j].two = two.codespace[i];
}
//交换
for(j = ,i =temp2;i<=temp3;j++,i++){
one.codespace[i] = temp[j].two;
two.codespace[i] = temp[j].one;
}
//建立映射表 在从剩下的表格中从新用这种方法再次寻找
again:for( k=,j=,i = ; i< temp3-temp2+;i++ ){
int position;
if( (temp[i].flag2 != ) ) //如果被找到了 那么就不找了 从新i++去找
{
continue;
}
//找到了才做标记
//先从one 里找
k=;
opt = ;
if( search(temp,temp3-temp2+,temp[i].one,&position,opt) ){
//找到了
//做标记 temp[i].flag1 = ;
temp[i].flag2 = ; temp[position].flag1 = ;
table[j].two = temp[i].two;
k = position; while( ( temp[i].two != temp[position].one ) && ( search(temp,temp3-temp2+,temp[k].one,&position,opt) ) ){
//不相等 并且能够找到下一个 就继续找
k = position;
temp[position].flag1 = ;
} //退出循环有两种情况 1.相等 2 没有找到
if( temp[i].two == temp[position].one )
continue;
else{
//没有找到
table[j].one = temp[k].one;
temp[k].flag1 =;
count++;//映射数量
j++;
}
}else{//没有找到 opt = ;//返过来从1中找
if( search(temp,temp3-temp2+,temp[i].two,&position,opt) ){
//找到了
//做标记
temp[i].flag1 = ;
temp[i].flag2 = ; temp[position].flag2 = ;
table[j].one = temp[i].one;
k = position;
while( ( temp[i].one != temp[position].two ) && ( search(temp,temp3-temp2+,temp[k].two,&position,opt) ) ){
//不相等 并且能够找到下一个 就继续找
k = position;
temp[position].flag2 = ;
}
//退出循环有两种情况 1.相等 2 没有找到
if( temp[i].one == temp[position].two )
continue;
else{
//没有找到
table[j].two = temp[k].two;
temp[k].flag2 =;
count++;//映射数量
j++;
}
}else{ //最后都没有找到 说明两个映射 没有循环
temp[i].flag1 = ;
temp[i].flag2 = ;
table[j].two = temp[i].two;
table[j].one = temp[i].one;
j++;
count++;
}/*iner if else */
} /*outer if else */
}
if(count >= )//需要第二次检查
{//说明count>0
if(lastcount != count)//两次的值不相等就再次检查 直到两次的值都是固定不变的
{
int flag = ;
lastcount =count;//记录上次的值
for( i = ; i< count;i++ ) //再次检查映射表
{
temp[i].one = table[i].one;
temp[i].two = table[i].two;
temp[i].flag1 = ; //标记清零
temp[i].flag2 = ;
temp2 = ;
temp3 = count-;
flag = ; }
if(flag ==){
count = ;
flag = ;
goto again;
}
}
}
for( i =;i< emp2;i++){
//修复前一段
for(j=;j<count;j++){
if( one.codespace[i] == table[j].two ){
one.codespace[i] = table[j].one;
break;
}
}
}
for( i =emp3+;i< length;i++){
for(j=;j<count;j++){
if( one.codespace[i] == table[j].two ){
one.codespace[i] = table[j].one;
break;
}
}
}
for( i =;i< emp2;i++){
//修复前一段
for(j=;j<count;j++){
if( two.codespace[i] == table[j].one ){
two.codespace[i] = table[j].two;
break;
}
}
}
for( i =emp3+;i< length;i++){
for(j=;j<count;j++){
if( two.codespace[i] == table[j].one ){
two.codespace[i] = table[j].two;
break;
}
}
} delete temp;
delete table;
}
#endif /*
Description: 运行算法
*/
void GA::GArun(){
int i,gen;
double rand1; //初始化种群
initializePop(); //排序 按适应值从小到大排序
Insert_Sort(old_pop,popsize); //保存好的个体
good_chr = old_pop[];
best_chr = old_pop[]; //迭代 选择 交叉 变异
for( gen =; gen < max_gen;gen++){ //选择好的个体进入new_pop
for(i = ; i< popsize;i++){
//选择好的染色体的 函数返回值用值返回 这些个体会有相同的部分所以在交叉的过程中要考虑
new_pop[i] = slectChromosome(old_pop);
} #if ISGA_crossover
//交叉
for( i = ;i<popsize/;i++){
rand1 = (rand()%)/9.0;
if( rand1 < PC ) {
crossoverChromosome( new_pop[*i],new_pop[*i+] );//前后两个交叉
}
}
#endif #if ISmutate
//对每个new_pop 中的个体 进行变异
for( i = ;i< popsize ;i++){
new_pop[i].mutate();
}
#endif //种群中的单个染色体进行更新适应值
for( i = ; i< popsize ; i++){
new_pop[i].evaluate();
} //将new_pop中的染色体 放到pool中,然后在从old_pop中选择elite_num个杰出的染色体 放到pool_pop中
for(i = ; i <popsize;i++){
pool_pop[i] = new_pop[i];
}
for(i = ; i< elite_num; i++){
pool_pop[popsize +i] = old_pop[i];
} //在按照适应值从小到大进行排序pool_pop
Insert_Sort(pool_pop,popsize+elite_num); //old_pop = pool_pop中的前popsize个染色体
for(i = ;i< popsize;i++){
old_pop[i] = pool_pop[i];
} //从old_pop中选择好的个体进行保存good_chr best_chr
good_chr = old_pop[];
if( good_chr.getFitness() < best_chr.getFitness())
best_chr = good_chr;//调用赋值函数
}/*for end*/
//输出最好的染色体 best_chr.printpath();
cout<<endl;
printBestChromosome();
}
void readTxt()
{
int i,j;
double temp1;
int ShopNum = CLIENT_NUM +;
ElementType *x,*y,temp; //读入位置坐标 之后计算商店之间的距离
Distance = new int*[ShopNum];
Capacity = new int [ShopNum]; x = new int[ShopNum];
y = new int[ShopNum];
fstream file("position.txt", ios::in);
fstream file2("capacity.txt",ios::in);
if(!file.is_open() || !file2.is_open() ){
cout << "Can not open the data file " << "遗传算法\\position.txt or capacity.txt" << endl;
exit();
}
else
cout <<"The file has been opened without problems "<<"遗传算法\\position.txt and capacity.txt"<<endl;
for( i = ;i< ShopNum ; i++){
Distance[i] = new int[ShopNum ]; //构造51 x 51 的矩阵
} //将数据存入到 一维数组中 之
for( i =; i< ShopNum; i++){
file>>temp>>x[i]>>y[i]; //存储距离
file2>>temp>>Capacity[i]; //存储商店需求量
}
for(i = ;i< ShopNum;i++ ){ //51x51大小的矩阵 存储距离
Distance[i][i] = ; //对角线上的元素为0
for(j = i+;j< ShopNum;j++ ){
temp1 = sqrt(pow(x[i] - x[j ], ) + pow(y[i] - y[j ], ));
temp1 = (int )(temp1 + 0.5);
Distance[i][j] = temp1;
Distance[j][i] = temp1;
}
}
file.close();
file2.close();
delete x; //释放空间
delete y;
} /*
Description: 释放Distance 和Capacity指向的空间
*/
void freeMem(){
int ShopNum = CLIENT_NUM+;
int i;
for( i = ;i< ShopNum ; i++){
delete Distance [i];
}
delete Distance ;
delete Capacity;
}
/*
Description: main
*/
int main(int argc, char** argv) {
int i,b=;
int count=;
clock_t start, finish;
double duration; srand(unsigned(time())); //置一个随机数种子 为了以后产生随机数
readTxt(); //从文件中读取 算法需要的数据
GA d(Population_size,iterations); //用两个宏 配置种群和迭代次数
start = clock(); //GA算法
d.GArun(); //释放存储商店和车容量所占资源
freeMem();
finish = clock();
duration = (double)(finish - start) / CLOCKS_PER_SEC;
printf("\n\n GA算法运行时间:%.4f秒 \n",duration);
printf("\n 迭代次数:%d \n",iterations);
printf("\n 种群大小:%d \n",Population_size); return ; }
下面是试验图片:
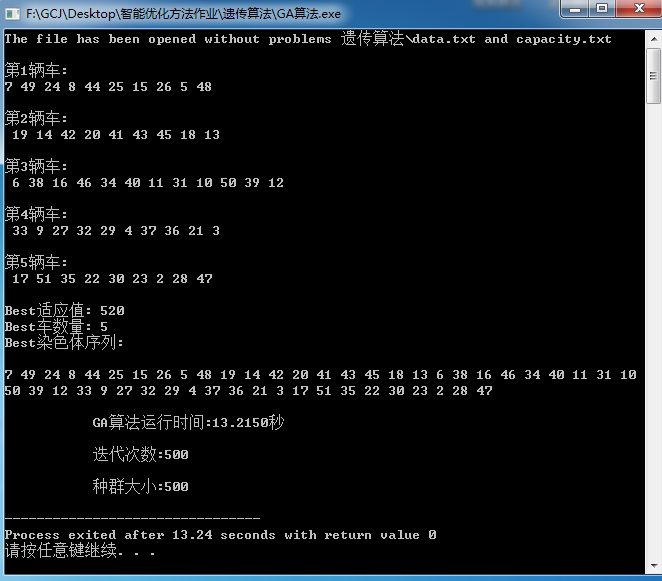
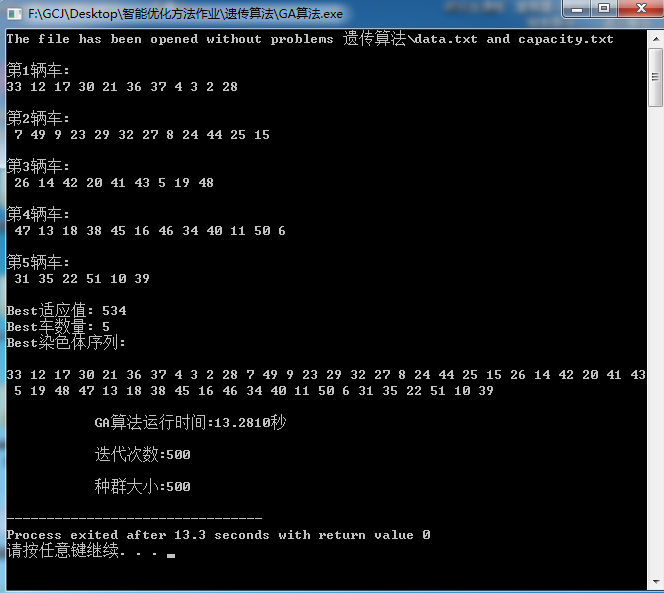
如果读者想要数据,可以与我联系,或者直接到下面网址下载完整工程
http://download.csdn.net/my
欢迎大家关注我的微信公众号「佛系师兄」,里面会更新一些相关的技术文章。
比如
「反复研究好几遍,我才发现关于 CMake 变量还可以这样理解!」
更多好的文章会优先在里面不定期分享!打开微信客户端,扫描下方二维码即可关注!

(原创)遗传算法C++实现的更多相关文章
- 简单遗传算法求解n皇后问题
版权声明:本文为博主原创文章,转载请注明出处. 先解释下什么是8皇后问题:在8×8格的国际象棋上摆放八个皇后,使其不能互相攻击,即任意两个皇后都不能处于同一行.同一列或同一斜线上,问有多少种摆法.在不 ...
- MIP启发式算法:遗传算法 (Genetic algorithm)
*本文主要记录和分享学习到的知识,算不上原创 *参考文献见链接 本文主要讲述启发式算法中的遗传算法.遗传算法也是以local search为核心框架,但在表现形式上和hill climbing, ta ...
- Python动态展示遗传算法求解TSP旅行商问题(转载)
版权声明:本文为博主原创文章,遵循 CC 4.0 by-sa 版权协议,转载请附上原文出处链接和本声明.本文链接:https://blog.csdn.net/jiang425776024/articl ...
- 【原创分享·支付宝支付】HBuilder打包APP调用支付宝客户端支付
前言 最近有点空余时间,所以,就研究了一下APP支付.前面很早就搞完APP的微信支付了,但是由于时间上和应用上的情况,支付宝一直没空去研究.然后等我空了的时候,发现支付宝居然升级了支付逻辑,虽然目前还 ...
- 【原创分享·微信支付】C# MVC 微信支付教程系列之现金红包
微信支付教程系列之现金红包 最近最弄这个微信支付的功能,然后扫码.公众号支付,这些都做了,闲着无聊,就看了看微信支付的其他功能,发现还有一个叫“现金红包”的玩意,想 ...
- 【原创分享·微信支付】 C# MVC 微信支付教程系列之扫码支付
微信支付教程系列之扫码支付 今天,我们来一起探讨一下这个微信扫码支付.何为扫码支付呢?这里面,扫的码就是二维码了,就是我们经常扫一扫的那种二维码图片,例如,我们自己添 ...
- 【原创分享·微信支付】 C# MVC 微信支付教程系列之公众号支付
微信支付教程系列之公众号支付 今天,我们接着讲微信支付的系列教程,前面,我们讲了这个微信红包和扫码支付.现在,我们讲讲这个公众号支付.公众号支付的应用环境常见的用户通过公众号,然后再通 ...
- 【原创分享·微信支付】C# MVC 微信支付之微信模板消息推送
微信支付之微信模板消息推送 今天我要跟大家分享的是“模板消息”的推送,这玩意呢,你说用途嘛,那还是真真的牛逼呐.原因在哪?就是因为它是依赖微信生存的呀,所以他能不 ...
- [原创]java使用JDBC向MySQL数据库批次插入10W条数据测试效率
使用JDBC连接MySQL数据库进行数据插入的时候,特别是大批量数据连续插入(100000),如何提高效率呢?在JDBC编程接口中Statement 有两个方法特别值得注意:通过使用addBatch( ...
随机推荐
- windows下怎么解决Python双版本问题
相信大家会在windows下会遇到Python双版本问题 当我们装了Python2和Python3时我们好只能在命令栏调出最高版本的那个低版本的难道消失了吗?今天我们就解决这个问题! 1.下载 我们在 ...
- Java高级工程师进阶路线
第一部分:宏观方面 一. JAVA.要想成为JAVA(高级)工程师肯定要学习JAVA.一般的程序员或许只需知道一些JAVA的语法结构就可以应付了.但要成为JAVA(高级) 工程师,您要对JAVA做比较 ...
- python urllib、urlparse、urllib2、cookielib
1.urllib模块 1.urllib.urlopen(url[,data[,proxies]]) 打开一个url的方法,返回一个文件对象,然后可以进行类似文件对象的操作.本例试着打开google i ...
- python中package注意事项
个人工作中的SSD.Cardreader.Camera.Audio模块文档组织形式如下: RclLib __init__.py RclLegacy.py modules AgilentOp.py Uv ...
- Python学习手册 :Python 学习笔记第一天
获取当前目录路径: import os os.getcwd() 在输入python程序时,尽量让不是嵌套结构的语句处于最左侧,要不然系统或许会出现"SyntaxError"错误 获 ...
- 轻松几句搞定【Javascript中的this指向】问题
this关键字在JavaScript中扮演了至关重要的角色,每次它的出现都伴随着它的指向问题,这也是很多初学者容易出错的地方. 不过,这篇文章将会带你一次性搞定this指向的问题,望能给大家提供帮助! ...
- 批量下载验证码 shell
#!/bin/sh seq 0 699 | xargs -i wget http://www.5184.com/gk/common/checkcode.php -O img/{}.png
- Ubuntu Mac OS主题分享
Ubuntu Mac OS主题分享 一直想搞一个Mac OS主题试试,结果很悲催,在网上搜索的Macbuntu主题在安装主题(macbuntu-os-themes-Its-v7)和 图标(macbun ...
- win10 uwp 从StorageFile获取文件大小
本文主要:获取文件大小 private async Task<ulong> FileSize(Windows.Storage.StorageFile file) { var size = ...
- 基于Python实现matplotlib中动态更新图片(交互式绘图)
最近在研究动态障碍物避障算法,在Python语言进行算法仿真时需要实时显示障碍物和运动物的当前位置和轨迹,利用Anaconda的Python打包集合,在Spyder中使用Python3.5语言和mat ...
