高斯混合模型GMM与EM算法的Python实现
GMM与EM算法的Python实现
高斯混合模型(GMM)是一种常用的聚类模型,通常我们利用最大期望算法(EM)对高斯混合模型中的参数进行估计。
1. 高斯混合模型(Gaussian Mixture models, GMM)
高斯混合模型(Gaussian Mixture Model,GMM)是一种软聚类模型。 GMM也可以看作是K-means的推广,因为GMM不仅是考虑到了数据分布的均值,也考虑到了协方差。和K-means一样,我们需要提前确定簇的个数。
GMM的基本假设为数据是由几个不同的高斯分布的随机变量组合而成。如下图,我们就是用三个二维高斯分布生成的数据集。
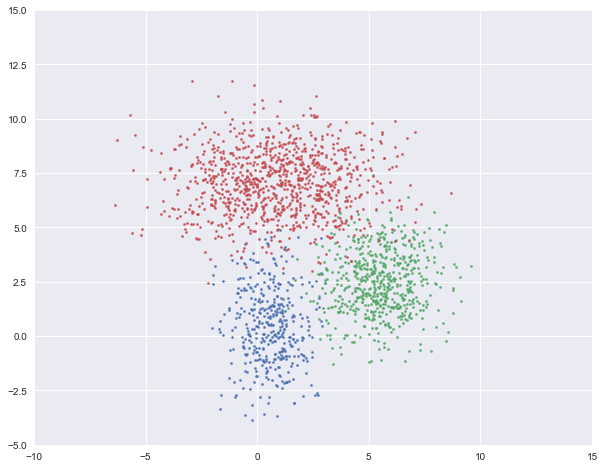
在高斯混合模型中,我们需要估计每一个高斯分布的均值与方差。从最大似然估计的角度来说,给定某个有n个样本的数据集X,假如已知GMM中一共有k 簇,我们就是要找到k组均值μ1,⋯,μk,k组方差σ1,⋯,σk 来最大化以下似然函数L
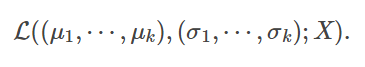
这里直接计算似然函数比较困难,于是我们引入隐变量(latent variable),这里的隐变量就是每个样本属于每一簇的概率。假设W是一个n×k的矩阵,其中 Wi,j 是第 i 个样本属于第 j 簇的概率。
在已知W的情况下,我们就很容易计算似然函数LW,
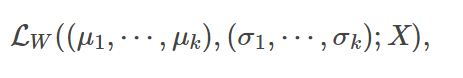
将其写成
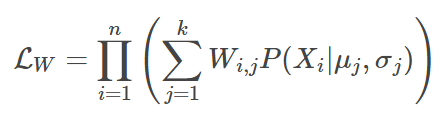
其中P(Xi | μj, σj) 是样本Xi在第j个高斯分布中的概率密度函数。
以一维高斯分布为例,
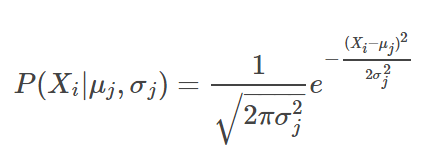
2. 最大期望算法(Expectation–Maximization, EM)
有了隐变量还不够,我们还需要一个算法来找到最佳的W,从而得到GMM的模型参数。EM算法就是这样一个算法。
简单说来,EM算法分两个步骤。
- 第一个步骤是E(期望),用来更新隐变量WW;
- 第二个步骤是M(最大化),用来更新GMM中各高斯分布的参量μj, σj。
然后重复进行以上两个步骤,直到达到迭代终止条件。
3. 具体步骤以及Python实现
完整代码在第4节。
首先,我们先引用一些我们需要用到的库和函数。
import numpy as np
import matplotlib.pyplot as plt
from matplotlib.patches import Ellipse
from scipy.stats import multivariate_normal
plt.style.use('seaborn')
接下来,我们生成2000条二维模拟数据,其中400个样本来自N(μ1,var1),600个来自N(μ2,var2),1000个样本来自N(μ3,var3)
# 第一簇的数据
num1, mu1, var1 = 400, [0.5, 0.5], [1, 3]
X1 = np.random.multivariate_normal(mu1, np.diag(var1), num1)
# 第二簇的数据
num2, mu2, var2 = 600, [5.5, 2.5], [2, 2]
X2 = np.random.multivariate_normal(mu2, np.diag(var2), num2)
# 第三簇的数据
num3, mu3, var3 = 1000, [1, 7], [6, 2]
X3 = np.random.multivariate_normal(mu3, np.diag(var3), num3)
# 合并在一起
X = np.vstack((X1, X2, X3))
数据如下图所示:
plt.figure(figsize=(10, 8))
plt.axis([-10, 15, -5, 15])
plt.scatter(X1[:, 0], X1[:, 1], s=5)
plt.scatter(X2[:, 0], X2[:, 1], s=5)
plt.scatter(X3[:, 0], X3[:, 1], s=5)
plt.show()

3.1 变量初始化
首先要对GMM模型参数以及隐变量进行初始化。通常可以用一些固定的值或者随机值。
n_clusters 是GMM模型中聚类的个数,和K-Means一样我们需要提前确定。这里通过观察可以看出是3。(拓展阅读:如何确定GMM中聚类的个数?)
n_points 是样本点的个数。
Mu 是每个高斯分布的均值。
Var 是每个高斯分布的方差,为了过程简便,我们这里假设协方差矩阵都是对角阵。
W 是上面提到的隐变量,也就是每个样本属于每一簇的概率,在初始时,我们可以认为每个样本属于某一簇的概率都是1/3。
Pi 是每一簇的比重,可以根据W求得,在初始时,Pi = [1/3, 1/3, 1/3]
n_clusters = 3
n_points = len(X)
Mu = [[0, -1], [6, 0], [0, 9]]
Var = [[1, 1], [1, 1], [1, 1]]
Pi = [1 / n_clusters] * 3
W = np.ones((n_points, n_clusters)) / n_clusters
Pi = W.sum(axis=0) / W.sum()
3.2 E步骤
E步骤中,我们的主要目的是更新W。第i个变量属于第m簇的概率为:

根据W,我们就可以更新每一簇的占比πm,
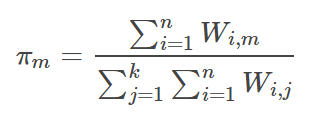
def update_W(X, Mu, Var, Pi):
n_points, n_clusters = len(X), len(Pi)
pdfs = np.zeros(((n_points, n_clusters)))
for i in range(n_clusters):
pdfs[:, i] = Pi[i] * multivariate_normal.pdf(X, Mu[i], np.diag(Var[i]))
W = pdfs / pdfs.sum(axis=1).reshape(-1, 1)
return W def update_Pi(W):
Pi = W.sum(axis=0) / W.sum()
return Pi
以下是计算对数似然函数的logLH以及用来可视化数据的plot_clusters。
def logLH(X, Pi, Mu, Var):
n_points, n_clusters = len(X), len(Pi)
pdfs = np.zeros(((n_points, n_clusters)))
for i in range(n_clusters):
pdfs[:, i] = Pi[i] * multivariate_normal.pdf(X, Mu[i], np.diag(Var[i]))
return np.mean(np.log(pdfs.sum(axis=1))) def plot_clusters(X, Mu, Var, Mu_true=None, Var_true=None):
colors = ['b', 'g', 'r']
n_clusters = len(Mu)
plt.figure(figsize=(10, 8))
plt.axis([-10, 15, -5, 15])
plt.scatter(X[:, 0], X[:, 1], s=5)
ax = plt.gca()
for i in range(n_clusters):
plot_args = {'fc': 'None', 'lw': 2, 'edgecolor': colors[i], 'ls': ':'}
ellipse = Ellipse(Mu[i], 3 * Var[i][0], 3 * Var[i][1], **plot_args)
ax.add_patch(ellipse)
if (Mu_true is not None) & (Var_true is not None):
for i in range(n_clusters):
plot_args = {'fc': 'None', 'lw': 2, 'edgecolor': colors[i], 'alpha': 0.5}
ellipse = Ellipse(Mu_true[i], 3 * Var_true[i][0], 3 * Var_true[i][1], **plot_args)
ax.add_patch(ellipse)
plt.show()
3.2 M步骤
M步骤中,我们需要根据上面一步得到的W来更新均值Mu和方差Var。 Mu和Var是以W的权重的样本X的均值和方差。
因为这里的数据是二维的,第m簇的第k个分量的均值,
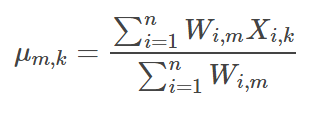
第m簇的第k个分量的方差,
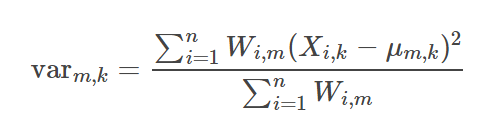
以上迭代公式写成如下函数update_Mu和update_Var。
def update_Mu(X, W):
n_clusters = W.shape[1]
Mu = np.zeros((n_clusters, 2))
for i in range(n_clusters):
Mu[i] = np.average(X, axis=0, weights=W[:, i])
return Mu def update_Var(X, Mu, W):
n_clusters = W.shape[1]
Var = np.zeros((n_clusters, 2))
for i in range(n_clusters):
Var[i] = np.average((X - Mu[i]) ** 2, axis=0, weights=W[:, i])
return Var
3.3 迭代求解
下面我们进行迭代求解。
图中实现是真实的高斯分布,虚线是我们估计出的高斯分布。可以看出,经过5次迭代之后,两者几乎完全重合。
loglh = []
for i in range(5):
plot_clusters(X, Mu, Var, [mu1, mu2, mu3], [var1, var2, var3])
loglh.append(logLH(X, Pi, Mu, Var))
W = update_W(X, Mu, Var, Pi)
Pi = update_Pi(W)
Mu = update_Mu(X, W)
print('log-likehood:%.3f'%loglh[-1])
Var = update_Var(X, Mu, W)
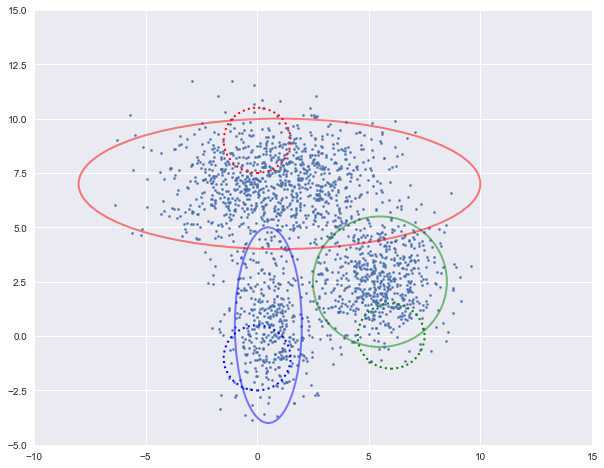
log-likehood:-8.054
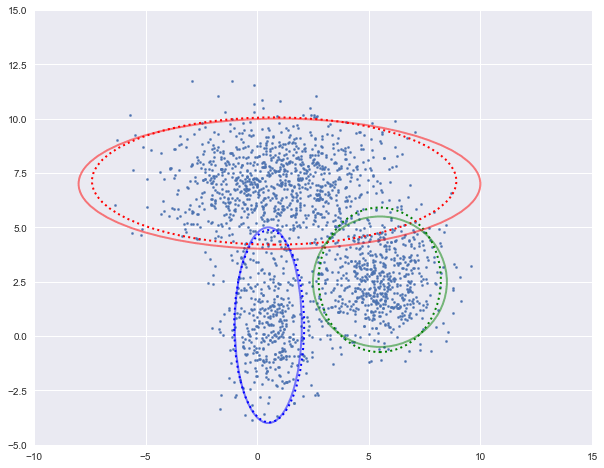
log-likehood:-4.731
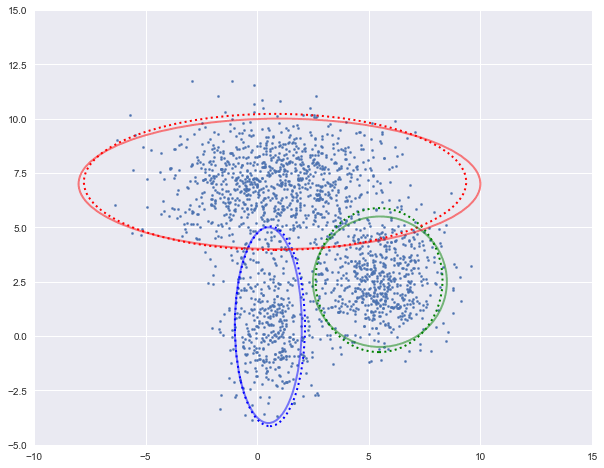
log-likehood:-4.729
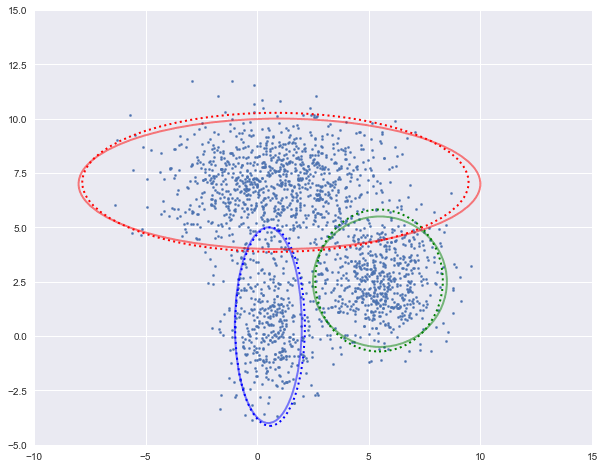
log-likehood:-4.728
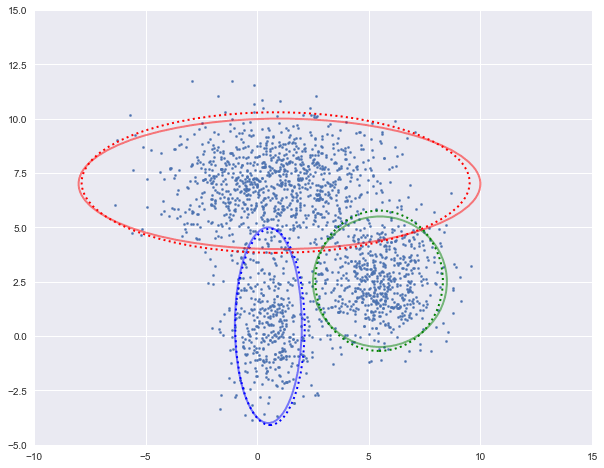
log-likehood:-4.728
4. 完整代码
import numpy as np
import matplotlib.pyplot as plt
from matplotlib.patches import Ellipse
from scipy.stats import multivariate_normal
plt.style.use('seaborn') # 生成数据
def generate_X(true_Mu, true_Var):
# 第一簇的数据
num1, mu1, var1 = 400, true_Mu[0], true_Var[0]
X1 = np.random.multivariate_normal(mu1, np.diag(var1), num1)
# 第二簇的数据
num2, mu2, var2 = 600, true_Mu[1], true_Var[1]
X2 = np.random.multivariate_normal(mu2, np.diag(var2), num2)
# 第三簇的数据
num3, mu3, var3 = 1000, true_Mu[2], true_Var[2]
X3 = np.random.multivariate_normal(mu3, np.diag(var3), num3)
# 合并在一起
X = np.vstack((X1, X2, X3))
# 显示数据
plt.figure(figsize=(10, 8))
plt.axis([-10, 15, -5, 15])
plt.scatter(X1[:, 0], X1[:, 1], s=5)
plt.scatter(X2[:, 0], X2[:, 1], s=5)
plt.scatter(X3[:, 0], X3[:, 1], s=5)
plt.show()
return X # 更新W
def update_W(X, Mu, Var, Pi):
n_points, n_clusters = len(X), len(Pi)
pdfs = np.zeros(((n_points, n_clusters)))
for i in range(n_clusters):
pdfs[:, i] = Pi[i] * multivariate_normal.pdf(X, Mu[i], np.diag(Var[i]))
W = pdfs / pdfs.sum(axis=1).reshape(-1, 1)
return W # 更新pi
def update_Pi(W):
Pi = W.sum(axis=0) / W.sum()
return Pi # 计算log似然函数
def logLH(X, Pi, Mu, Var):
n_points, n_clusters = len(X), len(Pi)
pdfs = np.zeros(((n_points, n_clusters)))
for i in range(n_clusters):
pdfs[:, i] = Pi[i] * multivariate_normal.pdf(X, Mu[i], np.diag(Var[i]))
return np.mean(np.log(pdfs.sum(axis=1))) # 画出聚类图像
def plot_clusters(X, Mu, Var, Mu_true=None, Var_true=None):
colors = ['b','g','r']
n_clusters = len(Mu)
plt.figure(figsize=(10,8))
plt.axis([-10,15,-5,15])
plt.scatter(X[:,0], X[:,1], s=5)
ax = plt.gca()for i in range(n_clusters):
plot_args ={'fc':'None','lw':2,'edgecolor': colors[i],'ls':':'}
ellipse =Ellipse(Mu[i],3*Var[i][0],3*Var[i][1],**plot_args)
ax.add_patch(ellipse)if(Mu_trueisnotNone)&(Var_trueisnotNone):for i in range(n_clusters):
plot_args ={'fc':'None','lw':2,'edgecolor': colors[i],'alpha':0.5}
ellipse =Ellipse(Mu_true[i],3*Var_true[i][0],3*Var_true[i][1],**plot_args)
ax.add_patch(ellipse)
plt.show()# 更新Mudef update_Mu(X, W):
n_clusters = W.shape[1]Mu= np.zeros((n_clusters,2))for i in range(n_clusters):Mu[i]= np.average(X, axis=0, weights=W[:, i])returnMu# 更新Vardef update_Var(X,Mu, W):
n_clusters = W.shape[1]Var= np.zeros((n_clusters,2))for i in range(n_clusters):Var[i]= np.average((X -Mu[i])**2, axis=0, weights=W[:, i])returnVarif __name__ =='__main__':# 生成数据
true_Mu =[[0.5,0.5],[5.5,2.5],[1,7]]
true_Var =[[1,3],[2,2],[6,2]]
X = generate_X(true_Mu, true_Var)# 初始化
n_clusters =3
n_points = len(X)Mu=[[0,-1],[6,0],[0,9]]Var=[[1,1],[1,1],[1,1]]Pi=[1/ n_clusters]*3
W = np.ones((n_points, n_clusters))/ n_clusters
Pi= W.sum(axis=0)/ W.sum()# 迭代
loglh =[]for i in range(5):
plot_clusters(X,Mu,Var, true_Mu, true_Var)
loglh.append(logLH(X,Pi,Mu,Var))
W = update_W(X,Mu,Var,Pi)Pi= update_Pi(W)Mu= update_Mu(X, W)print('log-likehood:%.3f'%loglh[-1])Var= update_Var(X,Mu, W)
本教程基于Python 3.6。
原创者:u_u | 修改校对:SofaSofa TeamM | 转自: http://sofasofa.io/tutorials/gmm_em/
高斯混合模型GMM与EM算法的Python实现的更多相关文章
- 高斯混合模型参数估计的EM算法
# coding:utf-8 import numpy as np def qq(y,alpha,mu,sigma,K,gama):#计算Q函数 gsum=[] n=len(y) for k in r ...
- 5. EM算法-高斯混合模型GMM+Lasso
1. EM算法-数学基础 2. EM算法-原理详解 3. EM算法-高斯混合模型GMM 4. EM算法-GMM代码实现 5. EM算法-高斯混合模型+Lasso 1. 前言 前面几篇博文对EM算法和G ...
- 4. EM算法-高斯混合模型GMM详细代码实现
1. EM算法-数学基础 2. EM算法-原理详解 3. EM算法-高斯混合模型GMM 4. EM算法-高斯混合模型GMM详细代码实现 5. EM算法-高斯混合模型GMM+Lasso 1. 前言 EM ...
- 3. EM算法-高斯混合模型GMM
1. EM算法-数学基础 2. EM算法-原理详解 3. EM算法-高斯混合模型GMM 4. EM算法-高斯混合模型GMM详细代码实现 5. EM算法-高斯混合模型GMM+Lasso 1. 前言 GM ...
- 6. EM算法-高斯混合模型GMM+Lasso详细代码实现
1. 前言 我们之前有介绍过4. EM算法-高斯混合模型GMM详细代码实现,在那片博文里面把GMM说涉及到的过程,可能会遇到的问题,基本讲了.今天我们升级下,主要一起解析下EM算法中GMM(搞事混合模 ...
- EM算法和高斯混合模型GMM介绍
EM算法 EM算法主要用于求概率密度函数参数的最大似然估计,将问题$\arg \max _{\theta_{1}} \sum_{i=1}^{n} \ln p\left(x_{i} | \theta_{ ...
- GMM及EM算法
GMM及EM算法 标签(空格分隔): 机器学习 前言: EM(Exception Maximizition) -- 期望最大化算法,用于含有隐变量的概率模型参数的极大似然估计: GMM(Gaussia ...
- 贝叶斯来理解高斯混合模型GMM
最近学习基础算法<统计学习方法>,看到利用EM算法估计高斯混合模型(GMM)的时候,发现利用贝叶斯的来理解高斯混合模型的应用其实非常合适. 首先,假设对于贝叶斯比较熟悉,对高斯分布也熟悉. ...
- Spark2.0机器学习系列之10: 聚类(高斯混合模型 GMM)
在Spark2.0版本中(不是基于RDD API的MLlib),共有四种聚类方法: (1)K-means (2)Latent Dirichlet allocation (LDA) ...
随机推荐
- [LeetCode] 525. Contiguous Array 相连的数组
Given a binary array, find the maximum length of a contiguous subarray with equal number of 0 and 1. ...
- mod35云掩膜产品用法
在网上一直没找到一个明确说怎么用MOD35产品的,都是说去看用户手册,第一次看了过一段时间我又忘记怎么搞了,赶紧记下来. 而且现在才发现第一次自己搞的都弄错了. 简单的判断是否是云,只要读取mod35 ...
- SQLAIchemy 学习(一)Session 相关
0. 前言 最近是使用 SQLAlchemy 框架作为一个 ORM 框架,现对其做简单整理 1. 创建 Session 说到数据库,就离不开 Session.Session 的主要目的是建立与数据库的 ...
- nodejs搭建web服务教程
nodejs搭建web服务教程 先安装nodejs 然后 命令node js文件 即可开启服务了 输出如下 Magic happens on port 9011 你查看端口可以看到tcp 0 0 :: ...
- First Step in luogu.
2019-11-21 21:58:32 在洛谷正式迈出第一步!!
- IScroll Unable to preventDefault inside passive event listener due to target being treated as passive
最近两天企业微信下IScroll突然无法滚动了,特别慢,之前好好的,发现主要是有红色的Unable to preventDefault inside passive event listener du ...
- 极简 Spring Boot 整合 Thymeleaf 页面模板
虽然现在慢慢在流行前后端分离开发,但是据松哥所了解到的,还是有一些公司在做前后端不分的开发,而在前后端不分的开发中,我们就会需要后端页面模板(实际上,即使前后端分离,也会在一些场景下需要使用页面模板, ...
- 在Mac 上搭建Linux虚拟机--MacOS & VMware10 & CentOS 7
在大型项目开发中, 需要使用Linux下的C语言对工程进行开发, 在个人PC或者工作站上搭建Linux系统十分容易且方便. 本篇文章将介绍操作系统和虚拟机的搭建: 1 操作系统2 虚拟机概念3 Lin ...
- golang学习笔记----并发
并发模型 并发目前来看比较主流的就三种: 多线程:每个线程一次处理一个请求,线程越多可并发处理的请求数就越多,但是在高并发下,多线程开销会比较大. 协程:无需抢占式的调度,开销小,可以有效的提高线程的 ...
- React学习之路之创建项目
React 开发环境准备 IDE工具 visual studio code 开发环境 开发环境需要安装nodejs和npm,nodejs工具包含了npm. nodejs下载官网:https://nod ...
