吴裕雄--天生自然 R语言开发学习:高级编程















运行的条件是一元逻辑向量(TRUE或FALSE)并且不能有缺失(NA)。else部分是可选的。如果
仅有一个语句,花括号也是可以省略的。
下面的代码片段是一个例子:
if(interactive()){
plot(x, y)
} else {
png("myplot.png")
plot(x, y)
dev.off()
}
如果代码交互运行,interactive()函数返回TRUE,同时输出一个曲线图。否则,曲线图被存
在磁盘里。你可以使用第21章中的if()函数。
. ifelse()
ifelse()是函数if()的量化版本。矢量化允许一个函数来处理没有明确循环的对象。
ifelse()的格式是:
ifelse(test, yes, no)
其中test是已强制为逻辑模式的对象,yes返回test元素为真时的值,no返回test元素为假时
的值。
比如你有一个p值向量,是从包含六个统计检验的统计分析中提取出来的,并且你想要标记
p<.05水平下的显著性检验。可以使用下面的代码:
> pvalues <- c(., ., ., ., ., .)
> results <- ifelse(pvalues <., "Significant", "Not Significant")
> results
[] "Not Significant" "Significant" "Significant"
[] "Not Significant" "Significant" "Not Significant"
ifelse()函数通过pvalues向量循环并返回一个包括"Significant"或"Not Significant"
的字符串。返回的结果依赖于pvalues返回的值是否大于0.。
同样的结果可以使用显式循环完成:
pvalues <- c(., ., ., ., ., .)
results <- vector(mode="character", length=length(pvalues))
for(i in :length(pvalues)){
if (pvalues[i] < .) results[i] <- "Significant"
else results[i] <- "Not Significant"
}
可以看出,向量化的版本更快且更有效。
有一些其他的控制结构,包括while()、repeat()和switch(),但是这里介绍的是最常用
的。有了数据结构和控制结构,我们就可以讨论创建函数了。
20.1. 创建函数
在R中处处是函数。算数运算符+、-、/和*实际上也是函数。例如, + 2等价于 "+"(, )。
本节将主要描述函数语法。语句环境将在20-2节描述。
. 函数语法
函数的语法格式是:
functionname <- function(parameters){
statements
return(value)
}
如果函数中有多个参数,那么参数之间用逗号隔开。
参数可以通过关键字和/或位置来传递。另外,参数可以有默认值。请看下面的函数:
f <- function(x, y, z=){
result <- x + (*y) + (*z)
return(result)
}
> f(,,)
[]
> f(,)
[]
> f(x=, y=)
[]
> f(z=, y=, )
[]
在第一个例子中,参数是通过位置(x=,y=,z=)传递的。在第二个例子中,参数也是通过
位置传递的,并且z默认为1。在第三个例子中,参数是通过关键字传递的,z也默认为1。在最后
一个例子中,y和z是通过关键字传递的,并且x被假定为未明确指定的(这里x=)第一个参数。
参数是可选的,但即使没有值被传递也必须使用圆括号。return()函数返回函数产生的对
象。它也是可选的;如果缺失,函数中最后一条语句的结果也会被返回。
你可以使用args()函数来观测参数的名字和默认值:
> args(f)
function (x, y, z = )
NULL
args()被设计用于交互式观测。如果你需要以编程方式获取参数名称和默认值,可以使用
formals()函数。它返回含有必要信息的列表。
参数是按值传递的,而不是按地址传递。请看下面这个函数语句:
result <- lm(height ~ weight, data=women)
women数据集不是直接得到的。需要形成一个副本然后传递给函数。如果women数据集很大的话,
内存(RAM)可能被迅速用完。这可能成为处理大数据问题时的难题能需要使用特殊的技术(见






#--------------------------------------------------------------------#
# R in Action (2nd ed): Chapter #
# Advanced R programming #
# requires packages ggplot2, reshape2, foreach, doParallel #
# install.packages(c("ggplot2", "reshap2e", "foreach", "doParallel"))#
#--------------------------------------------------------------------# # Atomic vectors
passed <- c(TRUE, TRUE, FALSE, TRUE)
ages <- c(, , , , )
cmplxNums <- c(+2i, +1i, +3i, +2i)
names <- c("Bob", "Ted", "Carol", "Alice") # Matrices
x <- c(,,,,,,,)
class(x)
print(x)
attr(x, "dim") <- c(,)
print(x)
class(x)
attributes(x)
attr(x, "dimnames") <- list(c("A1", "A2"),
c("B1", "B2", "B3", "B4"))
print(x)
attr(x, "dim") <- NULL
class(x)
print(x) # Generic vectors (lists)
head(iris)
unclass(iris)
attributes(iris) set.seed()
fit <- kmeans(iris[:], )
names(fit)
unclass(fit)
sapply(fit, class) # Indexing atomic vectors
x <- c(, , )
x[]
x[c(,)]
x <- c(A=, B=, C=)
x[c(,)]
x[c("B", "C")] # Indexing lists
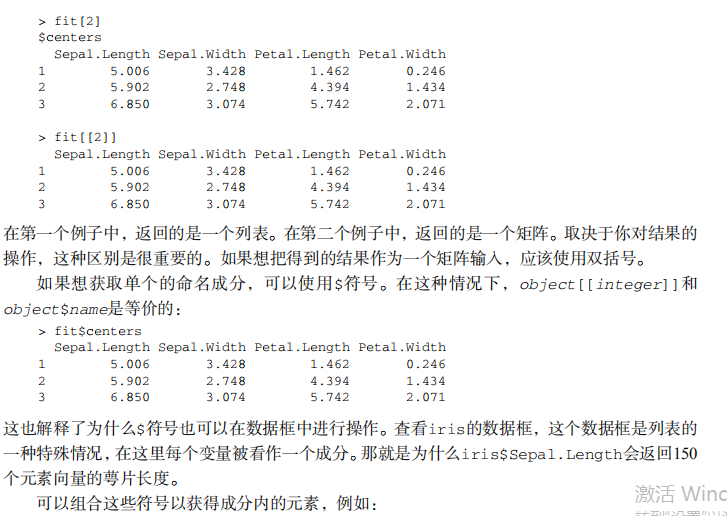
fit[c(,)]
fit[]
fit[[]]
fit$centers
fit[[]][,]
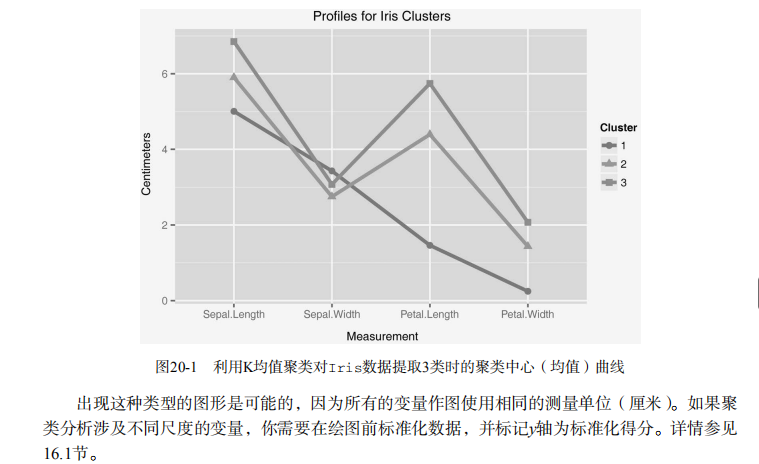
fit$centers$Petal.Width # should give an error # Listing 20.1 - Plotting the centroides from a k-mean cluster analysis
fit <- kmeans(iris[:], )
means <- fit$centers
library(reshape2)
dfm <- melt(means)
names(dfm) <- c("Cluster", "Measurement", "Centimeters")
dfm$Cluster <- factor(dfm$Cluster)
head(dfm)
library(ggplot2)
ggplot(data=dfm,
aes(x=Measurement, y=Centimeters, group=Cluster)) +
geom_point(size=, aes(shape=Cluster, color=Cluster)) +
geom_line(size=, aes(color=Cluster)) +
ggtitle("Profiles for Iris Clusters") # for loops
for(i in :) print(:i)
for(i in :)print(:i) # ifelse
pvalues <- c(., ., ., ., ., .)
results <- ifelse(pvalues <., "Significant", "Not Significant")
results pvalues <- c(., ., ., ., ., .)
results <- vector(mode="character", length=length(pvalues))
for(i in :length(pvalues)){
if (pvalues[i] < .) results[i] <- "Significant"
else results[i] <- "Not Significant"
}
results # Creating functions
f <- function(x, y, z=){
result <- x + (*y) + (*z)
return(result)
} f(,,)
f(,)
f(x=, y=)
f(z=, y=, )
args(f) # object scope
x <-
y <-
z <-
f <- function(w){
z <-
x <- w*y*z
return(x)
}
f(x)
x
y
z # Working with environments
x <-
myenv <- new.env()
assign("x", "Homer", env=myenv)
ls()
ls(myenv)
x
get("x", env=myenv) myenv <- new.env()
myenv$x <- "Homer"
myenv$x parent.env(myenv) # function closures
trim <- function(p){
trimit <- function(x){
n <- length(x)
lo <- floor(n*p) +
hi <- n + - lo
x <- sort.int(x, partial = unique(c(lo, hi)))[lo:hi]
}
trimit
}
x <- :
trim10pct <- trim(.)
y <- trim10pct(x)
y
trim20pct <- trim(.)
y <- trim20pct(x)
y ls(environment(trim10pct))
get("p", env=environment(trim10pct)) makeFunction <- function(k){
f <- function(x){
print(x + k)
}
} g <- makeFunction()
g ()
k <-
g () ls(environment(g))
environment(g)$k # Generic functions
summary(women)
fit <- lm(weight ~ height, data=women)
summary(fit) class(women)
class(fit)
methods(summary) # Listing 20.2 - An example of a generic function
mymethod <- function(x, ...) UseMethod("mymethod")
mymethod.a <- function(x) print("Using A")
mymethod.b <- function(x) print("Using B")
mymethod.default <- function(x) print("Using Default") x <- :
y <- :
z <- :
class(x) <- "a"
class(y) <- "b" mymethod(x)
mymethod(y)
mymethod(z) class(z) <- c("a", "b")
mymethod(z)
class(z) <- c("c", "a", "b")
mymethod(z) # Vectorization and efficient code
set.seed()
mymatrix <- matrix(rnorm(), ncol=)
accum <- function(x){
sums <- numeric(ncol(x))
for (i in :ncol(x)){
for(j in :nrow(x)){
sums[i] <- sums[i] + x[j,i]
}
}
}
system.time(accum(mymatrix)) # using loops
system.time(colSums(mymatrix)) # using vectorization # Correctly size objects
set.seed()
k <-
x <- rnorm(k) y <-
system.time(for (i in :length(x)) y[i] <- x[i]^) y <- numeric(k)
system.time(for (i in :k) y[i] <- x[i]^) y <- numeric(k)
system.time(y <- x^) # Listing 20.3 - Parallelization with foreach and doParallel
library(foreach)
library(doParallel)
registerDoParallel(cores=) eig <- function(n, p){
x <- matrix(rnorm(), ncol=)
r <- cor(x)
eigen(r)$values
}
n <-
p <-
k <- system.time(
x <- foreach(i=:k, .combine=rbind) %do% eig(n, p)
) system.time(
x <- foreach(i=:k, .combine=rbind) %dopar% eig(n, p)
) # Finding common errors
mtcars$Transmission <- factor(mtcars$a,
levels=c(,),
labels=c("Automatic", "Manual"))
aov(mpg ~ Transmission, data=mtcars) # generates error
head(mtcars[c("mpg", "Transmission")])
table(mtcars$Transmission) # here is the source of the error # Listing 20.4 - A sample debugging session
args(mad)
debug(mad)
mad(:)
# enters debugging mode
# Q to quit - see text
undebug(mad) # Listing 20.5 - Sample debugging session with recover()
f <- function(x, y){
z <- x + y
g(z)
}
g <- function(x){
z <- round(x)
h(z)
} h <- function(x){
set.seed()
z <- rnorm(x)
print(z)
}
options(error=recover) f(,)
f(, -) # enters debugging mode at this point
吴裕雄--天生自然 R语言开发学习:高级编程的更多相关文章
- 吴裕雄--天生自然 R语言开发学习:R语言的安装与配置
下载R语言和开发工具RStudio安装包 先安装R
- 吴裕雄--天生自然 R语言开发学习:数据集和数据结构
数据集的概念 数据集通常是由数据构成的一个矩形数组,行表示观测,列表示变量.表2-1提供了一个假想的病例数据集. 不同的行业对于数据集的行和列叫法不同.统计学家称它们为观测(observation)和 ...
- 吴裕雄--天生自然 R语言开发学习:导入数据
2.3.6 导入 SPSS 数据 IBM SPSS数据集可以通过foreign包中的函数read.spss()导入到R中,也可以使用Hmisc 包中的spss.get()函数.函数spss.get() ...
- 吴裕雄--天生自然 R语言开发学习:使用键盘、带分隔符的文本文件输入数据
R可从键盘.文本文件.Microsoft Excel和Access.流行的统计软件.特殊格 式的文件.多种关系型数据库管理系统.专业数据库.网站和在线服务中导入数据. 使用键盘了.有两种常见的方式:用 ...
- 吴裕雄--天生自然 R语言开发学习:R语言的简单介绍和使用
假设我们正在研究生理发育问 题,并收集了10名婴儿在出生后一年内的月龄和体重数据(见表1-).我们感兴趣的是体重的分 布及体重和月龄的关系. 可以使用函数c()以向量的形式输入月龄和体重数据,此函 数 ...
- 吴裕雄--天生自然 R语言开发学习:基础知识
1.基础数据结构 1.1 向量 # 创建向量a a <- c(1,2,3) print(a) 1.2 矩阵 #创建矩阵 mymat <- matrix(c(1:10), nrow=2, n ...
- 吴裕雄--天生自然 R语言开发学习:图形初阶(续二)
# ----------------------------------------------------# # R in Action (2nd ed): Chapter 3 # # Gettin ...
- 吴裕雄--天生自然 R语言开发学习:图形初阶(续一)
# ----------------------------------------------------# # R in Action (2nd ed): Chapter 3 # # Gettin ...
- 吴裕雄--天生自然 R语言开发学习:图形初阶
# ----------------------------------------------------# # R in Action (2nd ed): Chapter 3 # # Gettin ...
- 吴裕雄--天生自然 R语言开发学习:基本图形(续二)
#---------------------------------------------------------------# # R in Action (2nd ed): Chapter 6 ...
随机推荐
- 修改自己的centos输入法
当自己的centos连上网时,就可以修改自己的输入法了 http://jingyan.baidu.com/album/da1091fb3e7f8a027849d681.html?picindex=2
- 吴裕雄--天生自然ShellX学习笔记:Shell 输入/输出重定向
大多数 UNIX 系统命令从你的终端接受输入并将所产生的输出发送回到您的终端.一个命令通常从一个叫标准输入的地方读取输入,默认情况下,这恰好是你的终端.同样,一个命令通常将其输出写入到标准输出,默 ...
- 吴裕雄--天生自然ShellX学习笔记:Shell 数组
数组中可以存放多个值.Bash Shell 只支持一维数组(不支持多维数组),初始化时不需要定义数组大小(与 PHP 类似). 与大部分编程语言类似,数组元素的下标由0开始. Shell 数组用括号来 ...
- java的io字符流关闭和刷新.flush();
因为内置缓冲区的原因,如果不关闭输出流,无法写出字符到文件中. 但是关闭的流对象,是无法继续写出数据 的.如果我们既想写出数据,又想继续使用流,就需要 flush 方法了. flush :刷新缓冲区, ...
- 优秀的github java项目
转载:https://www.zhihu.com/question/24834285/answer/251369977 biezhi/blade:先推荐下自己的哈哈,一款轻量级.高性能.简洁优雅的MV ...
- Canvas 橡皮擦效果
引子 解决了第一个问题图像灰度处理之后,接着就是做擦除的效果. Origin My GitHub 思路 一开始想到 Canvas 的画布可以相互覆盖的特性,彩色原图作为背景,灰度图渲染到 Canvas ...
- CodeForces 992B Nastya Studies Informatics + Hankson的趣味题(gcd、lcm)
http://codeforces.com/problemset/problem/992/B 题意: 给你区间[l,r]和x,y 问你区间中有多少个数对 (a,b) 使得 gcd(a,b)=x lc ...
- coursera课程视频
#!/usr/bin/env python # coding=utf-8 import urllib import urllib2 import cookielib def setcookie(ena ...
- Django2.0——django-filter: TypeError at *** __init__() got an unexpected keyword argument 'name'
在使用 Django2.0 版本的 Django Rest Framwork 时,Django DeBug 报错 django-filter: TypeError at *** __init__() ...
- Spring Boot从入门到放弃-Spring Boot 整合测试
站长资讯摘要:使用Spring Boot 整合测试,对Controller 中某个方法进行测试或者对Service,Mapper等进行测试,不需要运行项目即可查看运行结果是否和期望值相同,Spring ...
