R语言使用 multicore 包进行并行计算
R语言是单线程的,如果数据量比较大的情况下最好用并行计算来处理数据,这样会获得运行速度倍数的提升。这里介绍一个基于Unix系统的并行程序包:multicore.
我们用三种不同的方式来进行一个简单的数据处理:
我们从 1000 genome project 数据库下载了VCF文件,现在需要手动提取出每个allele的 allele frequency(AF)值(vcftools 可以很好的解决这个问题,但是假设我的vcf文件没有genotype, 或者我要实现一些个性化功能,那么可能要手动解决)。我们仅仅提取VCF文件的前1,000,000行作测试。看一下文件前几行:
##fileformat=VCFv4.
##INFO=<ID=LDAF,Number=,Type=Float,Description="MLE Allele Frequency Accounting for LD">
##INFO=<ID=AVGPOST,Number=,Type=Float,Description="Average posterior probability from MaCH/Thunder">
##INFO=<ID=RSQ,Number=,Type=Float,Description="Genotype imputation quality from MaCH/Thunder">
##INFO=<ID=ERATE,Number=,Type=Float,Description="Per-marker Mutation rate from MaCH/Thunder">
##INFO=<ID=THETA,Number=,Type=Float,Description="Per-marker Transition rate from MaCH/Thunder">
##INFO=<ID=CIEND,Number=,Type=Integer,Description="Confidence interval around END for imprecise variants">
##INFO=<ID=CIPOS,Number=,Type=Integer,Description="Confidence interval around POS for imprecise variants">
##INFO=<ID=END,Number=,Type=Integer,Description="End position of the variant described in this record">
##INFO=<ID=HOMLEN,Number=.,Type=Integer,Description="Length of base pair identical micro-homology at event breakpoints">
##INFO=<ID=HOMSEQ,Number=.,Type=String,Description="Sequence of base pair identical micro-homology at event breakpoints">
##INFO=<ID=SVLEN,Number=,Type=Integer,Description="Difference in length between REF and ALT alleles">
##INFO=<ID=SVTYPE,Number=,Type=String,Description="Type of structural variant">
##INFO=<ID=AC,Number=.,Type=Integer,Description="Alternate Allele Count">
##INFO=<ID=AN,Number=,Type=Integer,Description="Total Allele Count">
##ALT=<ID=DEL,Description="Deletion">
##FORMAT=<ID=GT,Number=,Type=String,Description="Genotype">
##FORMAT=<ID=DS,Number=,Type=Float,Description="Genotype dosage from MaCH/Thunder">
##FORMAT=<ID=GL,Number=.,Type=Float,Description="Genotype Likelihoods">
##INFO=<ID=AA,Number=,Type=String,Description="Ancestral Allele, ftp://ftp.1000genomes.ebi.ac.uk/vol1/ftp/pilot_data/technical/reference/ancestral_alignments/README">
##INFO=<ID=AF,Number=,Type=Float,Description="Global Allele Frequency based on AC/AN">
##INFO=<ID=AMR_AF,Number=,Type=Float,Description="Allele Frequency for samples from AMR based on AC/AN">
##INFO=<ID=ASN_AF,Number=,Type=Float,Description="Allele Frequency for samples from ASN based on AC/AN">
##INFO=<ID=AFR_AF,Number=,Type=Float,Description="Allele Frequency for samples from AFR based on AC/AN">
##INFO=<ID=EUR_AF,Number=,Type=Float,Description="Allele Frequency for samples from EUR based on AC/AN">
##INFO=<ID=VT,Number=,Type=String,Description="indicates what type of variant the line represents">
##INFO=<ID=SNPSOURCE,Number=.,Type=String,Description="indicates if a snp was called when analysing the low coverage or exome alignment data">
##reference=GRCh37
#CHROM POS ID REF ALT QUAL FILTER INFO
rs58108140 G A PASS AVGPOST=0.7707;RSQ=0.4319;LDAF=0.2327;ERATE=0.0161;AN=;VT=SNP;AA=.;THETA=0.0046;AC=;SNPSOURCE=LOWCOV;AF=0.14;ASN_AF=0.13;AMR_AF=0.17;AFR_AF=0.04;EUR_AF=0.21
rs189107123 C G PASS AN=;THETA=0.0077;VT=SNP;AA=.;AC=;ERATE=0.0048;SNPSOURCE=LOWCOV;AVGPOST=0.9330;LDAF=0.0479;RSQ=0.3475;AF=0.02;ASN_AF=0.01;AMR_AF=0.03;AFR_AF=0.01;EUR_AF=0.02
rs180734498 C T PASS THETA=0.0048;AN=;AC=;VT=SNP;AA=.;RSQ=0.6281;LDAF=0.1573;SNPSOURCE=LOWCOV;AVGPOST=0.8895;ERATE=0.0058;AF=0.11;ASN_AF=0.02;AMR_AF=0.08;AFR_AF=0.21;EUR_AF=0.14
rs144762171 G C PASS AVGPOST=0.9698;AN=;VT=SNP;AA=.;RSQ=0.6482;AC=;SNPSOURCE=LOWCOV;ERATE=0.0012;LDAF=0.0359;THETA=0.0204;AF=0.03;ASN_AF=0.02;AMR_AF=0.03;AFR_AF=0.02;EUR_AF=0.04
rs201747181 TC T PASS AA=TC;AC=;AF=0.02;AFR_AF=0.02;AMR_AF=0.02;AN=;ASN_AF=0.01;AVGPOST=0.8711;ERATE=0.0065;EUR_AF=0.02;LDAF=0.0788;RSQ=0.2501;THETA=0.0100;VT=INDEL
rs151276478 T C PASS AN=;AC=;ERATE=0.0034;THETA=0.0139;RSQ=0.3603;LDAF=0.0525;VT=SNP;AA=.;AVGPOST=0.9221;SNPSOURCE=LOWCOV;AF=0.02;ASN_AF=0.02;AMR_AF=0.02;AFR_AF=0.01;EUR_AF=0.02
rs140337953 G T PASS AC=;AA=T;AN=;RSQ=0.5481;VT=SNP;THETA=0.0162;SNPSOURCE=LOWCOV;ERATE=0.0183;LDAF=0.6576;AVGPOST=0.7335;AF=0.73;ASN_AF=0.89;AMR_AF=0.80;AFR_AF=0.48;EUR_AF=0.73
rs199681827 C CTGT PASS AA=.;AC=;AF=0.0037;AFR_AF=0.01;AN=;ASN_AF=0.0017;AVGPOST=0.8325;ERATE=0.0072;LDAF=0.0903;RSQ=0.0960;THETA=0.0121;VT=INDEL
rs200430748 G GA PASS AA=G;AC=;AF=0.01;AFR_AF=0.06;AMR_AF=0.0028;AN=;AVGPOST=0.9041;ERATE=0.0041;LDAF=0.0628;RSQ=0.2883;THETA=0.0153;VT=INDEL
可以发现AF值在INFO这一列字符串里面,所以要取出这个值是比较容易的,只需要对字符串进行切割即可。
先通过data.table包读入数据到计算机内存,我们看到data.table包读入数据非常快,1,000,000行数据读入仅仅5秒。同时函数非常智能的通过后面的格式将前28行识别为注释,没有读入。
library(data.table)
head_vcf <- fread("head_1000000.vcf", sep = "\t", colClasses=list(character= ,))
### Read 1000000 rows and 8 (of 8) columns from 0.166 GB file in 00:00:05
head(head_vcf)
#CHROM POS ID REF ALT QUAL FILTER
1: 1 10583 rs58108140 G A 100 PASS
2: 1 10611 rs189107123 C G 100 PASS
3: 1 13302 rs180734498 C T 100 PASS
4: 1 13327 rs144762171 G C 100 PASS
5: 1 13957 rs201747181 TC T 28 PASS
6: 1 13980 rs151276478 T C 100 PASS
INFO
1: AVGPOST=0.7707;RSQ=0.4319;LDAF=0.2327;ERATE=0.0161;AN=2184;VT=SNP;AA=.;THETA=0.0046;AC=314;SNPSOURCE=LOWCOV;AF=0.14;ASN_AF=0.13;AMR_AF=0.17;AFR_AF=0.04;EUR_AF=0.21
2: AN=2184;THETA=0.0077;VT=SNP;AA=.;AC=41;ERATE=0.0048;SNPSOURCE=LOWCOV;AVGPOST=0.9330;LDAF=0.0479;RSQ=0.3475;AF=0.02;ASN_AF=0.01;AMR_AF=0.03;AFR_AF=0.01;EUR_AF=0.02
3: THETA=0.0048;AN=2184;AC=249;VT=SNP;AA=.;RSQ=0.6281;LDAF=0.1573;SNPSOURCE=LOWCOV;AVGPOST=0.8895;ERATE=0.0058;AF=0.11;ASN_AF=0.02;AMR_AF=0.08;AFR_AF=0.21;EUR_AF=0.14
4: AVGPOST=0.9698;AN=2184;VT=SNP;AA=.;RSQ=0.6482;AC=59;SNPSOURCE=LOWCOV;ERATE=0.0012;LDAF=0.0359;THETA=0.0204;AF=0.03;ASN_AF=0.02;AMR_AF=0.03;AFR_AF=0.02;EUR_AF=0.04
5: AA=TC;AC=35;AF=0.02;AFR_AF=0.02;AMR_AF=0.02;AN=2184;ASN_AF=0.01;AVGPOST=0.8711;ERATE=0.0065;EUR_AF=0.02;LDAF=0.0788;RSQ=0.2501;THETA=0.0100;VT=INDEL
6: AN=2184;AC=45;ERATE=0.0034;THETA=0.0139;RSQ=0.3603;LDAF=0.0525;VT=SNP;AA=.;AVGPOST=0.9221;SNPSOURCE=LOWCOV;AF=0.02;ASN_AF=0.02;AMR_AF=0.02;AFR_AF=0.01;EUR_AF=0.02
第一种方法是使用内建函数,如果使用单核处理数据,那么这是最推荐的方式,内建函数都是用底层语言写好封装并优化的,运算速度非常快。
#编写一个函数,函数的参数info_str是读入文件的第八列(INFO),这是一个字符串向量
get_af_fun1 <- function(info_str)
{
split1 <- strsplit(info_str, ";AF=")
str1 <- vector(length = length(split1))
for(i in :length(str1)) str1[i] <- split1[[i]][]
split2 <- strsplit(str1, ";")
str2 <- vector(length = length(split2))
for(i in :length(str2)) str2[i] <- split2[[i]][]
str2
}
system.time(result1 <- get_af_fun1(as.character(head_vcf$INFO)))
用户 系统 流逝
38.944 0.012 38.950 #因为是根据位置索引操作,所以随机抽取三行发现结果一致
> result1[43525]
[1] "0.01"
> head_vcf[43525,]
#CHROM POS ID REF ALT QUAL FILTER
1: 1 3607355 rs3765729 G A 100 PASS
INFO
1: AA=G;AN=2184;RSQ=0.7704;VT=SNP;AC=22;LDAF=0.0126;SNPSOURCE=LOWCOV;AVGPOST=0.9929;THETA=0.0020;ERATE=0.0006;AF=0.01;ASN_AF=0.03;AMR_AF=0.02 > head_vcf[654635,]
#CHROM POS ID REF ALT QUAL FILTER
1: 1 49595556 rs192800899 G T 100 PASS
INFO
1: AA=G;AN=2184;RSQ=0.8739;AC=6;VT=SNP;THETA=0.0006;LDAF=0.0030;SNPSOURCE=LOWCOV;ERATE=0.0003;AVGPOST=0.9992;AF=0.0027;AMR_AF=0.0028;EUR_AF=0.01
> result1[654635]
[1] "0.0027"
> head_vcf[9876,]
#CHROM POS ID REF ALT QUAL FILTER
1: 1 1322735 rs146506266 G A 100 PASS
INFO
1: RSQ=0.9882;AVGPOST=1.0000;THETA=0.0004;SNPSOURCE=LOWCOV,EXOME;AA=G;AN=2184;VT=SNP;LDAF=0.0019;AC=4;ERATE=0.0003;AF=0.0018;ASN_AF=0.01
> result1[9876]
[1] "0.0018"

通过第一个例子我们看到,通过向量化运算和内建函数处理一百万行数据仅仅花费40秒不到,而且对于for循环,预先分配内存会极大的提高速度。主要原因是R语言的数据赋值是拷贝而不是引用,因此如果预先对向量分配内存就避免了每次循环都对该向量重新赋值。同时我们看到在程序是单核在计算。
第二个例子是利用R语言内建循环函数lapply(), 算法和第一个例子相似,只不过把for循环用lapply执行。
fun_str1 <- function(str_v) strsplit(str_v, ";AF=")[[]][]
fun_str2 <- function(str_v) strsplit(str_v, ";")[[]][] get_af_fun2 <- function(info_str)
{
str1 <- unlist(lapply(info_str, fun_str1))
str2 <- unlist(lapply(str1, fun_str2))
str2
}
system.time(result2 <- get_af_fun2(as.character(head_vcf$INFO)))
用户 系统 流逝
76.512 0.028 76.556 #随机抽取三行发现结果一致 > result2[543654]
[1] "0.0009"
> result2[75676]
[1] "0.0014"
> result2[8765]
[1] "0.0018"
> head_vcf[543654]
#CHROM POS ID REF ALT QUAL FILTER
1: 1 40941743 rs143883355 G A 100 PASS
INFO
1: AN=2184;THETA=0.0005;LDAF=0.0010;VT=SNP;AA=.;AVGPOST=0.9998;RSQ=0.9135;SNPSOURCE=LOWCOV;ERATE=0.0003;AC=2;AF=0.0009;AFR_AF=0.0041
> head_vcf[75676]
#CHROM POS ID REF ALT QUAL FILTER
1: 1 5569843 rs148207486 C T 100 PASS
INFO
1: ERATE=0.0004;RSQ=0.7694;AA=C;AN=2184;VT=SNP;LDAF=0.0019;AVGPOST=0.9989;THETA=0.0012;SNPSOURCE=LOWCOV;AC=3;AF=0.0014;AFR_AF=0.01
> head_vcf[8765]
#CHROM POS ID REF ALT QUAL FILTER
1: 1 1254001 rs190286788 G C 100 PASS
INFO
1: ERATE=0.0005;RSQ=0.5499;AA=G;AN=2184;AVGPOST=0.9979;VT=SNP;THETA=0.0006;SNPSOURCE=LOWCOV;AC=4;LDAF=0.0017;AF=0.0018;AMR_AF=0.0028;EUR_AF=0.0040

第二个例子我们可以看出,计算这一百万行运行时间大概70多秒,lapply是代替循环的一个非常好的函数, 这里之所以运行速度比第一种方法慢,我们从程序可以看出,是因为对这个长度为一百万的字符串向量分别调用了一百万次fun_str1和fun_str2。程序在函数调用上花费了一定时间,如果将我们自己写的fun_str1, fun_str2函数换成内建函数,速度也会提高不少。
有了第二个例子的铺垫,我们再来介绍第三个例子,也就是利用multicore 包进行并行处理。这个包是基于Unix系统的,这里简单介绍里面一个函数mclapply(), 和lapply()函数用法基本一致。具体看例子:
fun_str1 <- function(str_v) strsplit(str_v, ";AF=")[[]][]
fun_str2 <- function(str_v) strsplit(str_v, ";")[[]][] get_af_fun3 <- function(info_str)
{
str1 <- unlist(mclapply(info_str, fun_str1, mc.cores = ))
str2 <- unlist(mclapply(str1, fun_str2, mc.cores = ))
str2
}
system.time(result3 <- get_af_fun3(as.character(head_vcf$INFO)))
用户 系统 流逝
56.816 65.512 39.958 #检查三次运行的结果显示三个结果向量完全一致
> all(result1 == result2)
[1] TRUE
> all(result3 == result2)
[1] TRUE
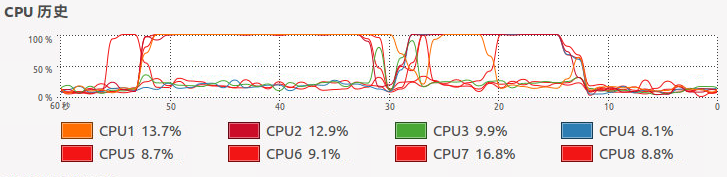
查看CPU使用状态我们可以看到通过mc.cores调用四个核进行并行运算,确实比单核的lapply运算速度提高不少,但是没有四倍的效果是并行运算系统中一些线程活动的额外开销。
再次加大运算量,将文件提高到10,000,000行, 为了避免宕机,这里请用大内存机器进行实验。
head_vcf <- fread("head_10000000.vcf", sep = "\t", colClasses=list(character= 1,6))
Read 10000000 rows and 8 (of 8) columns from 1.660 GB file in 00:02:04
方案一,使用内建函数和for循环来处理。
> system.time(result1 <- get_af_fun1(as.character(head_vcf$INFO)))
用户 系统 流逝
603.936 0.248 604.060
方案二,使用lapply()函数单核处理。
> system.time(result2 <- get_af_fun2(as.character(head_vcf$INFO)))
用户 系统 流逝
933.176 0.344 933.115
方案三,使用mclapply()函数并行处理。
> system.time(result3 <- get_af_fun3(as.character(head_vcf$INFO)))
用户 系统 流逝
984.936 251.176 351.119
这个一千万行的例子我们看出,系统内建函数的运行效率还是比lapply快, 开四个核并行的速度最快,而且这个效率还会随着计算核心数的提升而进一步获得较大的提升,有条件的用户可以选择方案三。
R语言使用 multicore 包进行并行计算的更多相关文章
- R语言︱H2o深度学习的一些R语言实践——H2o包
每每以为攀得众山小,可.每每又切实来到起点,大牛们,缓缓脚步来俺笔记葩分享一下吧,please~ --------------------------- R语言H2o包的几个应用案例 笔者寄语:受启发 ...
- R语言:recommenderlab包的总结与应用案例
R语言:recommenderlab包的总结与应用案例 1. 推荐系统:recommenderlab包整体思路 recommenderlab包提供了一个可以用评分数据和0-1数据来发展和测试推荐算 ...
- 使用R语言的RTCGA包获取TCGA数据--转载
转载生信技能树 https://mp.weixin.qq.com/s/JB_329LCWqo5dY6MLawfEA TCGA数据源 - R包RTCGA的简单介绍 - 首先安装及加载包 - 指定任意基因 ...
- R语言中文分词包jiebaR
R语言中文分词包jiebaR R的极客理想系列文章,涵盖了R的思想,使用,工具,创新等的一系列要点,以我个人的学习和体验去诠释R的强大. R语言作为统计学一门语言,一直在小众领域闪耀着光芒.直到大数据 ...
- 用R语言的quantreg包进行分位数回归
什么是分位数回归 分位数回归(Quantile Regression)是计量经济学的研究前沿方向之一,它利用解释变量的多个分位数(例如四分位.十分位.百分位等)来得到被解释变量的条件分布的相应的分位数 ...
- R语言︱文本挖掘——jiabaR包与分词向量化的simhash算法(与word2vec简单比较)
每每以为攀得众山小,可.每每又切实来到起点,大牛们,缓缓脚步来俺笔记葩分享一下吧,please~ --------------------------- <数据挖掘之道>摘录话语:虽然我比 ...
- R语言 文本挖掘 tm包 使用
#清除内存空间 rm(list=ls()) #导入tm包 library(tm) library(SnowballC) #查看tm包的文档 #vignette("tm") ##1. ...
- R语言安装xlsx包,读入excel表格
开学的时候,男神给了数据(.xlsx格式)让用R语言分析分析,作为编程小白,读了一天都没读近R,更别提如何分析了. 现在小伙伴们都喜欢读txt 和csv格式的,好多xlsx的表格读不进R,将xlsx格 ...
- R语言中常用包(二)
数据导入 以下R包主要用于数据导入和保存数据 feather:一种快速,轻量级的文件格式.在R和python上都可使用readr:实现表格数据的快速导入.中文介绍可参考这里readxl:读取Micro ...
随机推荐
- Ecplise 配置本地 https 测试
今天做项目,需要关联Office 365.为了实现Office365的用户邮件信息与项目的实时同步,需要建立webhook订阅. Office 365 API 连接 https://graph.mi ...
- Codeforces Round #426 (Div. 2)
http://codeforces.com/contest/834 A. The Useless Toy 题意: <,>,^,v这4个箭头符号,每一个都可以通过其他及其本身逆时针或者顺时针 ...
- Android:CheckBox控件
1)ChexkBox继承自CompoundButton组件: 2)isChecked()--确定是否选中:setChecked(bool checked)--设置选中或取消选中: 3)监听事件:Com ...
- hadoop fs:du统计hdfs文件(目录下文件)大小的用法
hadoop fs 更多用法,请参考官网:http://hadoop.apache.org/docs/r1.0.4/cn/hdfs_shell.html 以下是我的使用统计文件时使用的记录: [t@d ...
- POJ-2485 Highways---最小生成树中最大边
题目链接: https://vjudge.net/problem/POJ-2485 题目大意: 求最小生成树中的最大边 思路: 是稠密图,用prim更好,但是规模不大,kruskal也可以过 #inc ...
- css水平垂直居中的方法与 vertical-align 的用法
前言:这是笔者学习之后自己的理解与整理.如果有错误或者疑问的地方,请大家指正,我会持续更新! 1. 已知元素宽度 方法一:已知宽高,可以用position定位 + margin负值的方法 : 绝对定位 ...
- MySQL操作与修改表
插入数据(insert) insert语句的3个主要组成部分: 所要插入数据的表的名称: 表终需要使用的列的名称: 需要插入到列的值. 数字型主键生成机制 数字型主键生成机制,除了随机选择数字外,还可 ...
- TP-LINK | TL-WR842N设置无线转有线
首先点击右上角的"高级设置". 点击左侧的"无线设置"栏,点击"WDS无线桥接",然后一步步设置可以使路由器连接到当前的一个无线网络. 然后 ...
- 解决将/etc/passwd文件中1000改为0后只能guest进入系统的问题
一, 进入正题之前我先在这里介绍一下vi编辑器的几条主要的编辑命令,以为一会会用的到的.(悔不改当初没好好学unix啊啊啊) 最重要的一点是要知道vi编辑器分为编辑模式和命令模式,按esc键就能从编辑 ...
- [LeetCode] Find Mode in Binary Search Tree 找二分搜索数的众数
Given a binary search tree (BST) with duplicates, find all the mode(s) (the most frequently occurred ...
