吴裕雄--天生自然 R语言开发学习:分类














#-----------------------------------------------------------------------------#
# R in Action (2nd ed): Chapter 17 #
# Classification #
# requires packaged rpart, party, randomForest, kernlab, rattle #
# install.packages(c("rpart", "party", "randomForest", "e1071", "rpart.plot") #
# install.packages(rattle, dependencies = c("Depends", "Suggests")) #
#-----------------------------------------------------------------------------# par(ask=TRUE) # Listing 17.1 - Prepare the breast cancer data
loc <- "http://archive.ics.uci.edu/ml/machine-learning-databases/"
ds <- "breast-cancer-wisconsin/breast-cancer-wisconsin.data"
url <- paste(loc, ds, sep="") breast <- read.table(url, sep=",", header=FALSE, na.strings="?")
names(breast) <- c("ID", "clumpThickness", "sizeUniformity",
"shapeUniformity", "maginalAdhesion",
"singleEpithelialCellSize", "bareNuclei",
"blandChromatin", "normalNucleoli", "mitosis", "class") df <- breast[-1]
df$class <- factor(df$class, levels=c(2,4),
labels=c("benign", "malignant")) set.seed(1234)
train <- sample(nrow(df), 0.7*nrow(df))
df.train <- df[train,]
df.validate <- df[-train,]
table(df.train$class)
table(df.validate$class) # Listing 17.2 - Logistic regression with glm()
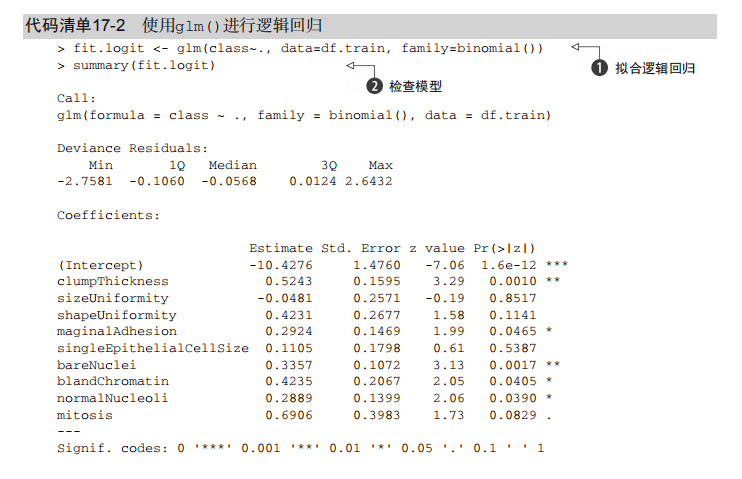
fit.logit <- glm(class~., data=df.train, family=binomial())
summary(fit.logit)
prob <- predict(fit.logit, df.validate, type="response")
logit.pred <- factor(prob > .5, levels=c(FALSE, TRUE),
labels=c("benign", "malignant"))
logit.perf <- table(df.validate$class, logit.pred,
dnn=c("Actual", "Predicted"))
logit.perf # Listing 17.3 - Creating a classical decision tree with rpart()
library(rpart)
set.seed(1234)
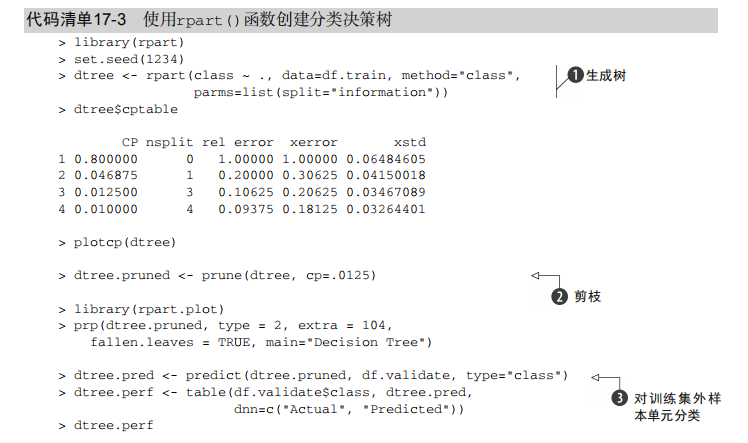
dtree <- rpart(class ~ ., data=df.train, method="class",
parms=list(split="information"))
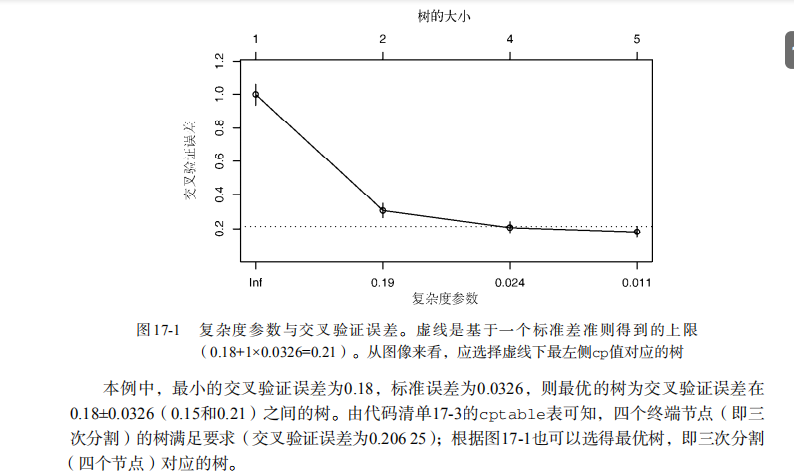
dtree$cptable
plotcp(dtree) dtree.pruned <- prune(dtree, cp=.0125) library(rpart.plot)
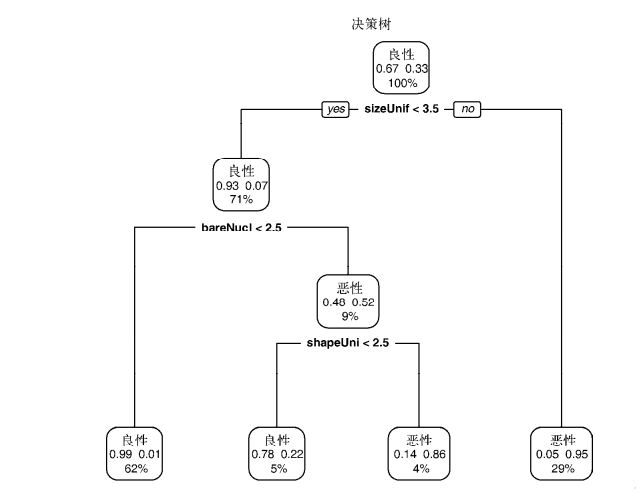
prp(dtree.pruned, type = 2, extra = 104,
fallen.leaves = TRUE, main="Decision Tree") dtree.pred <- predict(dtree.pruned, df.validate, type="class")
dtree.perf <- table(df.validate$class, dtree.pred,
dnn=c("Actual", "Predicted"))
dtree.perf # Listing 17.4 - Creating a conditional inference tree with ctree()
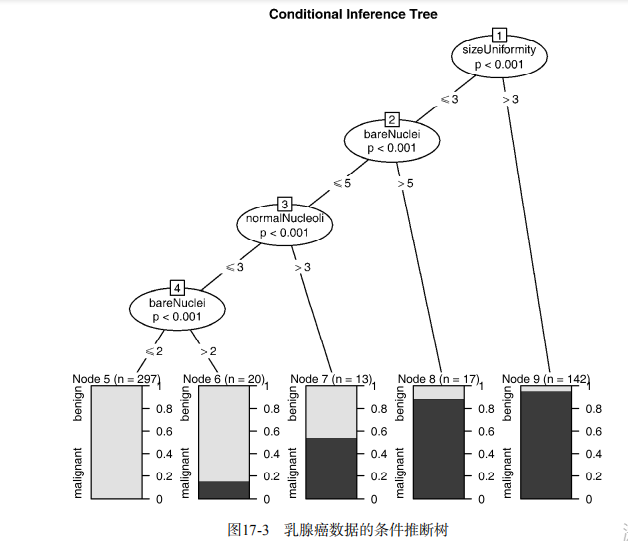
library(party)
fit.ctree <- ctree(class~., data=df.train)
plot(fit.ctree, main="Conditional Inference Tree") ctree.pred <- predict(fit.ctree, df.validate, type="response")
ctree.perf <- table(df.validate$class, ctree.pred,
dnn=c("Actual", "Predicted"))
ctree.perf # Listing 17.5 - Random forest
library(randomForest)
set.seed(1234)
fit.forest <- randomForest(class~., data=df.train,
na.action=na.roughfix,
importance=TRUE)
fit.forest
importance(fit.forest, type=2)
forest.pred <- predict(fit.forest, df.validate)
forest.perf <- table(df.validate$class, forest.pred,
dnn=c("Actual", "Predicted"))
forest.perf # Listing 17.6 - A support vector machine
library(e1071)
set.seed(1234)
fit.svm <- svm(class~., data=df.train)
fit.svm
svm.pred <- predict(fit.svm, na.omit(df.validate))
svm.perf <- table(na.omit(df.validate)$class,
svm.pred, dnn=c("Actual", "Predicted"))
svm.perf # Listing 17.7 Tuning an RBF support vector machine (this can take a while)
set.seed(1234)
tuned <- tune.svm(class~., data=df.train,
gamma=10^(-6:1),
cost=10^(-10:10))
tuned
fit.svm <- svm(class~., data=df.train, gamma=.01, cost=1)
svm.pred <- predict(fit.svm, na.omit(df.validate))
svm.perf <- table(na.omit(df.validate)$class,
svm.pred, dnn=c("Actual", "Predicted"))
svm.perf # Listing 17.8 Function for assessing binary classification accuracy
performance <- function(table, n=2){
if(!all(dim(table) == c(2,2)))
stop("Must be a 2 x 2 table")
tn = table[1,1]
fp = table[1,2]
fn = table[2,1]
tp = table[2,2]
sensitivity = tp/(tp+fn)
specificity = tn/(tn+fp)
ppp = tp/(tp+fp)
npp = tn/(tn+fn)
hitrate = (tp+tn)/(tp+tn+fp+fn)
result <- paste("Sensitivity = ", round(sensitivity, n) ,
"\nSpecificity = ", round(specificity, n),
"\nPositive Predictive Value = ", round(ppp, n),
"\nNegative Predictive Value = ", round(npp, n),
"\nAccuracy = ", round(hitrate, n), "\n", sep="")
cat(result)
} # Listing 17.9 - Performance of breast cancer data classifiers
performance(dtree.perf)
performance(ctree.perf)
performance(forest.perf)
performance(svm.perf) # Using Rattle Package for data mining loc <- "http://archive.ics.uci.edu/ml/machine-learning-databases/"
ds <- "pima-indians-diabetes/pima-indians-diabetes.data"
url <- paste(loc, ds, sep="")
diabetes <- read.table(url, sep=",", header=FALSE)
names(diabetes) <- c("npregant", "plasma", "bp", "triceps",
"insulin", "bmi", "pedigree", "age", "class")
diabetes$class <- factor(diabetes$class, levels=c(0,1),
labels=c("normal", "diabetic"))
library(rattle)
rattle()
吴裕雄--天生自然 R语言开发学习:分类的更多相关文章
- 吴裕雄--天生自然 R语言开发学习:R语言的安装与配置
下载R语言和开发工具RStudio安装包 先安装R
- 吴裕雄--天生自然 R语言开发学习:数据集和数据结构
数据集的概念 数据集通常是由数据构成的一个矩形数组,行表示观测,列表示变量.表2-1提供了一个假想的病例数据集. 不同的行业对于数据集的行和列叫法不同.统计学家称它们为观测(observation)和 ...
- 吴裕雄--天生自然 R语言开发学习:导入数据
2.3.6 导入 SPSS 数据 IBM SPSS数据集可以通过foreign包中的函数read.spss()导入到R中,也可以使用Hmisc 包中的spss.get()函数.函数spss.get() ...
- 吴裕雄--天生自然 R语言开发学习:使用键盘、带分隔符的文本文件输入数据
R可从键盘.文本文件.Microsoft Excel和Access.流行的统计软件.特殊格 式的文件.多种关系型数据库管理系统.专业数据库.网站和在线服务中导入数据. 使用键盘了.有两种常见的方式:用 ...
- 吴裕雄--天生自然 R语言开发学习:R语言的简单介绍和使用
假设我们正在研究生理发育问 题,并收集了10名婴儿在出生后一年内的月龄和体重数据(见表1-).我们感兴趣的是体重的分 布及体重和月龄的关系. 可以使用函数c()以向量的形式输入月龄和体重数据,此函 数 ...
- 吴裕雄--天生自然 R语言开发学习:基础知识
1.基础数据结构 1.1 向量 # 创建向量a a <- c(1,2,3) print(a) 1.2 矩阵 #创建矩阵 mymat <- matrix(c(1:10), nrow=2, n ...
- 吴裕雄--天生自然 R语言开发学习:图形初阶(续二)
# ----------------------------------------------------# # R in Action (2nd ed): Chapter 3 # # Gettin ...
- 吴裕雄--天生自然 R语言开发学习:图形初阶(续一)
# ----------------------------------------------------# # R in Action (2nd ed): Chapter 3 # # Gettin ...
- 吴裕雄--天生自然 R语言开发学习:图形初阶
# ----------------------------------------------------# # R in Action (2nd ed): Chapter 3 # # Gettin ...
- 吴裕雄--天生自然 R语言开发学习:基本图形(续二)
#---------------------------------------------------------------# # R in Action (2nd ed): Chapter 6 ...
随机推荐
- UML-领域模型-如何找到概念类
有3个方法 方法1:对已有的重用和修改(这是最好的方法) 重用和修改现有模型.这些模型来源于之前的项目.网上的 方法2:使用分类列表 从网上搜索该领域的常见分类,或参考书籍Martin Fowler的 ...
- 你需要了解的JIT Debugging
原总结debug调试dump转储文件windbgprocdumpJIT Debugger 如果你还不清楚什么是转储文件,不知道什么时候需要转储文件,请参考转储文件系列文章的第一篇 -- 转储文件知多少 ...
- RegressionTree(回归树)
1.概述 回归树就是用树模型做回归问题,每一片叶子都输出一个预测值.预测值一般是该片叶子所含训练集元素输出的均值, 即
- AFN Post请求,报错400(code:-1011)
解决方法: 声明请求的参数格式是json, post的数据格式还是传字典. 声明代码: AFHTTPSessionManager *manager = [AFHTTPSessionManager ma ...
- 两个tomcat使用同一个jvm可能会出错
如果两个tomcat中的项目的某些类具有完全相同的包路径和类名的话,jvm可能会“弄混”这两个类,所以一般要求包名“必须”唯一. 当然,如果两个类中的代码和import的类完全一样,弄混了也就弄混了, ...
- [转载]markown语法
目录 Cmd Markdown 公式指导手册 一.公式使用参考 1.如何插入公式 2.如何输入上下标 3.如何输入括号和分隔符 4.如何输入分数 5.如何输入开方 6.如何输入省略号 7.如何输入矢量 ...
- JS事件高级
1. 注册事件(绑定事件) 1.1注册事件概述 1.2 addEventListener 事件监听方式 1.3 attachEvent 事件监听方式 1.4 注册事件兼容性解决方案 2. 删除事件(解 ...
- ILSVRC2012下载
http://www.image-net.org/challenges/LSVRC/2012/nnoupb/ILSVRC2012_img_test.tarhttp://www.image-net.or ...
- LeetCode——919.完全二叉树插入器
完全二叉树是每一层(除最后一层外)都是完全填充(即,结点数达到最大)的,并且所有的结点都尽可能地集中在左侧. 设计一个用完全二叉树初始化的数据结构 CBTInserter,它支持以下几种操作: CBT ...
- 安装R的h5包
系统 redhat7 安装H5的包, 依赖系统hdf5包,如下安装完, 发现版本不一致, sudo yum install hdf5-devel 解决办法: 移花接木 sudo ln -s /opt/ ...
