5.MCScanX 与circos下载、安装、运用
一、MCSCAN
参考 :http://chibba.pgml.uga.edu/mcscan2/MCScanX.zip http://chibba.pgml.uga.edu/mcscan2/#tm
安装 :unzip MCscanX.zip && cd MCScanX && make
安装报错:
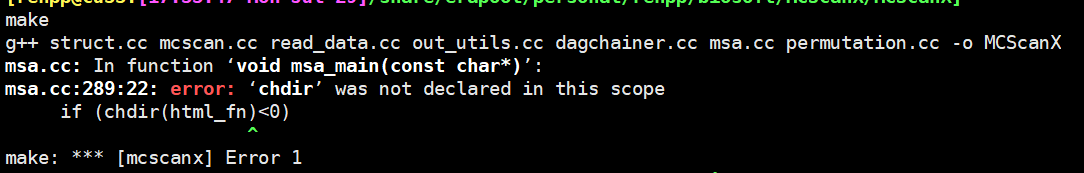
报错解决:
这个错误的原因是,MCScanX 不支持64位系统。如果要在 64位上运行,需要修改下源代码。只需要给 msa.h, dissect_multiple_alignment.h, and detect_collinear_tandem_arrays.h 这三个文件 前面添加 #include <unistd.h>
1.1 准备 *.blast
/export/software/python-2.7.13/bin/python /home/fanjp/bin/gffStat.py -g A.hypogaea.Chrom.gene.gff3 ##提取最长转录本
getGene.pl gffStat.out/A.hypogaea.Chrom.gene.gff3.longest.gff3 A.hypogaea.genome.fasta >A.hypogaea.genome.fasta.cds
/export/personal/zoum/bin/cds2aa.pl A.hypogaea.genome.fasta.cds >A.hypogaea.genome.fasta.pep
makeblastdb -dbtype prot -parse_seqids -in A.hypogaea.genome.fasta.pep -out A.hypogaea.genome.fasta.pep ##建库,物种间共线性
blastp -query A.hypogaea.genome.fasta.pep -db A.hypogaea.genome.fasta.pep -out Dr_An.blast -evalue 1e-5 -num_threads 16 -outfmt 6 -num_alignments 5 ##Dr_An.blast
1.2 准备 *.gff
perl -lane 'if($F[2]=~/mRNA/){/ID=(.*?)\;/;print join("\t",$F[0],$1,$F[3],$F[4])}' ../A.nigrocauda/A.nigrocauda.final.gff.longest.new.gff3 >Dr_An.gff ##基于最长转录本提取gff, 获得Dr_An.gff
perl -lane 'if($F[2]=~/mRNA/){/ID=(.*?)\;/;print join("\t",$F[0],$1,$F[3],$F[4])}' ../D.rerio/D.rerio.gff >>Dr_An.gff
1.3 共线性分析
sort -nk1 A.hypogaea.genome.fasta.fai|perl -lane 'BEGIN{$a=0}{print join("\t","chr","-",$F[0],$F[0],"0",$F[1],"chr".$a);$a++}' >chr.txt
/export/personal1/mengmh/1.software/MCScanX/MCScanX/MCScanX Dr_An ## Dr_An.collinearity Dr_An.html
perl /share/erapool/personal/renpp/biosoft/circos/script/convert_McScanX_to_links.pl -i1 Dr_An.gff -i2 Dr_An.collinearity >links.txt ##结果如下:
perl -lane 'print join("\t",@F,"color=".lc($F[3]))' links.txt ##增加第七列颜色
二、circos
参考 :http://circos.ca/software/download/ http://circos.ca/documentation/tutorials/ https://www.jianshu.com/p/17117766573a http://blog.sina.com.cn/s/blog_485b444b0102whp4.html https://www.jianshu.com/p/e7ebb8f0100c
安装 :tar xf circos-0.69-9.tgz -C ./ && ./circos-0.69-9/bin/circos -h && circos -modules
/share/erapool/personal/renpp/biosoft/circos/circos-0.69-9/bin/circos -h
用法:
source /share/erapool/personal/renpp/.bashrc
circos -conf circos.conf
2.1 circos流程 和 主要的配置 (1.配置文件 2、输入文件)
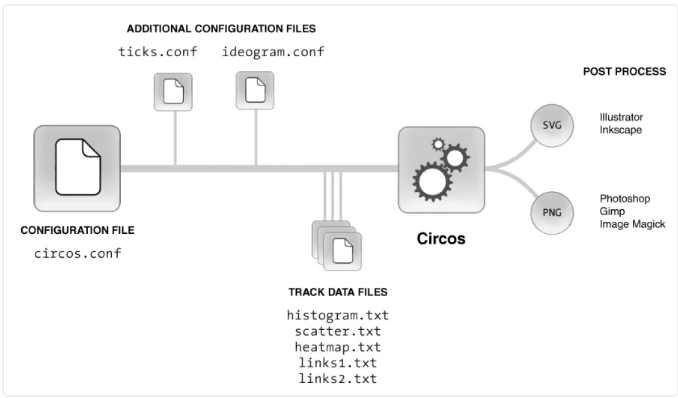
2.2 基本的circos配置文件 变量
karyotype = data/karyotype/karyotype.human.txt
<ideogram>
<spacing>
default = 0.005r
</spacing>
radius = 0.90r
thickness = 20p
fill = yes
stroke_color = dgrey
stroke_thickness = 2p
</ideogram>
<image>
<<include etc/image.conf>>
</image>
<<include etc/colors_fonts_patterns.conf>> ###colors.ucsc.conf 1500p
<<include etc/housekeeping.conf>>
2.3 karyotype:共有7列:chr - ID LABEL START END COLOR
perl -lane '{$a+=1;print join("\t","chr","-",$F[0],$F[0],"0",$F[1],"chr$a")}' ../0HWJHB.final_Chr.fasta.fai|head -24 >chr.txt
perl -lane '{$a+=1;print join("\t","chr","-",$F[0],$a,"0",$F[1],"chr$a")}' ../3D.rerio.fna.fai |less|head -25 >>chr.txt
karyotype = ./chr.txt
chromosomes_units = 2000000
chromosomes_display_default = yes
chromosomes = -ContigUN;
#chromosomes = hs1;hs2;hs3;-hs4;hs5:1-100;-hs6:50-);/hs[7-9]$/
#chromosomes_reverse = hs2;hs3;/hs[234]/
#chromosomes_order = hs1;hs3;hs2;^,hs5;hs4,$
#chromosomes_colors = hs1=red,hs2=orange,hs3=green,hs4=blue
#chromosomes_radius = hs1:0.5r;hs2:0.55r;hs3:0.6r
#chromosomes_scale = /hs[234]/=0.5rn
2.4 ideogram
<ideogram>
<spacing>
default = 0.005r
<pairwise Chr01;Chr02>
spacing = 4r
</pairwise>
</spacing>
#position configuration
radius = 0.80r
thickness = 20p
fill = yes
#fill_color = black
stroke_thickness = 3
stroke_color = dgrey
#label configuration
show_label = yes
label_font = default
label_radius = dims(ideogram,radius) + 0.065r
label_size = 30
label_parallel = yes
#band configuration
# show_bands = yes
# fill_bands = yes
# band_stroke_thickness = 2
# band_stroke_color = white
# band_transparency = 0
</ideogram>
2.3 ticks
show_ticks = yes
show_tick_labels = yes
<ticks>
radius = dims(ideogram,radius_outer)
color = black
thickness = 2p
multiplier = 1e-6
format = %d
<tick>
spacing = 1u
size = 10p
color = lgrey
show_label = no
</tick>
<tick>
spacing = 5u
size = 15p
show_label = yes
label_size = 20p
label_offset = 10p
format = %d
</tick>
</ticks>
2.4 plots
<plots>
<plot>
type = histogram
file = ./chr1.txt.11
r0 = 0.70r
r1 = 0.75r
max = 1
min = 0
orientation = out
fill_color = blue ##line heatmap histogram
</plot>
<plot>
type = heatmap
file = ./chr1.txt.12
r0 = 0.60r
r1 = 0.65r
max = 1
min = 0
color = yellow
</plot>
<plot>
type = scatter ##scatter line heatmap
file = ./chr1.txt.13
r0 = 0.50r
r1 = 0.55r
max = 1
min = 0
fill_color = black
stroke_color = black
</plot>
</plots>
2.5 links.conf
示例1:hs1 400 550 hs3 500 750 color=red ##1、共有7列定义不同染色体links的染色
<links>
<link>
file = ref/MCSCAN/Dr_An_links.txt
radius = 0.90r
bezier_radius = 0r
color = black_a4
thickness = 2
</link>
</links>
示例2:hs1 400 550 hs3 500 750 ##共有六列
<rules>
<rule>
condition = var(intrachr) ##染色体内的 links 对不显示
show = no
</rule>
<rule>
condition = 1
color = var(chr2) #2、所有links的颜色都是第二条染色体的颜色,也就是结束一端的颜色。如果写成color = var(chr2)那所有的线就是都是2号染色体的颜色了。前提的是:颜色的配置文件中要有自定义好的别名(如chr1 chr2 等),组型文件中至少有一个是染色体提用到别名 etc/colors.ucsc.conf 3、通过列表获得:https://www.jianshu.com/p/3fd9175abad0
flow = continue
</rule>
<rule>
condition = between(hs1,hs2) ###两个染色体间的 links 显示的染色
color = green
z = 10
flow = continue
</rule>
<rule>
condition = between(hs2,hs3)
color = blue
thickness = 4
z = 15
</rule>
</rule>
condition支持的函数 :
1、value 获取指定字段的值,var返回值可以分成以下3类 字符串,数字,逻辑值
CHRn var(chr1), var(chr2) #字符串
STARTn var(start1), var(start2)
SIZEn var(size1), var(size2) #数字
INTERCHR var(interchr) 如果一个link连接的两个区域位于两条染色体上,返回值为1 #逻辑值
INTRACHR var(intrachr) 如果一个link连接的两个区域位于同一条染色体上,返回值为1
2、between
condition = between(hs1, hs2)
http://circos.ca/documentation/tutorials/links/rules2/ https://www.jianshu.com/p/3fd9175abad0 ##links
5.MCScanX 与circos下载、安装、运用的更多相关文章
- Visual Studio for Mac Preview离线下载安装
Visual Studio for Mac离线下载安装. 环境:OS X EI Caption 10.11.2 .NET Core SDK 1.1 需预先安装 .NET Core 1.1 SDK ma ...
- jdk1.8下载安装
jdk8环境变量 jdk8图解安装 java8安装 1 2 3 4 5 6 7 分步阅读 JDK8 是JDK的最新版本,加入了很多新特性,如果我们要使用,需要下载安装: JDK8在windows ...
- Mac下载安装Android Studio教程
今天把公司闲置的一台Mac-mini重装了下系统感觉用着速度还不错,平时上班用的机器USB有些问题,所以打算用这台Mac.以往开发用Intellij Idea就够用,但是这次项目引用的jar包太多,遭 ...
- mac版 android破解软件下载安装
1 apktool下载安装 下载地址https://code.google.com/p/android-apktool/ [1].下载apktool.jar — 解压 [2].下载Mac上的辅助工具a ...
- 不通过App Store实现ios应用分发下载安装
最近公司的项目准备着手宣传工作了,宣传手册上要印制App的下载地址二维码,但是客户端应用还未上线,需要一种临时的方案解决应用分发下载问题,通常ios应用必须通过苹果应用商店才能下载安装,但是也可以看到 ...
- ERWin 7.2下载安装及注册机
ERWin 7.2下载安装及注册机 ERWin 7.2 下载地址: ftp://ftp.ca.com/CAproducts/erwin/ServicePacks/AFEDM72-b1644.exe ...
- cocoapod的下载安装解释
本文不提供cocoapod的下载安装的流程,因为那些只要百度一下就有的东西,而是对里面的代码进行解释,希望对iOS小白安装cocoapod有帮助: 一.cocoapod是什么? 开发过程中,我们会用到 ...
- 配置ActiveX控件在网页中下载安装
先检查客户端浏览器是否安装了ActiveX控件,如果没有安装ActiveX,就需要先给浏览器提示下载并允许安装.否则就直接使用该ActiveX控件.我们可以使用CodeBase来满足我们的要求:下面是 ...
- Xamarin Anroid开发教程之下载安装Xamarin
Xamarin Anroid开发教程之下载安装Xamarin Xamarin在过去安装时都会检查系统中是否安装了前面所提供的内容.而后来,Xamarin安装时只提供安装步骤,其它内容都需要读者自己下载 ...
随机推荐
- LeetCode--146--LRU缓存机制(python)
运用你所掌握的数据结构,设计和实现一个 LRU (最近最少使用) 缓存机制.它应该支持以下操作: 获取数据 get 和 写入数据 put . 获取数据 get(key) - 如果密钥 (key) 存 ...
- SpringBoot路径映射
当然这个功能并非是springboot特有的,只是springboot提供了更简便的方法以供使用. 传统情况下我们跳转一个动态页面且并没有数据,也需要在controller中写一个跳转的con ...
- vim插件cscope使用方法
一.安装cscope 安装方式比较多样,可以在各自linux软件管理工具中安装,也可以去官网下载安装. sudo apt-get install cscope 二.插件安装 这里选择的是Vundle来 ...
- Spring read-only="true" 只读事务的一些概念
概念:从这一点设置的时间点开始(时间点a)到这个事务结束的过程中,其他事务所提交的数据,该事务将看不见!(查询中不会出现别人在时间点a之后提交的数据) 应用场合: 如果你一次执行单条查询语句,则没有必 ...
- 【WINDOWS】设置路由表实现有线内网,无线外网
前提!!! 需要有线无线双网卡
- BZOJ 4939: [Ynoi2016]掉进兔子洞(莫队+bitset)
传送门 解题思路 刚开始想到了莫队+\(bitset\)去维护信息,结果发现空间不太够..试了各种奇技淫巧都\(MLE\),最后\(\%\)了发题解发现似乎可以分段做..这道题做法具体来说就是开\(3 ...
- 3D Computer Grapihcs Using OpenGL - 12 Rotation Matrix
为了证明我们上节渲染出来的是一个立方体而不是一个平面,我们决定将它旋转一定角度,这样我们就需要一个旋转矩阵(也属于ModelTransformMatrix的一部分) 上一节我们的ModelTransf ...
- UIWebView 禁止检测链接弹出UIActionSheet
解决方法一: 添加以下代码禁止检测类型 webView.dataDetectorTypes = UIDataDetectorTypeNone; 解决方法二: - (void)webViewDidFin ...
- python3下multiprocessing、threading和gevent性能对比----暨进程池、线程池和协程池性能对比
python3下multiprocessing.threading和gevent性能对比----暨进程池.线程池和协程池性能对比 标签: python3 / 线程池 / multiprocessi ...
- 四种方法给Vmware虚拟机清理瘦身
随着VMware虚拟机使用时间的增长,其所占用的空间也越来越大,本文来说说怎么给VMware虚拟机占用的空间进行瘦身. **方法一:VMware自带的清理磁盘 **这个方法是VMware自带,具有普适 ...
